Scanpy数据结构:AnnData
AnnData是python中存储单细胞数据的一种格式
1. AnnData数据结构:
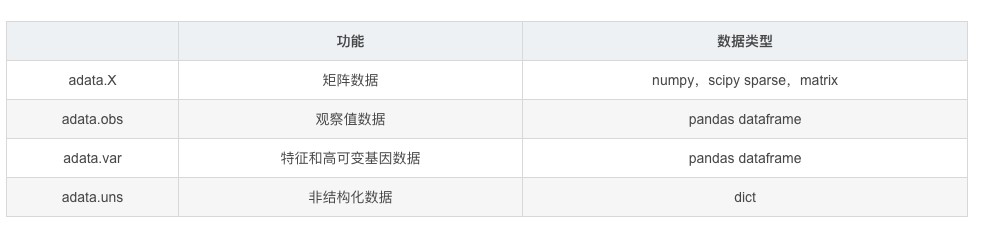
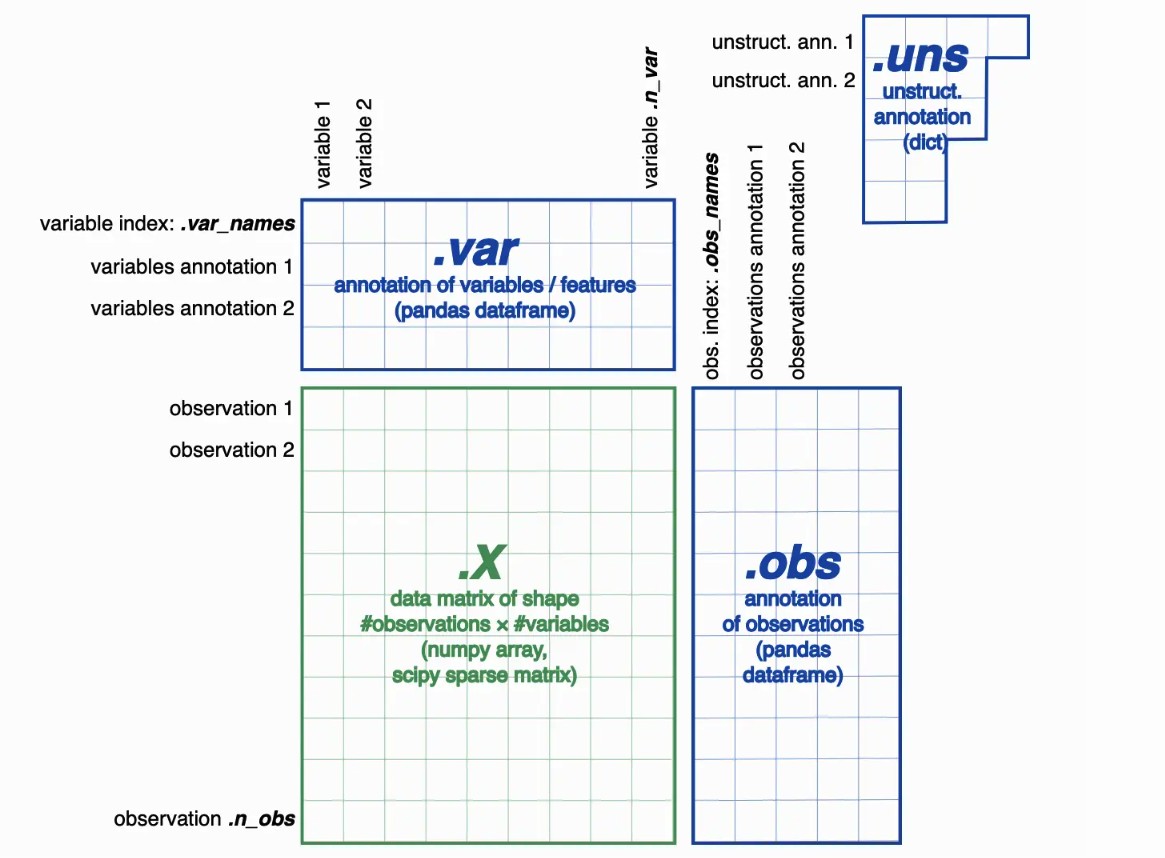
主要包含四个slots:
X(数据矩阵): 存储单细胞数据的核心矩阵,通常是一个二维数组,其中行表示细胞,列表示特征(基因或其他测量值)。
obs(观测信息): 包括每个细胞的元信息,如样本名称、细胞类型、质量信息等。obs 是一个观测特征的字典。
var(变量信息): 包括每个特征(基因)的元信息,如基因名、功能注释等。var 是一个变量特征的字典。
layers(层): 可以存储其他数据层,如归一化后的数据或差异表达分析的结果。
uns(未排序的数据): 用于存储其他未排序的数据和元信息
AnnData object with n_obs × n_vars = 6050 × 32285
obs: 'in_tissue', 'array_row', 'array_col', 'gender', 'age', 'tissue', 'Strain', 'n_genes_by_counts', 'log1p_n_genes_by_counts', 'total_counts', 'log1p_total_counts', 'pct_counts_in_top_50_genes', 'pct_counts_in_top_100_genes', 'pct_counts_in_top_200_genes', 'pct_counts_in_top_500_genes', 'total_counts_mt', 'log1p_total_counts_mt', 'pct_counts_mt', 'n_counts', 'n_genes'
var: 'highly_variable', 'means', 'dispersions', 'dispersions_norm'
uns: 'spatial', 'log1p', 'hvg', 'pca', 'neighbors', 'umap', 'tsne', 'tissue_colors'
obsm: 'spatial', 'X_pca', 'X_umap', 'X_tsne'
varm: 'PCs'
obsp: 'distances', 'connectivities'
Obsm:对于观测的多维注释,它是可变的ndarray,就是说维度是可变的!这里的维度一般是2至多维哦。而Obsm这里的m指的就是multi-dim多个维度的,obs_m对应于obs,但obs的每个成员都是一维的观测注释,obs_m的每个成员(X_pac和X_umap)都是多维的观测注释。哦对了,忘了告诉你,obs就是矩阵的行,也就是样本!
Varm:与obsm对应,是基因的多维观测~
uns:非结构化数据,也就是字典。
rank_genes_groups 识别差异表达的基因(潜在的marker),这个函数将获取每组细胞,并将每组中每个基因的分布与不在该组中的所有其他细胞中的分布进行比较最后进行识别。
pca:主成分分析降低数据维度。
umap降维:umap比tsne保留全局结构,允许大数据输入,运行时间短,这是umap和tsne的区别,一定要牢记喔!
obsp(pair):表示细胞和细胞之间的距离和连通性。
n_genes_by_counts:每个细胞中,有表达的基因的个数;
total_counts:每个细胞的基因总计数(总表达量)
pct_counts_mt:每个细胞中,线粒体基因表达量占该细胞所有基因表达量的百分比
1.2 创建 Anndata 对象: 可以使用 Anndata 构造函数创建一个 Anndata 对象。
import anndata as ad
adata = ad.AnnData(X=data_matrix, obs=obs_info, var=var_info)
1.3 数据操作: Anndata 允许您执行多种数据操作,包括切片、过滤、转置、连接数据、添加元信息等。
# 切片数据
subset_data = adata[:, list_of_genes]
# 过滤细胞
adata = adata[adata.obs['quality'] > 0.9]
# 转置数据
adata_T = adata.T
1.4 数据可视化: Anndata 可以与 scanpy 或其他可视化工具结合使用,以可视化数据、绘制UMAP、t-SNE图等。
import scanpy as sc
sc.tl.pca(adata)
sc.pl.umap(adata, color='cell_type')
1.5 数据存储: Anndata 可以将数据存储为HDF5文件,以便将数据持久化和共享。
adata.write('my_data.h5ad')
- 发表于 2024-10-11 15:42
- 阅读 ( 1361 )
- 分类:转录组
你可能感兴趣的文章
- 10X Visium空间转录组图像 897 浏览
