R语言绘制维恩图空值NA处理
行数不同的数据读入R,批量去除NA,绘制韦恩图。
绘制venn图的时候,我们数据的ID中有缺失值,也就是不同的ID行数不一样,需要绘制韦恩图,那么空值就读成了NA,需要去除才行;
数据如下,不同比较组的得到的差异基因ID,需要用R绘制韦恩图:
代码及注意事项如下:
library(Vennerable)
#由于行数不一样,设置fill参数;na.strings 设置空值为NA:
All_DEG_venn<-read.table("All_DEG_venn.genes",header=TRUE,comment.char="",sep = "\t",check.names=FALSE,fill=T,na.strings="")
colnames(All_DEG_venn)<-c("DAF2 vs GDAF2","DAF5 vs GDAF5","DAF11 vs GDAF11","DAF16 vs GDAF16")
#去除数据中的NA,以免多计数
All_DEP_ID=apply(All_DEG_venn,2,na.omit)
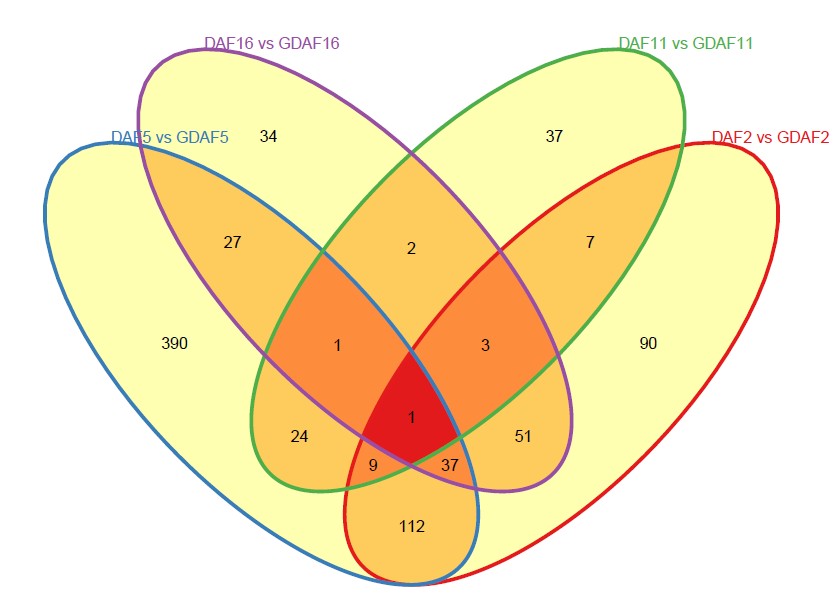
datav<-Venn(All_DEG_venn)
plot(datav,doWeight=F,type="ellipses")
- 发表于 2018-07-05 16:16
- 阅读 ( 8951 )
- 分类:R
你可能感兴趣的文章
- 已知集合交集数量绘制韦恩图 273 浏览
- 绘制Upset 图 1420 浏览
- R公式中如何加入变量 697 浏览
- R语言基础入门—数据结构(2) 700 浏览
- R语言基础入门—数据结构(1) 676 浏览
- R语言基础入门—R语言概述与安装! 858 浏览
0 条评论
请先 登录 后评论
