快速解读:3-5分的蛋白质组学文章思路
iTRAQ (Isobaric tag for relative and absolute quantitation,iTRAQ)和TMT(Tandem Mass Tag)是近年来应用最广泛的差异蛋白质组学技术,它们均采用体外标记的方法,利用同位素试剂标记蛋白质酶解后产生的多肽,对两个或多个样本,在全蛋白质组层面上展开相对定量分析。
随着实验技术的不断深入和完善,越来越多的研究者开始将研究重点从转录表达层面转移到了蛋白层面。
做了蛋白质组学分析之后,随之而来的问题就是数据的深入和文章的撰写,这让很初学者都头痛不已。
这里,小编就用一篇今年发表在frontiers in plant science的蛋白质组学文章给大家讲解一下相应的文章思路。
灰霉病是猕猴桃最重要的采后病害,本研究的目的是了解猕猴桃和灰霉病菌的相互作用,为灰霉病的防控提供新的见解和依据。
具体来说,文章是这样的:
实验材料的准备
选取大小均与且无伤口无腐烂的猕猴桃,经过消毒清洗风干后,用灰霉病菌孢子悬浮液和无菌水对果实进行接种,经过24h温育后进行取样。处理和对照各三个生物学重复,共6个样品。

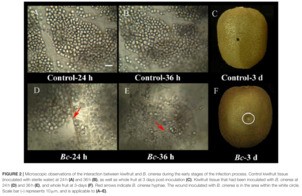
显微镜下观察病症发展状况
收集接种24小时、36小时以及3天后的猕猴桃,在显微镜下观察病症的发展状况。每个时间点重复三次,每个重复检查五个果实。
蛋白质组学分析
通过与猕猴桃基因组比较,共鉴定出2487个蛋白。按照P<0.05、上调>1.33倍、下调<o.75倍的差异标准,共鉴定出292个差异蛋白,其中196个上调,96个下调。

将鉴定出来的差异蛋白与KEGG、GO以及COG等数据库进行注释分析,发现许多差异蛋白参与了植物与病原体的相互作用、信号转导、翻译后修饰以及防御反应机制。
从差异分析的结果中挑选出了一些和细胞壁降解、MAPK级联反应、ROS信号转导以及PR蛋白等相关的差异蛋白进行详细的功能分析,并结合前人的研究结果进行相应的讨论和阐述。
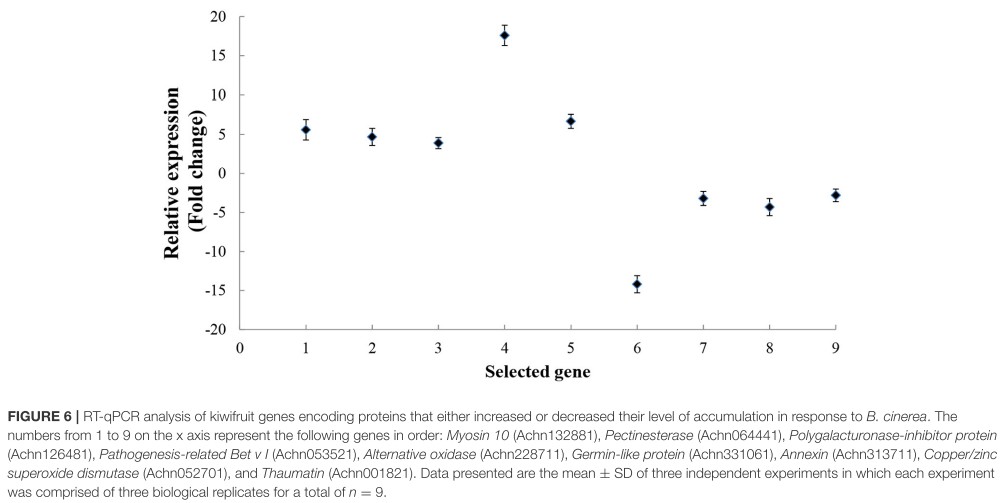
RT-qPCR验证
从差异分析的结果中挑选了9个差异蛋白,并找到了对应的9个基因,用于RT-qPCR分析,以验证差异蛋白的准确性。结果表明所选择的差异蛋白对应基因的表达情况与蛋白组学分析结果一致。
到此,这篇文章也基本完成了。文章思路并不复杂,设计简单明了,样品不多,性价比较高。
文章的重点是找到相关的差异蛋白或者代谢通路,并结合前人的研究结果进行相应的讨论,得出自己的结论。
如果您需要文章原文,您可以扫描下方二维码关注“组学大讲堂公众号”,然后回复“蛋白质组学文章”获取原文链接。
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课,学习链接:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章,学习链接:TCGA-差异基因分析、GEO芯片数据挖掘、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他课程链接:二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读。
参考文献:
Liu J, Sui Y, Chen H, et al. Proteomic Analysis of Kiwifruit in Response to the Postharvest Pathogen, Botrytis cinerea[J]. Frontiers in Plant Science, 2018, 9: 158.

- 发表于 2018-07-11 15:36
- 阅读 ( 12407 )
- 分类:蛋白质组学
你可能感兴趣的文章
- 3分2区蛋白质组学文章怎么写? 6478 浏览
- 蛋白质组与转录组的关联分析如何做? 19689 浏览
- iTRAQ/TMT蛋白质组学分析基本知识点 15435 浏览
- 简单实验+蛋白质组=4分文章, So Easy! 4457 浏览
- 简单的蛋白质组实验怎么炼成4分文章 4425 浏览
