TCGA 数据的多组学WGCNA联合分析
一般的分析都是针对单一类型的分子进行分析,比如lncRNA, mRNA, miRNA 等,而文章《identifying mirna and gene modules of colon cancer associated with pathological stage by weighted gene co-expression network analysis》则结合Gene,miRNA 分别进行WGCNA的分析,获得性状相关的Hub Gene和 Hub miRNA, 再基于miRNA能调控Gene 的表达,从而构建两者之间的互作网络。
分析流程图
整个的分析流程如下图所示: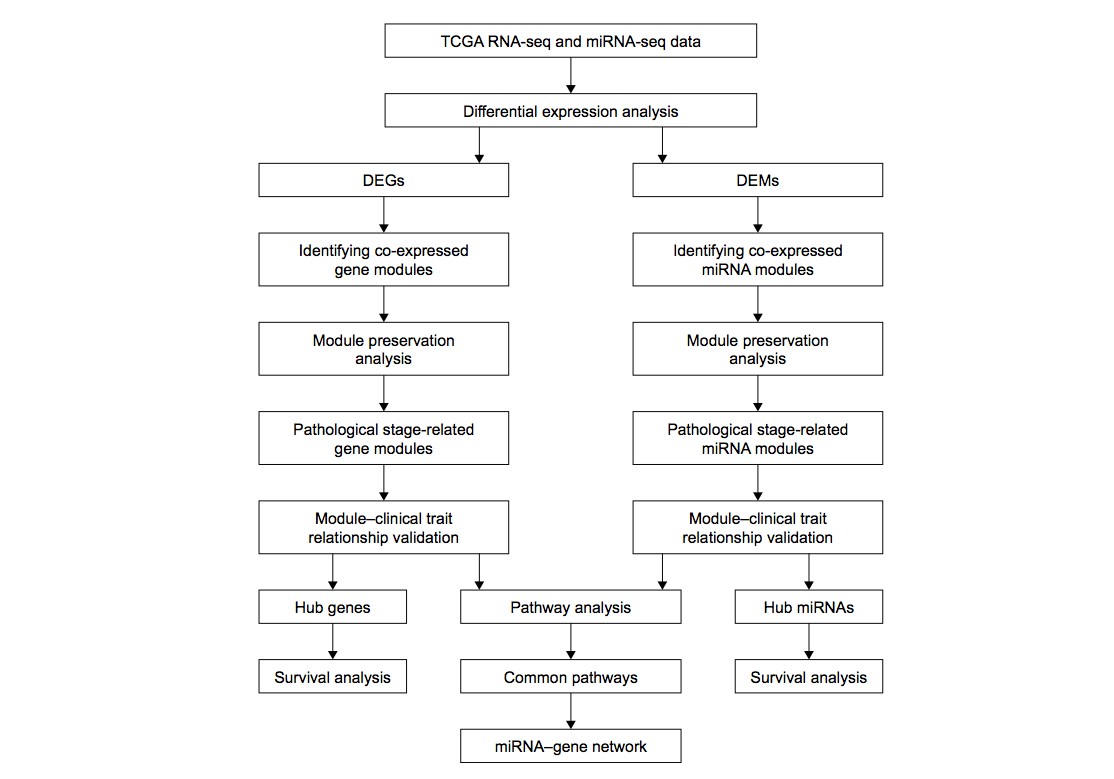
分析流程的步骤简述:
1. 从TCGA下载直肠癌的基因,miRNA表达数据
2. 分别针对基因,miRNA进行表达差异分析
3. 分别针对基因和miRNA进行共表达网络(WGCNA)分析,获得聚类模块,并与病理分期这一性状相关联
4. 选择模块中的Hub Gene, Hub miRNA 进行生存分析
5. 病理分期相关的Gene, miRNA 进行代谢通路分析,构建miRNA-gene 调控网络
分析结果
- 数据下载和筛选
从TCGA数据库中下载并筛选基因表达数据和miRNA表达数据,其中基因表达数据中包含450个癌症组织和41个正常组织样本,而miRNA表达数据中包含455个癌症组织,8个正常组织样本。 - 差异分析
分别就癌症和正常组织中的基因,miRNA进行表达差异分析。6486基因发生了显著差异,其中3678个基因上调,2808个基因下调,而miRNA 则有544个发生了显著的差异,其中350个上调,194个下调。 - 共表达网络分析(WGCNA)对基因进行聚类,形成module
基因,miRNA分别构建WGCNA调控网络。
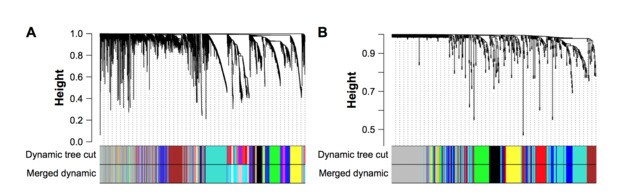
WGCNA module与性状(病理分期)进行相关分析,并筛选Hub Gene 和miRNA
基因module与病理分期进行相关分析, 找到了一些与病理分期相关度较高的模块

miRNA module 与病理分期进行相关分析,筛选那些与病理分期显著相关的模块进行下面的研究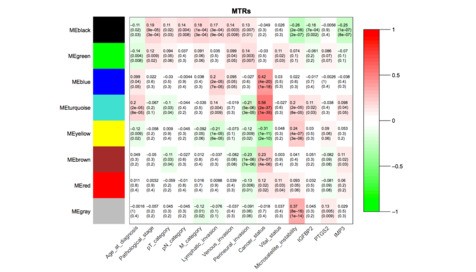

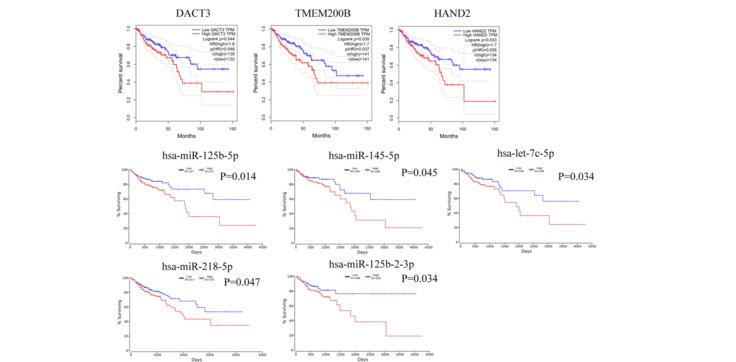
生存分析
显著相关的module 中的hub Gene和miRNA 进行生存分析,可以看到一些Gene和miRNA与癌症的预后有显著的相关性。
miRNA-Gene 调控网络 通过miRNA 预测靶基因,发现miRNA 调控的基因,并将这种调控关系与代谢通路(pathway)相联系,构建miRNA-Gene的调控网络

如果您对TCGA数据挖掘感兴趣,请学习我的TCGA系列课程:
- 发表于 2018-07-20 10:58
- 阅读 ( 14892 )
- 分类:TCGA
你可能感兴趣的文章
- R语言中转换成dummy dummy-variables 虚拟变量 1830 浏览
- GEO数据库挖掘—WGCNA鉴定骨肉瘤转移相关基因 6851 浏览
- 基于多数据集分析ANLN在宫颈癌所起到的功能 7755 浏览
- WGCNA、生存分析、ROC共同筛选biomarker 6591 浏览
- 采用差异表达基因进行WGCNA分析是不合理的 11389 浏览
- TCGA临床数据中的TNM分期,如何转换成WGCNA分析中的性状数据 8066 浏览
相关问题
- wgCNA一个模块中找hub基因 我把cytoScapeinput-edges文件输入cytoscape中 用cytohubba计算 但是每次出来的结果都不一样 试了很多遍也不行 求解答 谢谢! 0 回答
- 我得到转录组分析的wgcna数据需通过cytoscape筛选关键模块的hub基因,边的权重应该设定多少来筛好 1 回答
- WGCNA中过滤低表达量(0.5)时datExpr0=datExpr0[1:n,datExpr0[n+1,] > meanFPKM]不成功,且变量从上步30769变成了0. ? 2 回答
- 跑WGCNA,用的独享服务器,报错 1 回答
- 我想咨询一下wgcna问题 0 回答
- WGCNA表型信息表 1 回答
