18年上半年,利用GEO/TCGA数据库发表文章超过1000+
前几期小编给大家介绍了几篇利用GEO数据库、TCGA数据库发表的SCI论文:如:
1:An integrated lncRNA, microRNA and mRNA signature to improve prognosis prediction of colorectal cancer ,该文采用TCGA数据库中结直肠癌及癌旁组织的mRNA,lncRNA,miRNA进行差异表达,获得显著变化的分子,用于构建生存分析模型,获得疾病风险评估方法。
2:Elevated circulating miR-182 acts as a diagnostic biomarker for early colorectal cancer,该文章利用公共数据(GEO,TCGA数据),筛选出两个miRNA(miR-182 and miR-20a)作为结直肠癌诊断的分子标志物,然后在组织和血液中进行验证,最终确定诊断参数。
3:Expression profiles analysis identifies anovel three-mRNA signature to predict overall survival in oral squamous cellcarcinoma,利用GSE13601,GSE30784, GSE37991三个mRNA芯片表达数据以及TCGA中OSCC表达数据分别做对照和患者差异基因分析及GO和KEGG通路富集分析、PPI网络分析,IPA通路分析,生存分析等,最终筛选到PLAU, CLDN8 and CDKN2A与OSCC预后相关,并且在另一个GEO数据GSE41613中得到了验证。
类似文章还有千千万,这里就不一一列举了,但仔细看来,发现这类文章具有类似的思路构架,这里我们不妨归纳一下此类文章的套路,如下:
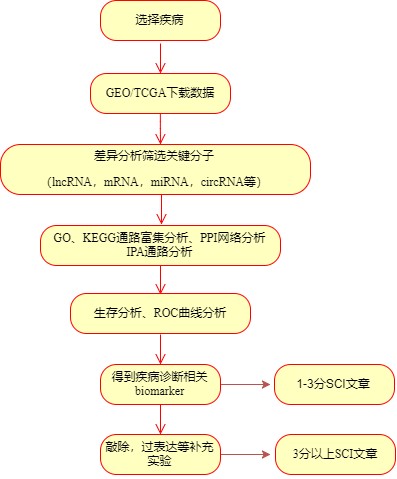
这类文章18年到底还好不好发?我带着这个疑问搜索了下2018年1-7月10日此类文章发表数量才发现,原来有这么多人在闷声发文章:利用GEO数据库,2018年1-7月10日期间共发表了433篇;而利用TCGA数据库,1-7月间共发表文章846篇;真是不少!当你在实验室兢兢业业做实验的时候,别人就在悄无声息地利用你公开的数据,整理自己的论文!你是不是也该开开窍了呢?
小编为了大家查阅18年最新的此类文章方便,特地将2018年利用GEO数据库和TCGA数据库发表的所有文章信息整理成EXCEL;此表包含信息如下:文献题目、期刊名称、期刊影响因子、一作姓名、通讯作者姓名、发表时间、全文下载链接、一作单位名称,如下图:(文末有索取该Excel列表的方法)
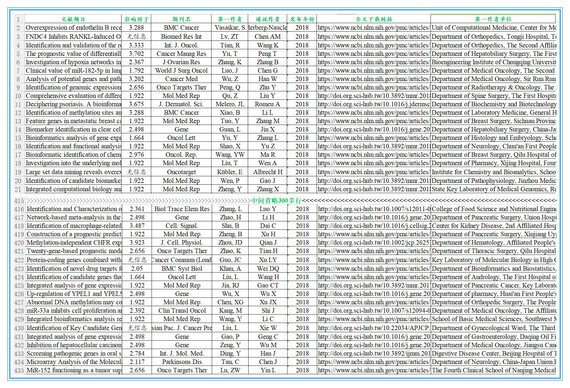
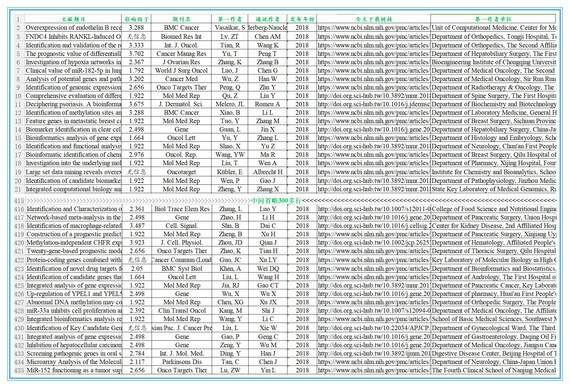
从上表中可以看到,此类文章影响因子最高到30+,也不乏8、9,更有1以下的;这类文章比起从实验做起时间耗费要短的多,是真正的短平快类型的文章。无论你是没经费,还是没材料;是没基础,还是没思路,都可以按照以上文章思路来一篇,这一套分析下来,不仅仅一篇论文有内容了,更是开创了一个有待深入挖掘、验证的课题了。
怎么样?你是不是有点心动呢?那赶快行动起来,今年先定个小目标:两篇起步!
获取数据库文章Excel列表方法:
请先关注“组学大讲堂”微信公众号,然后在公众号对话框内发送 “数据库文章列表” 即可获得该Excel版数据库挖掘文章列表。

- 发表于 2018-07-23 11:42
- 阅读 ( 6000 )
- 分类:TCGA
