做基因定位?大家可能忽略了这个最熟悉的技术
用测序技术做基因定位是老生常谈了,各路大牛们用大群体做的高密度遗传图谱或者GWAS的文章前些年已经席卷CNS了。除了重测序,各路简化基因组技术GBS、RAD、dd-RAD、2b-RAD、SLAF等等都在基因定位领域各显神通。而作为测序界应用最广的转录组在这里反而默默无闻,其实转录组在无参考基因组物种、定位特殊性状的情况下也有自己的优势。
今天先和大家分享一篇用转录组测序来做BSA定位(称为BSR)的文章,作为开胃菜,后续还会和大家陆续探讨用转录组来做基因定位的话题。
想要做定位,先得有什么?
基因分离定律:父母本中决定一个性状的等位基因,在子代中会分离。
第一位当然是实验材料:以抗病的品种(Jazz)和非抗病品种(ACDC)进行杂交,子代F1群体中的抗病特性会存在分离。
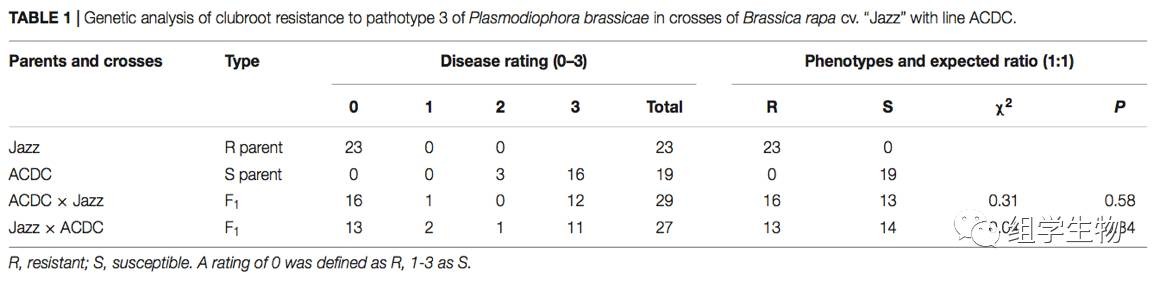
从F1代的抗性测试数据来看,F1群体的抗病特性存在1:1的分离比,那么可以推定决定抗病特性的基因是一个显性遗传的基因,而抗病品种Jazz的基因型是杂合型抗病。
来点不一样的定位方法
连锁与互换定律:在减数分裂时,姐妹染色单体会进行染色体互换,而位于同一条染色体上的两个基因,基因间距越短,越容易出现伴同遗传。
用个不一样的方法:分别选择F1群体中极端表型(抗病,非抗病)的个体30个,形成两个极端表型库(pool),采用转录组测序,call突变位点(SNP)位点。基于两个极端表型库中突变位点与抗病表型的关联分析,在A03染色体上关联到一个4M的区域。

检阅下BSR的结果是不是靠谱?
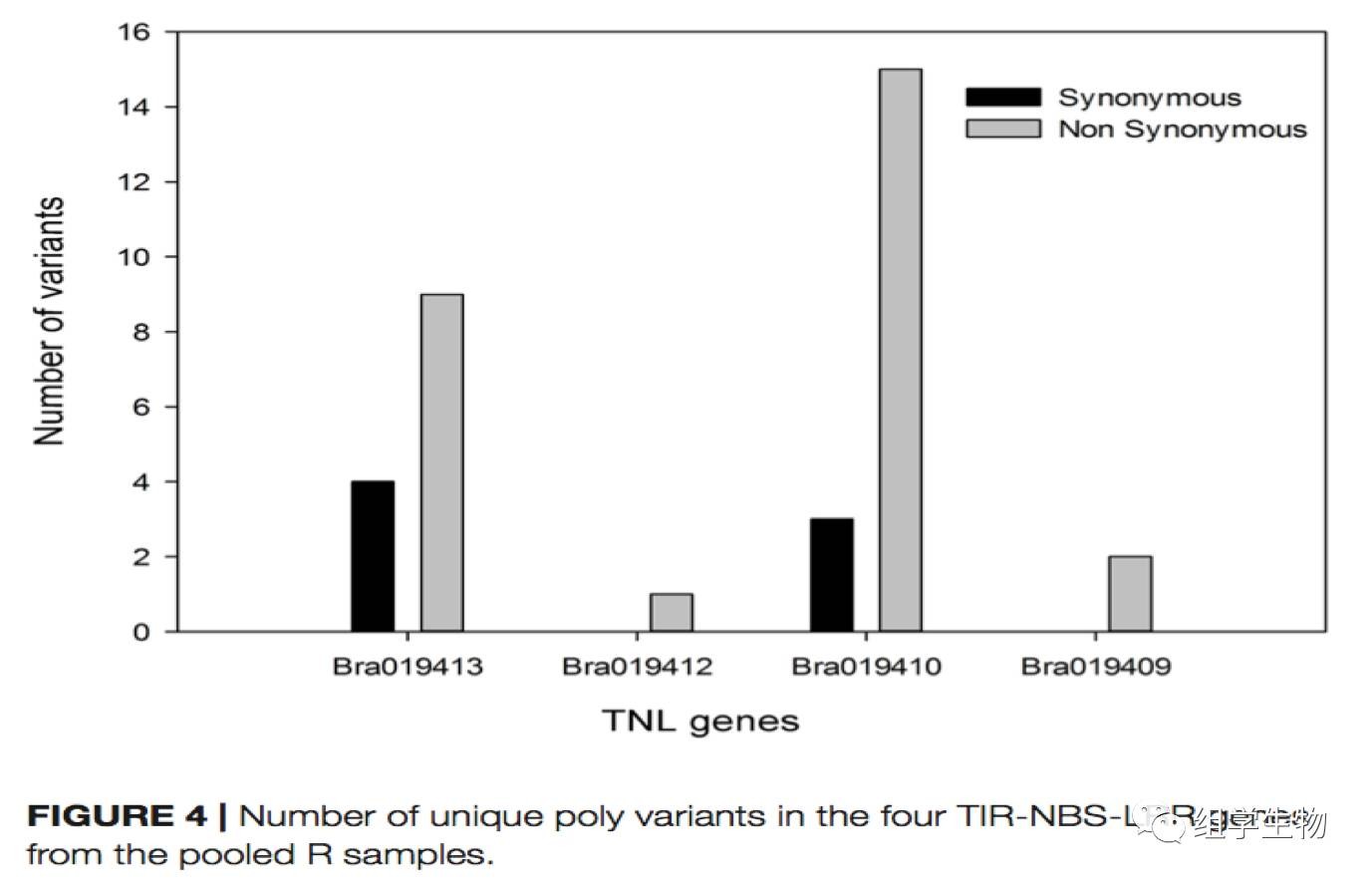
在4M的关联区域,查找与抗病相关的功能基因,找到4个TIR-NBS-LRR家族的抗病基因。将这4个抗病基因上的突变位点按照同义突变和非同义突变进行统计。由图可见Bra019410和Bra019413含有大量的非同义突变,而另外两个基因上的突变位点都比较少,所以基本可以确定,抗性特性应该与Bra019410和Bra019413相关。
为了科学的严谨,预测到的抗病基因还需要采用基因敲除的方法进行实验验证。
总结一下:
基因定位 = 1种材料 + 2个极端表型的关联群体 + 转录组测序
Huang Z, Peng G, Liu X, et al. Fine Mapping of a Clubroot Resistance Gene in Chinese Cabbage Using SNP Markers Identified from Bulked Segregant RNA Sequencing. Frontiers in Plant Science. 2017;8:1448. doi:10.3389/fpls.2017.01448.
- 发表于 2018-04-21 22:52
- 阅读 ( 6156 )
- 分类:转录组
