饼图pie-ggplot2
绘制饼图的过程中,利用ggplot2的geom_bar结合coord_polar实现,需要理解的点是饼图的排布是按照aes(fill)的因子顺序确定的。譬如数据如下
> dat
type Num
1 A 90
2 B 34
3 C 56
4 D 99
5 E 15
必须根据数据先确定mapping 中aes(fill)的因子顺序,譬如这里会按照dat$type 填充,这种非有序因子会基于字母顺序来默认其填充顺序。
为了确定数据填充的先后,同时方便在不同区域上填写上对应数据的大小,所以会先去创建有序因子,从而使数据列dat$Num的自然顺序和因子的顺序在一定程度上一致(一致的同向对应或反向对应)。譬如如下使方向一致:
dat$type=factor(dat$type,levels = dat$type,order=T)
dat$type
有序因子的结果则如下,和dat$Num的顺序能够一致上,不会出现对应错乱问题
[1] A B C D E
Levels: A < B < C < D < E
画图:
p_pie=ggplot(dat,aes(x="",y=dat[,2],fill=dat[,1]))+
geom_bar(stat="identity",width=1)+
coord_polar(theta="y",direction=1)+
scale_fill_brewer(palette ="Set3",direction = 1)+
labs(x="",y="",fill="Type")+
ggtitle(label ="test",subtitle=NULL)
p_pie
结合下图结果可以看出坐标轴方向使顺时针,而颜色设置scale_fill_brewer(palette ="Set3",direction = 1) 设定了第一个颜色填充到第一个因子对应的“A”上,这样就反映出在图片实际分布中数据和因子是反向对应的。虽然在dat数据框中设置是顺序一致方向相同的对应,但图片分布中会改变。
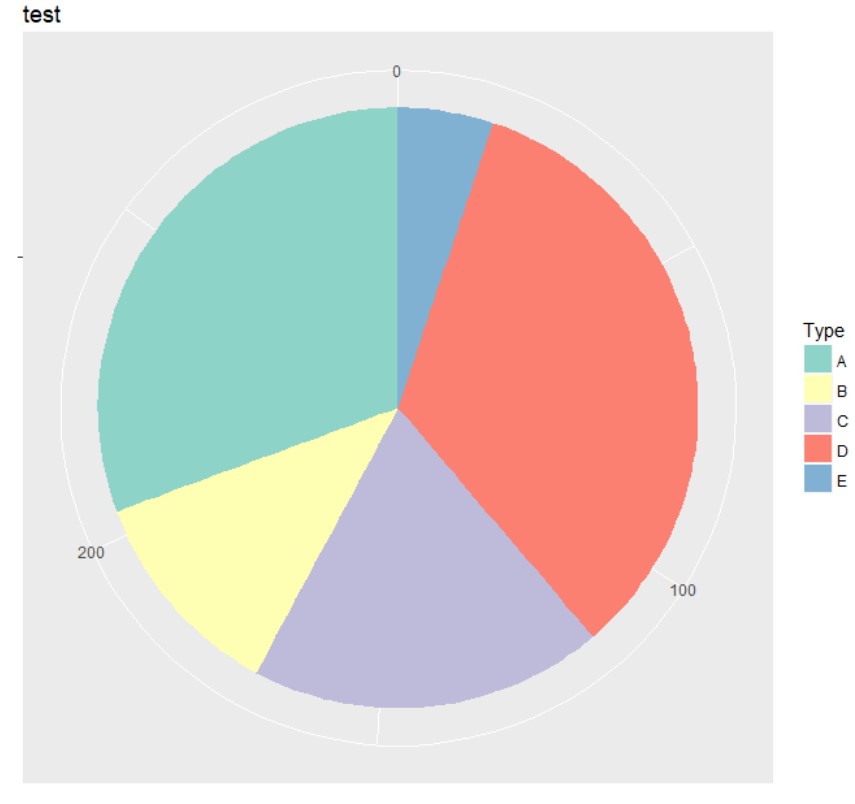
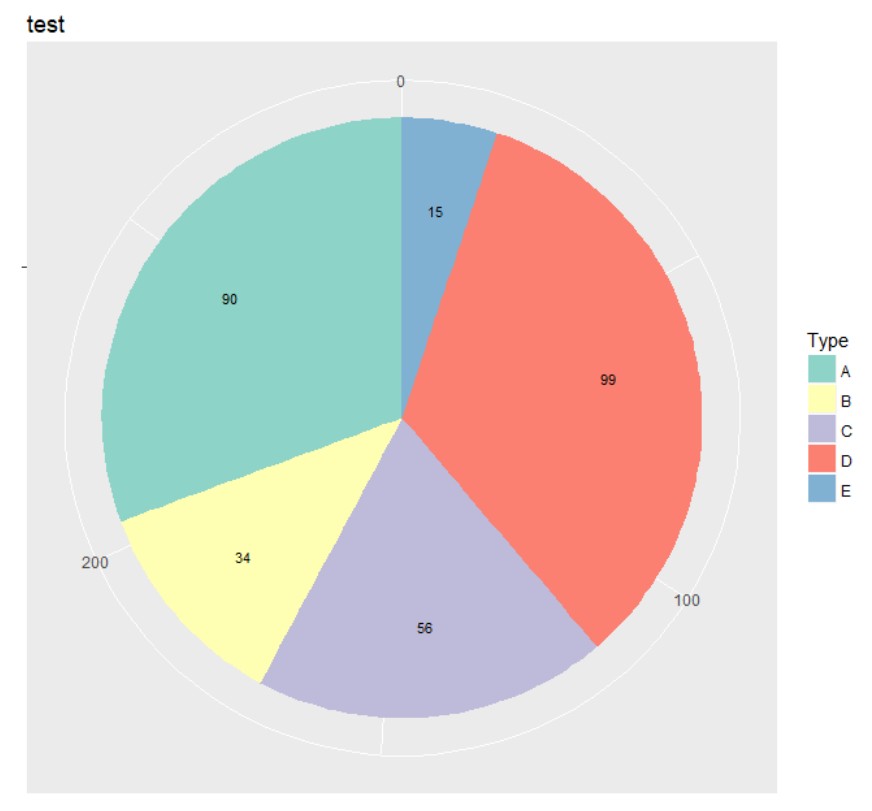
结合图片中反向对应的关系,在A区块上填充上对应的文字Num:90,它的坐标因该是 sum(dat$Num)-90 +90/2,如果是B区块对应的应该坐标为sum(dat$Num)-90-34 +34/2
归纳为sum(dat$Num)-cumsum(dat$Num)+dat$Num/2
sum(dat$Num)-cumsum(dat$Num)+dat$Num/2
[1] 249.0 187.0 142.0 64.5 7.5
结合geom_text(aes(x,y))的位置设置,保证中间文字填写不会出错
p_pie=p_pie+
geom_text(aes(x=1.2,y=sum(dat$Num)-cumsum(dat$Num)+dat$Num/2 ,label=as.character(dat[,2])),size=3)
p_pie
 ##########################如果最初构建有序因子的方向和实际数据的方向反向对应呢?
##########################如果最初构建有序因子的方向和实际数据的方向反向对应呢?
dat$type=factor(dat$type,levels = rev(dat$type),order=T)
dat$type
p_pie=ggplot(dat,aes(x="",y=dat[,2],fill=dat[,1]))+
geom_bar(stat="identity",width=1)+
coord_polar(theta="y",direction=1)+
scale_fill_brewer(palette ="Set3",direction = 1)+
labs(x="",y="",fill="Type")+
ggtitle(label ="test",subtitle=NULL)
p_pie
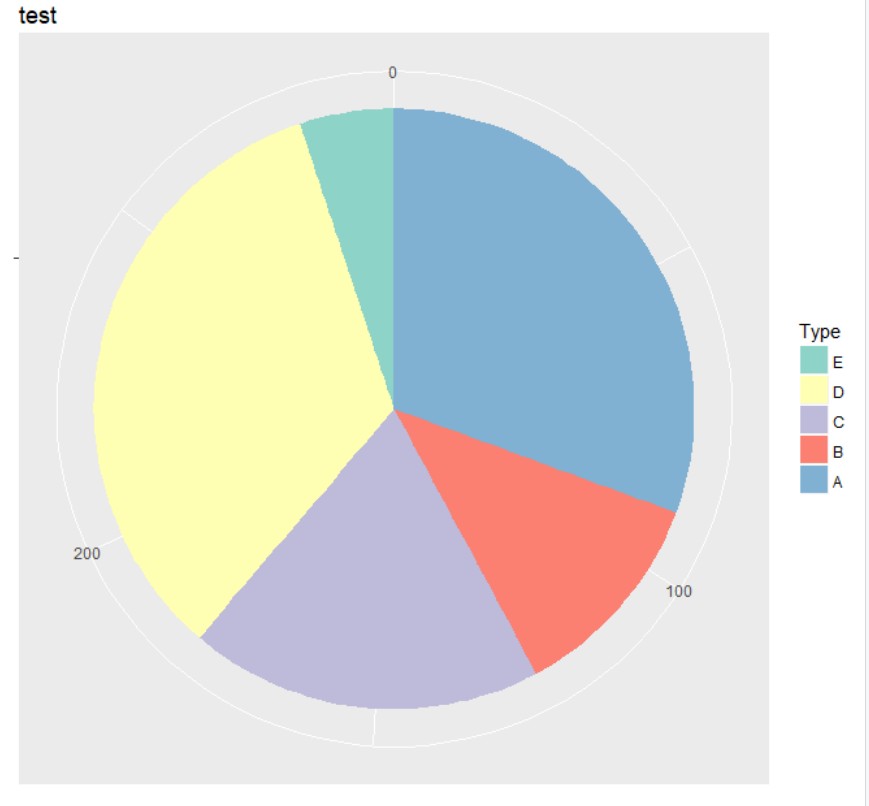 结合图片可以知道,第一个因子”E“对应了第一个颜色,不过可以看到,图片显示坐标中,"A"在前,而"A"在dat$Num中对应的数据也在前 90,这样计算位置就会发生改变了,90-90/2,”B”将对应90+34-34/2
结合图片可以知道,第一个因子”E“对应了第一个颜色,不过可以看到,图片显示坐标中,"A"在前,而"A"在dat$Num中对应的数据也在前 90,这样计算位置就会发生改变了,90-90/2,”B”将对应90+34-34/2
归纳为cumsum(dat$Num)-dat$Num/2
cumsum(dat$Num)-dat$Num/2
[1] 45.0 107.0 152.0 229.5 286.5
而且图例也是反向的,需要结合guides(fill=guide_legend(reverse=T)) 并且希望第一个颜色对应最后一个因子“A",scale_fill_brewer(palette ="Set3",direction = -1)
dat$type=factor(dat$type,levels = rev(dat$type),order=T)
dat$type
p_pie=ggplot(dat,aes(x="",y=dat[,2],fill=dat[,1]))+
geom_bar(stat="identity",width=1)+
coord_polar(theta="y",direction=1)+
scale_fill_brewer(palette ="Set3",direction = -1)+
labs(x="",y="",fill="Type")+
ggtitle(label ="test",subtitle=NULL)+
guides(fill=guide_legend(reverse = T))+
geom_text(aes(x=1.2,y=cumsum(dat$Num)-dat$Num/2 ,label=as.character(dat[,2])),size=3)
p_pie
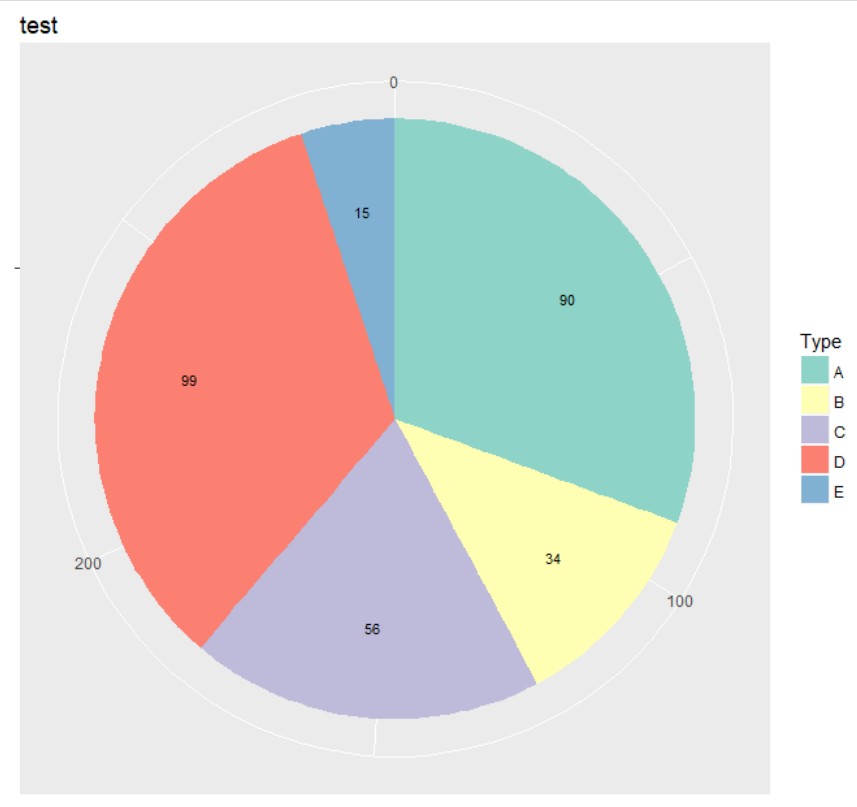 总结可知:ggplot2在画饼图的过程中设定填充的因子方向总和图片坐标中的方向相反,不过因子的顺序和数据dat$Num的对应关系是正向对应或者反向对应,会影响相关区块的中心位置值计算的方式,从而影响geom_text中文字定位。
总结可知:ggplot2在画饼图的过程中设定填充的因子方向总和图片坐标中的方向相反,不过因子的顺序和数据dat$Num的对应关系是正向对应或者反向对应,会影响相关区块的中心位置值计算的方式,从而影响geom_text中文字定位。
不借助公式计算的方法参考:
https://www.omicsclass.com/article/415
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-08-17 14:28
- 阅读 ( 9573 )
- 分类:R
你可能感兴趣的文章
- 不均等坐标系绘制 530 浏览
- ggplot 柱状图,y轴从0 开始,最高柱子 留有余地代码: 2026 浏览
- cnv_dumbbell_plot.r 绘制哑铃图ggplot2包 2721 浏览
- 绘制展示基因在样本中表达量与数量的柱状图 4060 浏览
- ggplot中用字符窜传递变量名称列名等 2451 浏览
- ggplot2中各种主题元素修改对应方法函数速查表 4548 浏览
