ggplot2几个主题类型
演示ggplot2几个默认的主题类型,直接套用对应的函数即可
数据参考
https://www.omicsclass.com/article/92
dat
type Sample Num
1 A sample1 90
2 B sample1 34
3 C sample1 56
4 D sample1 99
5 E sample1 15
6 A sample2 50
7 B sample2 20
8 C sample2 24
9 D sample2 70
10 E sample2 14
基础绘图,如果不设置theme具体的参数:
p = ggplot(dat, aes(x = type,y = Num,fill = Sample))+
#####这部分的position = "dodge",并排肩并肩的柱状图
geom_bar(stat ="identity",width = 0.6,position = "dodge")+
scale_fill_manual(values = c("red","blue"))+ #########设定颜色
labs(x = "",y = "", title = "test")+ ############坐标标签和图片title
geom_text(aes(label = dat$Num),position=position_dodge(width = 0.5),size = 5,vjust = -0.25)
p
获得结果如下:
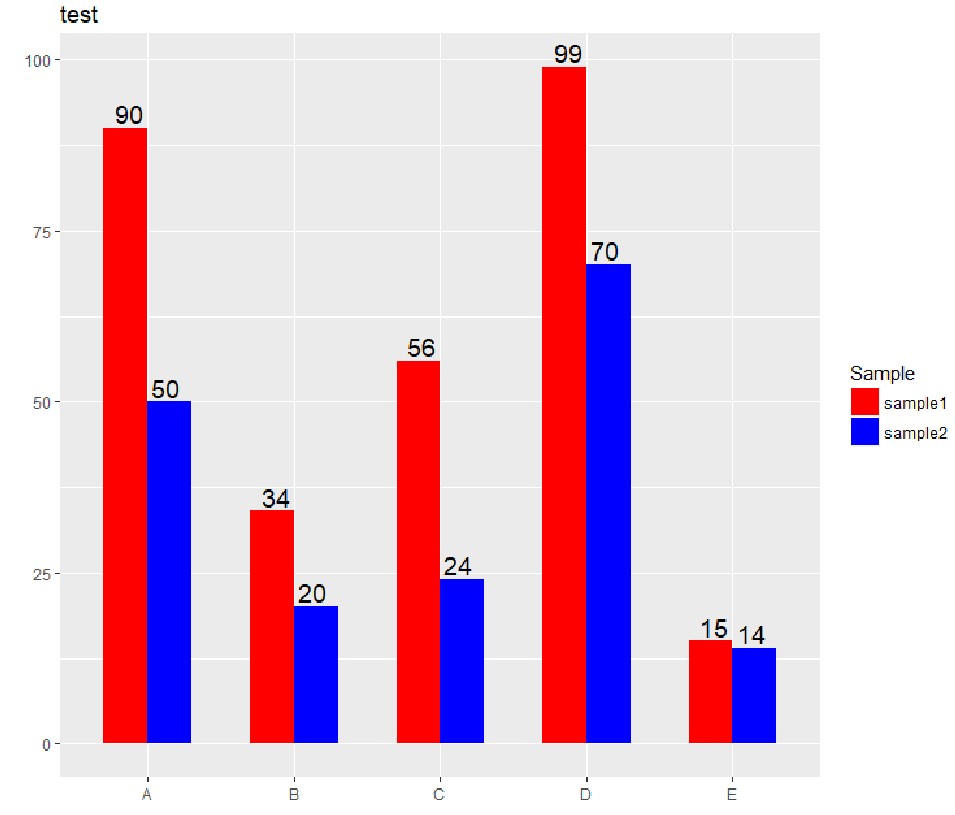
套用theme_bw():
p1=p+theme_bw()
p1
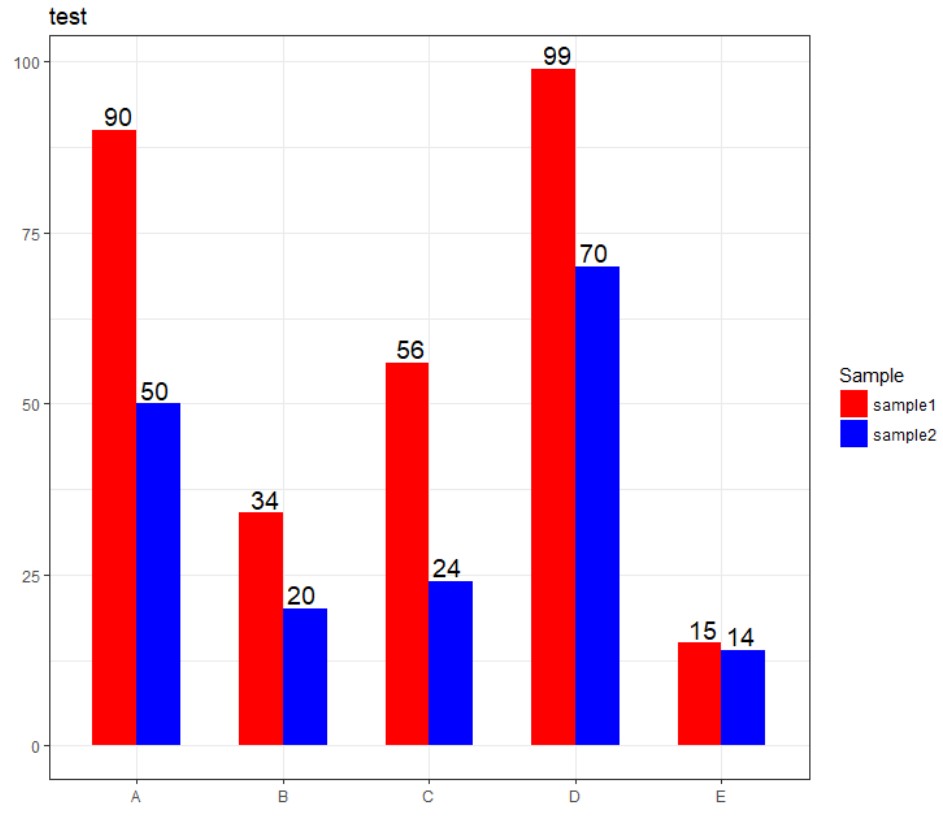 套用theme_classic():
套用theme_classic():
p2=p+theme_classic()
p2
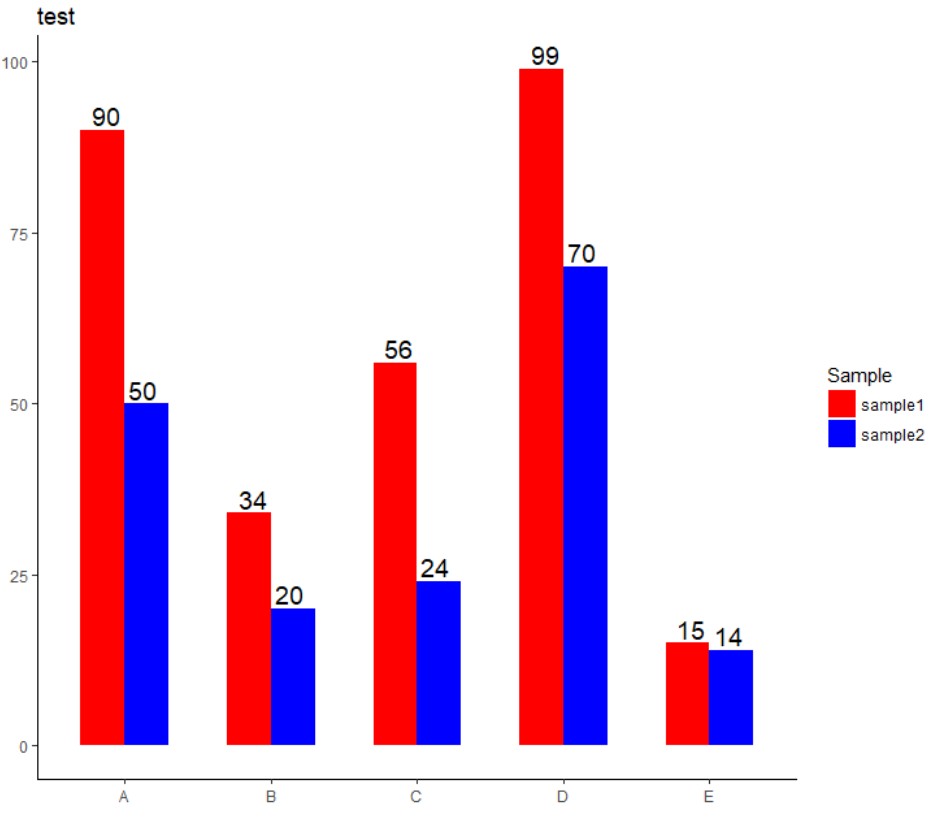 套用theme_void():
套用theme_void():
p3=p+theme_void()
p3
其他的还包括多种:
theme_dark() theme_grey() theme_gray() theme_light() theme_get() theme_linedraw() ....等等,具体在在RGUI 或者Rstudio中查看
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-08-17 16:33
- 阅读 ( 4868 )
- 分类:R
你可能感兴趣的文章
- 不均等坐标系绘制 1335 浏览
- ggplot 柱状图,y轴从0 开始,最高柱子 留有余地代码: 2801 浏览
- cnv_dumbbell_plot.r 绘制哑铃图ggplot2包 3152 浏览
- 绘制展示基因在样本中表达量与数量的柱状图 4622 浏览
- ggplot中用字符窜传递变量名称列名等 2909 浏览
- ggplot2中各种主题元素修改对应方法函数速查表 5080 浏览
