怎么做一张好看的分类学树状图
在做微生物多样性分析时,经常会接触到分类学树状图。今天分享一款很不错的分类学组成信息可视化软件——GraPhlAn,用它生成的图会更好看哦!
在做微生物多样性分析时,经常会接触到分类学树状图。今天分享一款很不错的分类学组成信息可视化软件——GraPhlAn,用它生成的图会更好看哦!
GraPhlAn 安装
首先,是GraPhlAn 的下载安装。
在linux环境下使用如下命令进行下载:
hg clone https://hg@bitbucket.org/nsegata/graphlan
然后将路径添加到bash配置文件中,之后就可以使用了。命令如下:
export PATH=`pwd`/graphlan/:$PATH
GraPhlAn 主程序
GraPhlAn有两个主要的脚本:
graphlan_annotate.py
graphlan.py
其中第一个(graphlan_annotate.py)用于将注释文件绑定到输入树。
graphlan_annotate.py --annot annotation_file.txt input_file.txt new_input_file_name.xml
注释文件可以与树输入文件多次绑定哦。
第二个(graphlan.py)用于生成不同格式的输出图像:png、pdf、ps、eps、svg。还可以使用 --dpi(默认值为72)和 --size(默认值为7.0,以英寸表示)来设置图像的分辨率和尺寸。用法如下:
graphlan.py new_input_file_name.xml image_name.png --dpi 150 --size 7
graphlan.py new_input_file_name.xml image_name.svg --dpi 150 --size 7
GraPhlAn运行
输入树文件(guide.txt 部分)的格式如下,各水平之间用 "." 分隔。
Bacillaceae.Anoxybacillus.Aflavithermus
Bacillaceae.Bacillus.Bamyloliquefaciens
Bacillaceae.Bacillus.Banthracis
Bacillaceae.Bacillus.Batrophaeus
Bacillaceae.Bacillus.Bcellulosilyticus
Bacillaceae.Bacillus.Bcereus
Bacillaceae.Bacillus.Bclausii
Bacillaceae.Bacillus.Bcoahuilensis
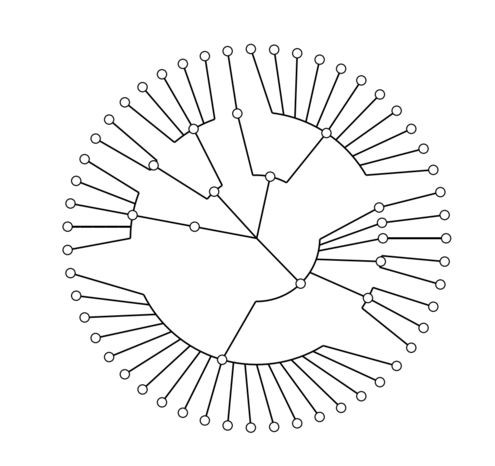
我们可以使用下面的命令做一张最基础的分类等级树。
graphlan.py guide.txt step_0.png --dpi 300 --size 3.5
例图如下:
之后可以用注释文件对该树进行修饰。注释文件(annot.txt 部分)的格式如下:
clade_separation 0.5
branch_thickness 1.5
branch_bracket_depth 0.8
branch_bracket_width 0.25
clade_marker_size 40
clade_marker_edge_color #555555
clade_marker_edge_width 1.2
[..]
Bacillus clade_marker_color b
Bacillus clade_marker_size 120
Bacillus clade_marker_shape h
[..]
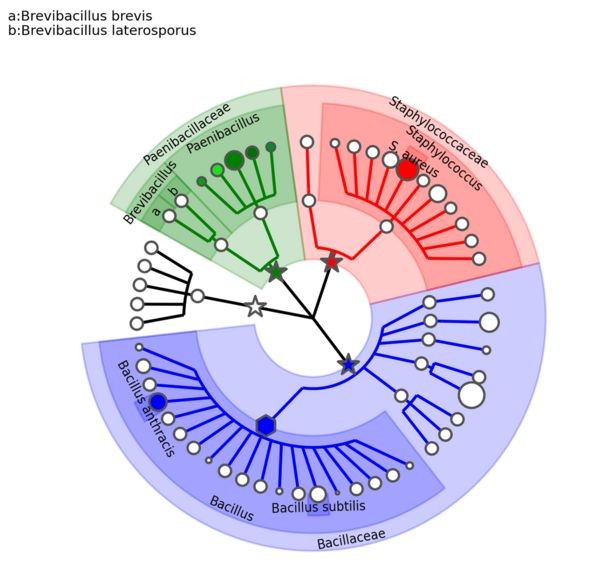
使用下面的命令对输入树文件进行注释,并用输出的guide.xml文件作图。
graphlan_annotate.py --annot annot.txt guide.txt guide.xml
graphlan.py guide.xml step_1.png --dpi 300 --size 3.5
修饰之后的分类等级树为:
图片不好看?因为只是示例图啊,下面才是实例图!
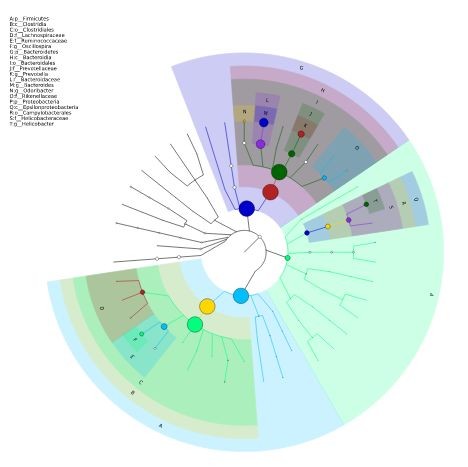
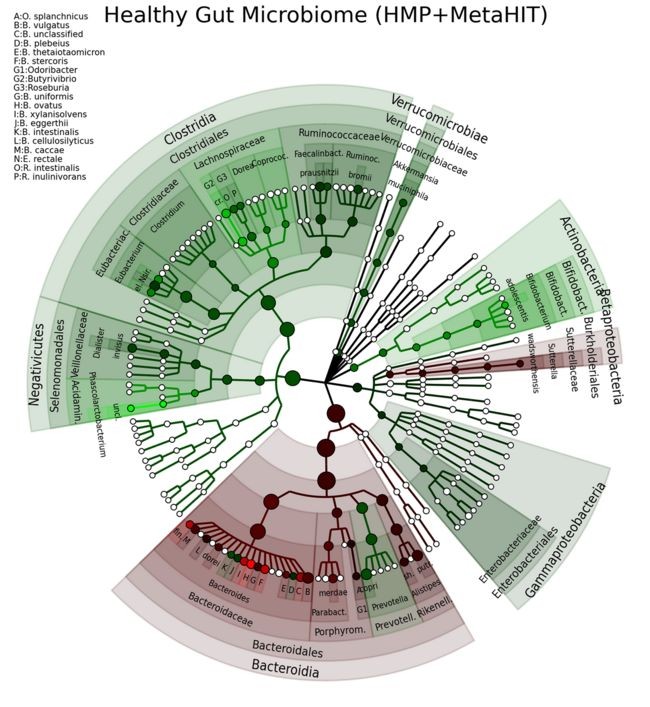
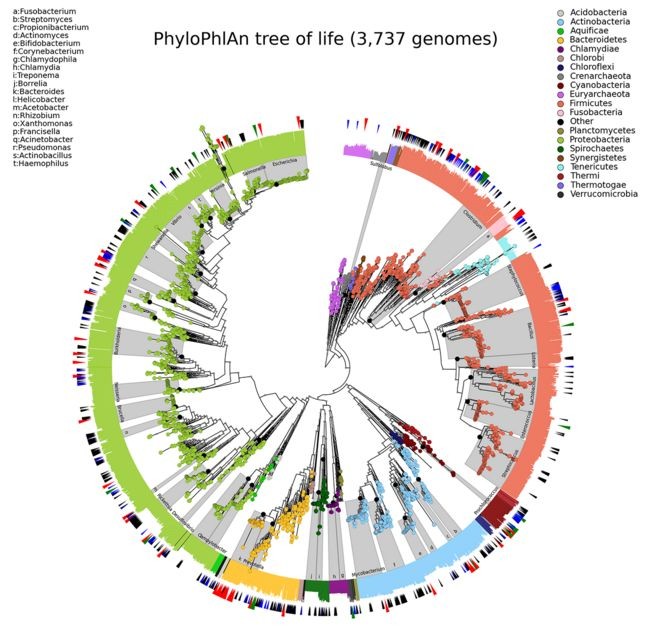
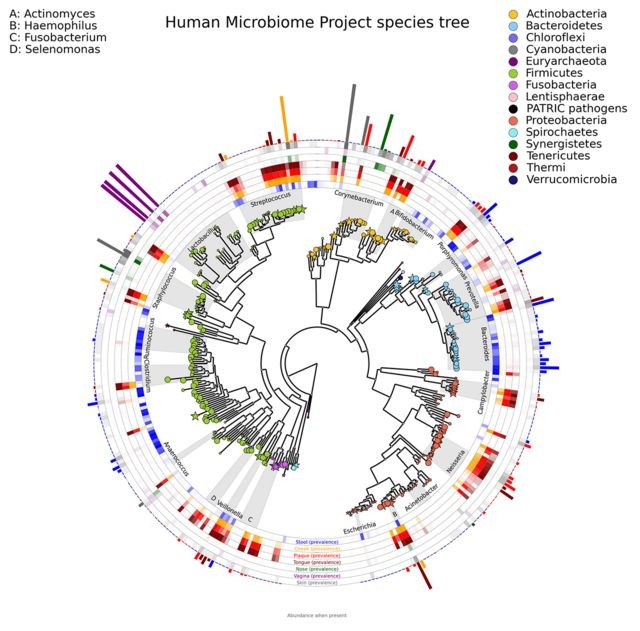
由于修饰文件的属性较多,所以这里就不一一介绍了,具体内容可在官网学习哦,说明很详细,小编做的时候就是按照人家的步骤来学习的。大家也可以根据自己的需要做出更个性化的图,相信肯定会为自己的文章增色不少。
链接送上:https://bitbucket.org/nsegata/graphlan/wiki/Home
- 发表于 2018-04-22 10:55
- 阅读 ( 7507 )
- 分类:软件工具
你可能感兴趣的文章
相关问题
0 条评论
请先 登录 后评论
