这种机器学习算法已经在微生物分析中成功应用
机器学习与微生物分析
去年随着人工智能概念的大热,机器学习也成了各路报道里的常见词。其实早在2012年《Nature》(Yatsunenko et al., 2012)上就有文章证明了某种机器学习算法能够对微生物群落样本进行有效且准确的分类,并且可以找出能够区分组间差异的关键成分(OTU或物种)。下面我们就来介绍这种算法的实现方式和应用(该分析已加入组学生物微生物多样性项目分析内容套餐)。
算法简介
随机森林是一种基于决策树的高效机器学习算法,可以用于对样本进行分类,也可以用于回归分析。它属于非线性分类器,因此可以挖掘变量之间复杂的非线性的相互依赖关系,对于经常呈现离散、不连续分布的微生物群落数据而言尤其适用。
简单的说,随机森林就是用随机的方式建立一个森林,森林里面有很多的决策树,并且每棵树之间是没有关联的。得到一个森林后,当有一个新的样本输入,森林中的每一棵决策树会分别进行一下判断,进行类别归类(针对分类算法),最后比较一下被判定哪一类最多,就预测该样本为哪一类。
具体实现过程
随机森林算法有两个主要环节:决策树的生长和投票过程。
(1)原始训练集为N,应用bootstrap法有放回地随机抽取k个新的自助样本集,并由此构建k棵分类树,每次未被抽到的样本组成了k个袋外数据;
(2)设有M个变量,则在每一棵树的每个节点处随机抽取m个变量(m < M),然后在m中选择一个最具有分类能力的变量,变量分类的阈值通过检查每一个分类点确定;
(3)每棵树最大限度地生长, 不做任何修剪;
(4)将生成的多棵分类树组成随机森林,用随机森林分类器对新的数据进行判别与分类,分类结果按树分类器的投票多少而定。
随机森林应用
在R语言中,我们调用randomForest包中的randomForest()函数来实现随机森林算法。
首先安装这个R包
install.packages("randomForest")
安装成功后,导入randomForset包
library(randomForset)
之后需要读入准备好的数据,使用的数据是属水平的微生物丰度矩阵,其中行为样本ID,列为物种名,以及最后一列样本分组(野生或温室)。然后就可以构建随机森林,代码如下:
random <- randomForest(Group ~ ., data=otu, importance=TRUE,proximity=TRUE,ntree= 1000)
#Group:为分类变量,即数据中的样本分组列名。
#otu:读入的数据集
# ntree:指定随机森林所包含的决策树数目,默认为500
#importance:逻辑参数,是否计算计算每个变量的重要性,
# proximity:逻辑参数,是否计算模型的临近矩阵,主要结合MDSplot()函数使用
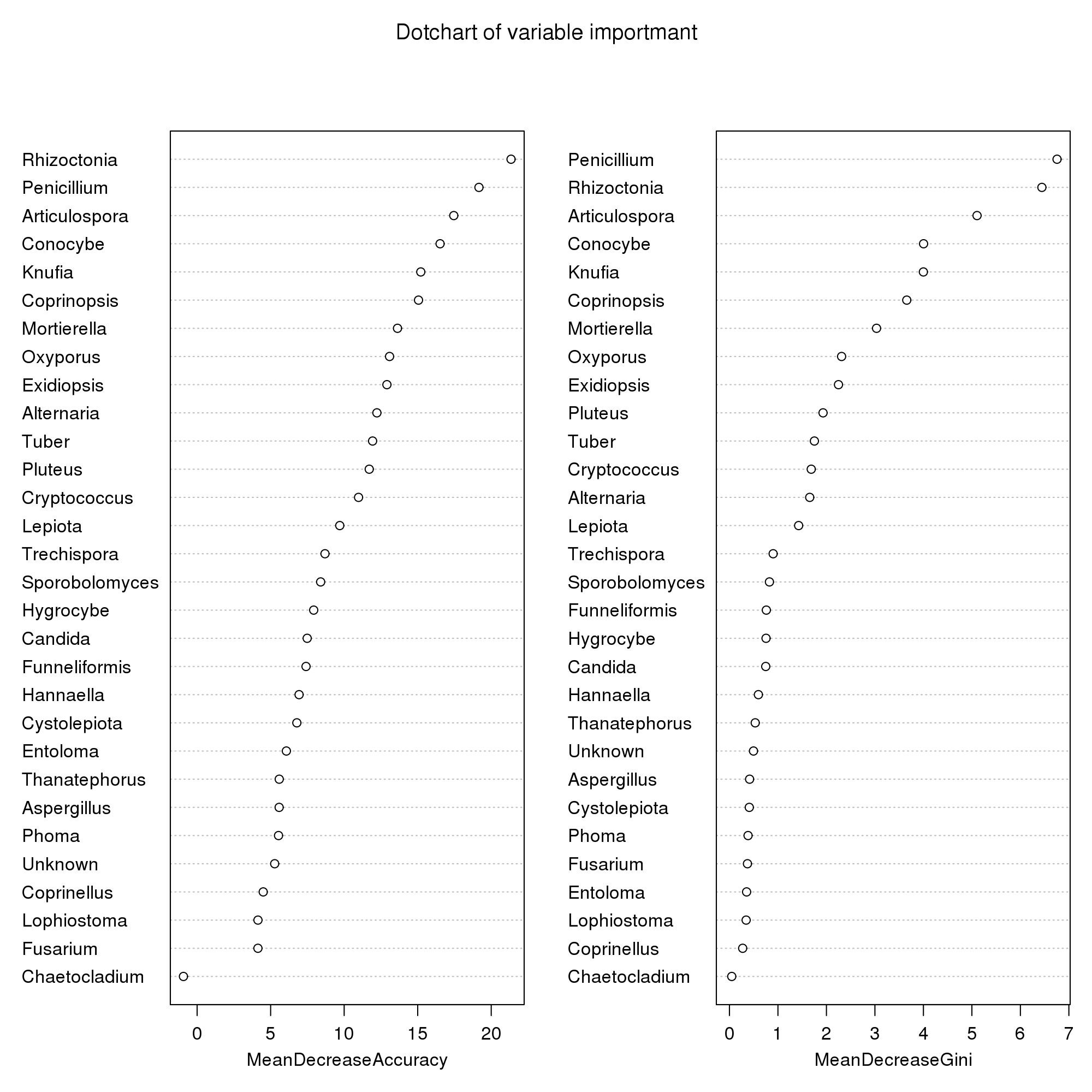
使用varImpPlot()函数可以查看变量重要性点图,默认显示对于分组贡献度最大的30个物种,并根据这30个物种的贡献度权重高低进行排序。
varImpPlot(random,main = "Dotchart of variable importmant")
用MDSPlot()函数可以绘制MDS二维图,不同颜色或形状的点代表不同环境或条件下的样本组,横、纵坐标轴的刻度是相对距离,无实际意义。Dim1、Dim2 分别代表对于两组样本微生物组成发生偏移的疑似影响因素,需要结合样本特征信息归纳总结。
MDSplot(random,otu$Group, palette = mycol, pch = as.numeric(otu$Group) )
#展示数据集在二维情况下各类别的具体分布情况
结果例图: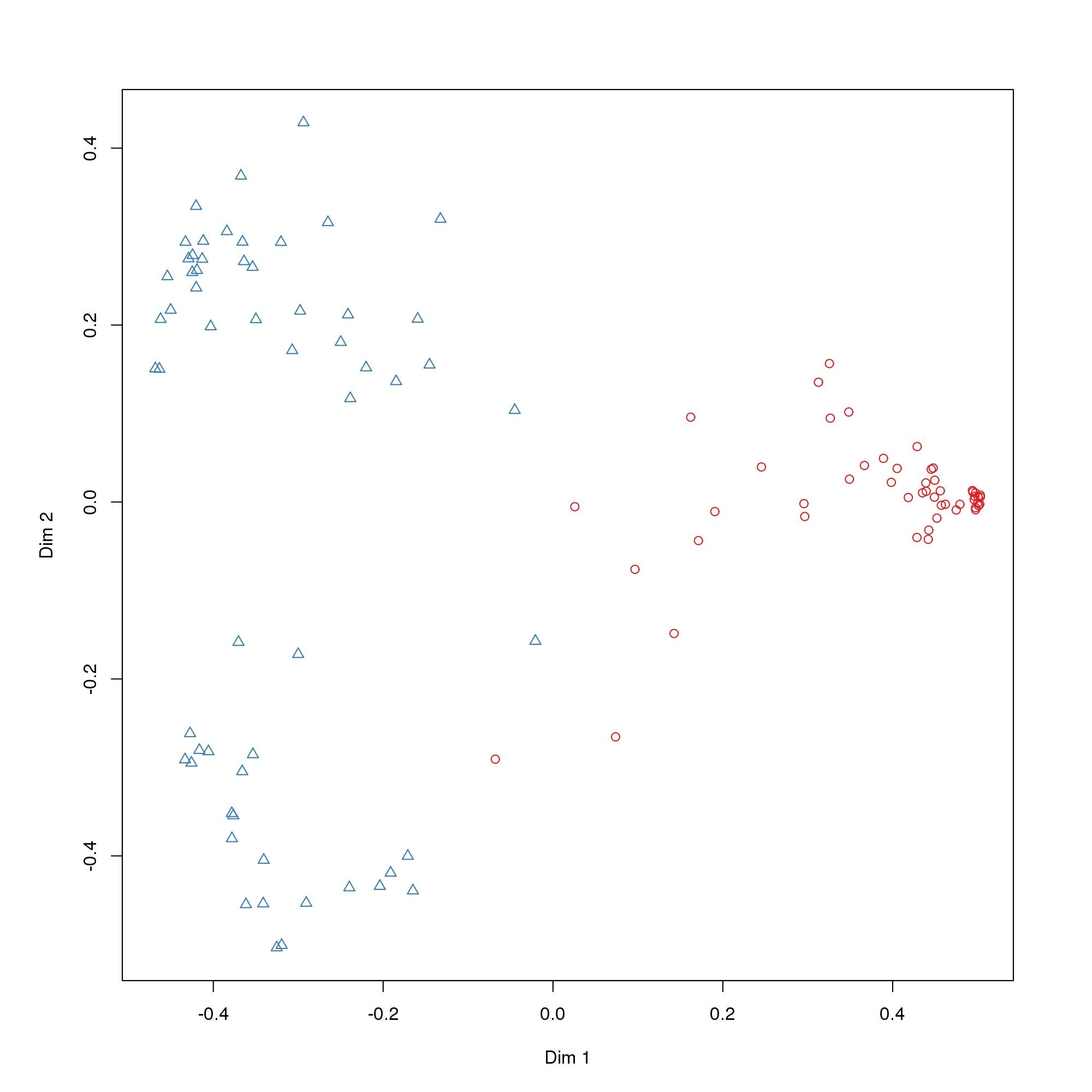
参考文献:
Yatsunenko, T., Rey, F.E., Manary, M.J., Trehan, I., Dominguez-Bello, M.G., Contreras, M., Magris, M., Hidalgo, G., Baldassano, R.N., Anokhin, A.P., et al. (2012). Human gut microbiome viewed across age and geography. Nature 486, 222-227.
- 发表于 2018-04-22 10:58
- 阅读 ( 6149 )
- 分类:R
你可能感兴趣的文章
- Root microbiota assembly and adaptive differentiation among European Arabidopsis populations 3373 浏览
- 微生物生态研究中常用数据库简介--转载 7545 浏览
- beta多样性中的距离矩阵总结 13817 浏览
- 微生物PCA分析和PCOA分析差别 23127 浏览
- Beta多样性分析 21535 浏览
相关问题
- 在进行物种累积丰度曲线时提醒没有该命令 1 回答
