直方图---ggplot2(每组内添加对应的count 或者density文本标签)
利用ggplot2绘制直方图,基于几何对象geom_histogram()函数
geom_histogram(mapping = NULL, data = NULL, stat = "bin", position = "stack", ..., binwidth = NULL, bins = NULL, na.rm = FALSE, show.legend = NA, inherit.aes = TRUE)
其中mapping图形属性映射涉及多个,需统计的数据映射到X轴,Y轴映射统计类型(包括频数count 或频率density等),还涉及其他的图像属性等。
统计变换 stat="bin",对应要设置组距间隔 binwidth大小,由此统计该组间间隔内的频数情况或频率情况
现利用数据绘制直方图,同时将绘图中计算得到的count添加到柱子上。
数据基本情况:
> head(dat)
ID CK
1 AT1G01010 3.581590
2 AT1G01020 5.036775
3 AT1G01030 5.035380
4 AT1G01040 2.692716
5 AT1G01046 0.000000
6 AT1G01050 114.336000
> dim(dat)
[1] 32483 2
数据第二列CK 表示基因表达量FPKM,再次进行转换log10(CK)进行绘图
基础代码:
p=ggplot(dat,aes(x=log10(CK)))+geom_histogram(stat="bin",binwidth = 1)+xlim(-3,5)
print(p)
其y轴默认count类型,不做设置是等同于 aes(y=..count..),对应频数类型
注意书写方式 该数值为统计变换所得衍生,故前后各有两点
p=ggplot(dat,aes(x=log10(CK),y=..count..))+geom_histogram(stat="bin",binwidth = 1)+xlim(-3,5)
print(p)
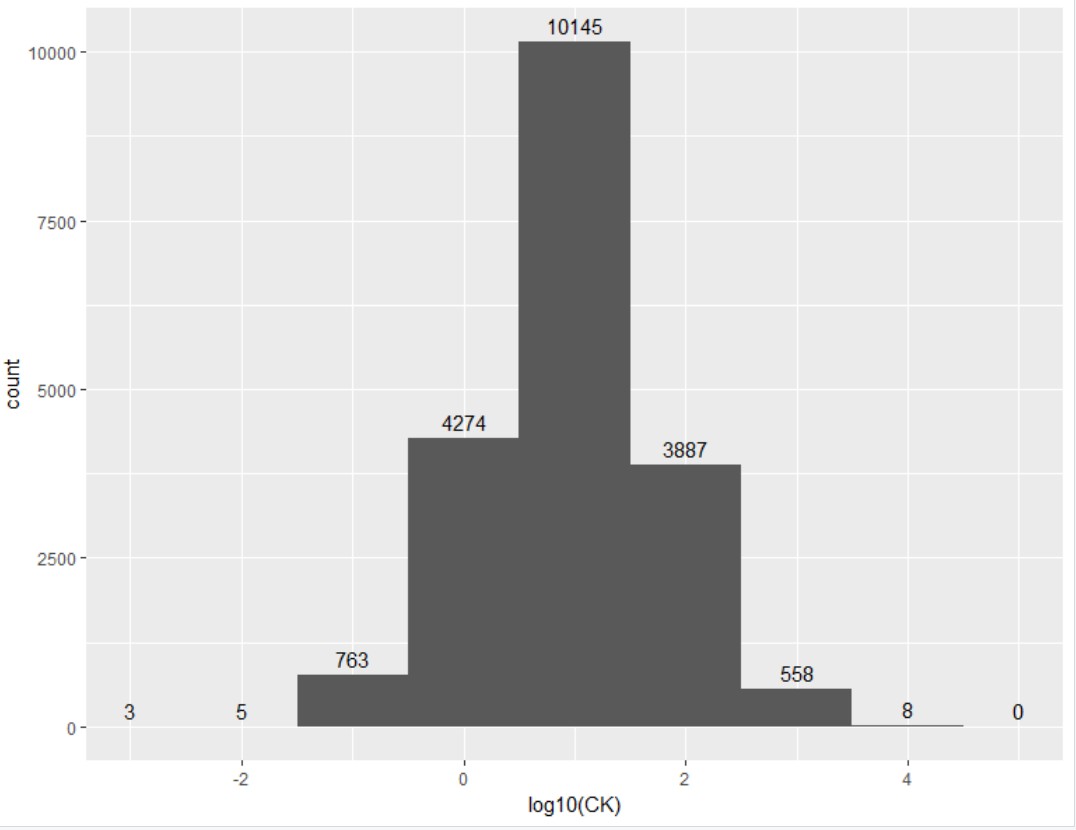
进行文字添加则利用geom_text函数,其aes(label)需由..count..映射,同时在stat 和binwidth的设置必须和此前一致,方可在图片上添加计算到的频数
p=p+geom_text(aes(label=as.character(..count..)),stat="bin",binwidth=1,vjust=-0.5)
print(p)
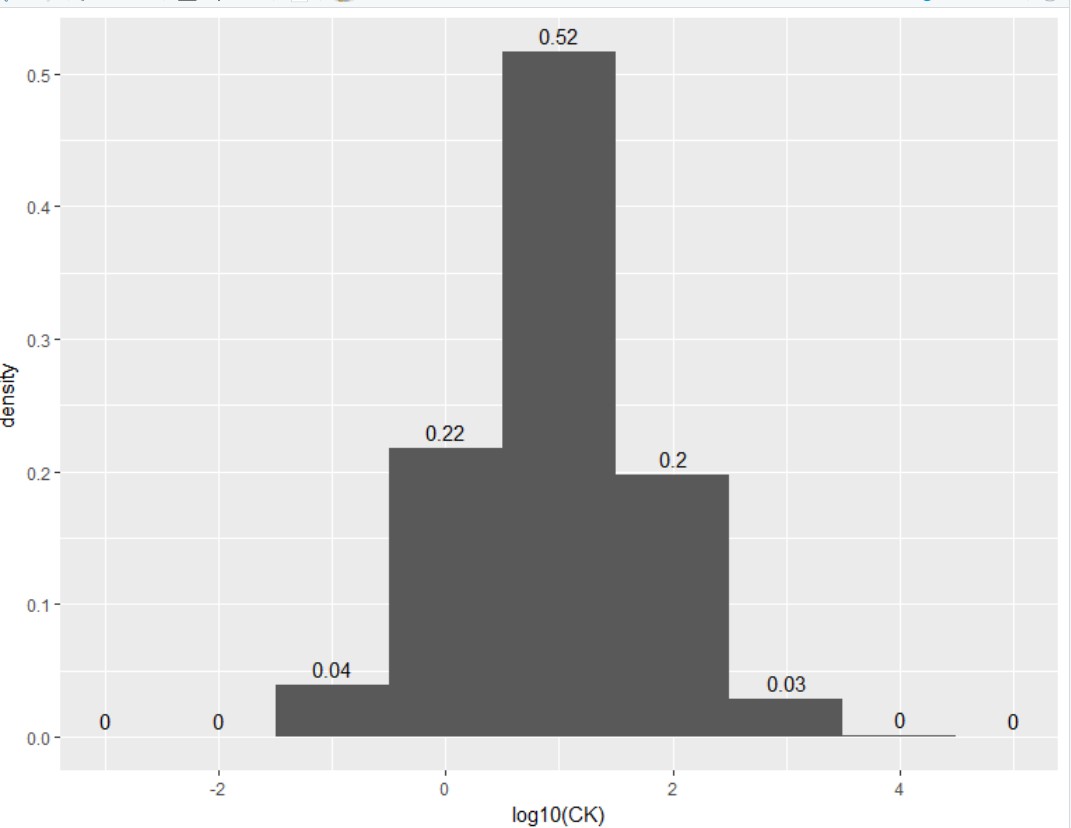
频率图绘制及density数值添加过程类似:1、aes(y=..density) 2、统计变换等一致 3、density 仅保留两位小数利用round()
p=ggplot(dat,aes(x=log10(CK),y=..density..))+geom_histogram(stat="bin",binwidth = 1)+xlim(-3,5)
p=p+geom_text(aes(label=as.character(round(..density..,2))),stat="bin",binwidth=1,vjust=-0.5)
print(p)
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-09-14 00:08
- 阅读 ( 18760 )
- 分类:R
你可能感兴趣的文章
- 不均等坐标系绘制 1530 浏览
- ggplot 柱状图,y轴从0 开始,最高柱子 留有余地代码: 2843 浏览
- cnv_dumbbell_plot.r 绘制哑铃图ggplot2包 3173 浏览
- 绘制展示基因在样本中表达量与数量的柱状图 4649 浏览
- ggplot中用字符窜传递变量名称列名等 2924 浏览
- ggplot2中各种主题元素修改对应方法函数速查表 5103 浏览
