qRT-PCR相对定量计算详解 qPCR
做完转录组分析之后,一般都要求做qRT-PCR来验证二代测序得到的转录本表达是否可靠。荧光定量PCR是一种相对表达定量的方法,他的计算方法有很多,常用的相对定量数据分析方法有双标曲线法,ΔCt法,2^-ΔΔCt法(Livak法),用参照基因的ΔCt法和Pfaffl法。这里主要讲解常用的2^-ΔΔCt法(Livak法)如何计算;
qRT-PCR原理:
以基因的cDNA为模板进行PCR扩增,在PCR扩增过程中,通过收集荧光信号,对PCR进程进行实时检测。由于在PCR扩增的指数时期,模板的Ct 值和该模板的起始拷贝数存在线性关系,所以可以定量。
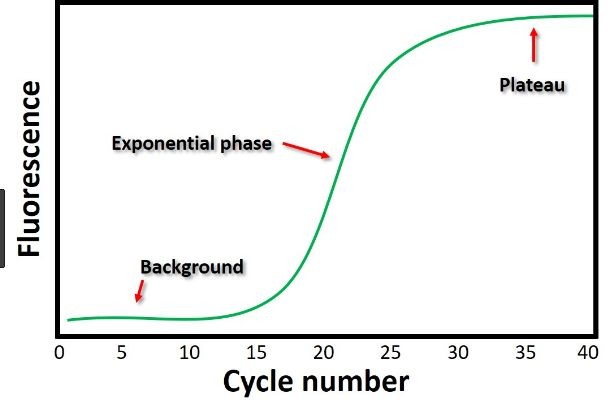
Ct值是什么意思呢?
Ct 值的含义是:每个反应管内的荧光信号达到设定的域值时所经历的循环数 (cycle)。 qRT-PCR在扩增的时候都会有平台期,在平台期之前,PCR 扩增就是简单的指数增长,也就是 1 变 2,2 变 4,4 变 8 …扩增。数学形式就是 2 的 ct 次方,到了平台期所有基因扩增的数目是一致的,而唯一有区别的则是 ct 值的不同。所以不难推断出 ct 值越小,反应扩增到达平台期所需循环数越少,目的基因起始含量越高。这里可以得到公式:

计算-ΔΔCt
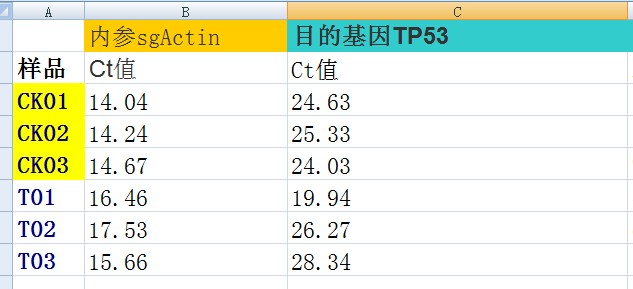
在这里,我们有一个对照组,一个处理组,还有一个内参基因和目的基因,想看一下目的基因在处理组中相对与对照中的表达差异,也就是计算-ΔΔCt:数据如下:
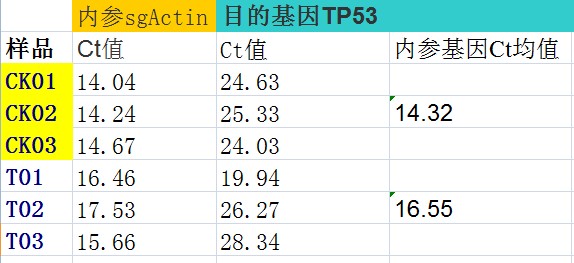
1.计算每组内参基因sgAction Ct均值
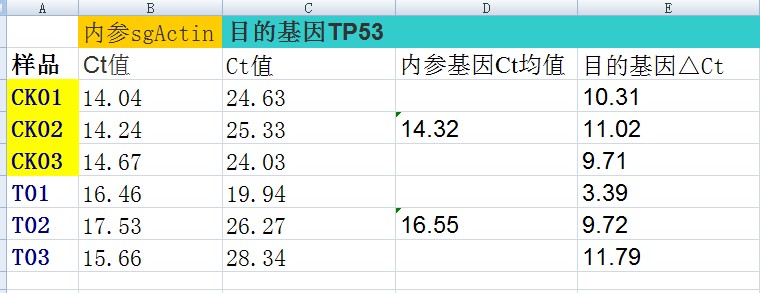
计算第一个 Δct,即每组的待检目的基因减去内参基因的 Ct 值
3.计算对照CK组中 Δct 的均值,再用处理组的 每一个Δct 减去刚刚计算的对照CK组的 Δct 均值,得到 ΔΔct(红框框)
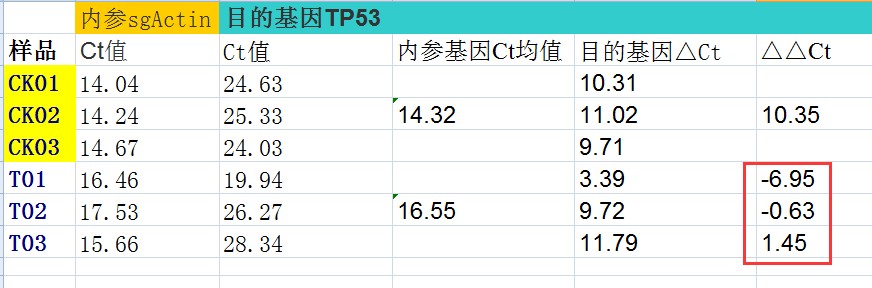
4.相对表达量计算,也就是相对于对照组: 2^-ΔΔct:
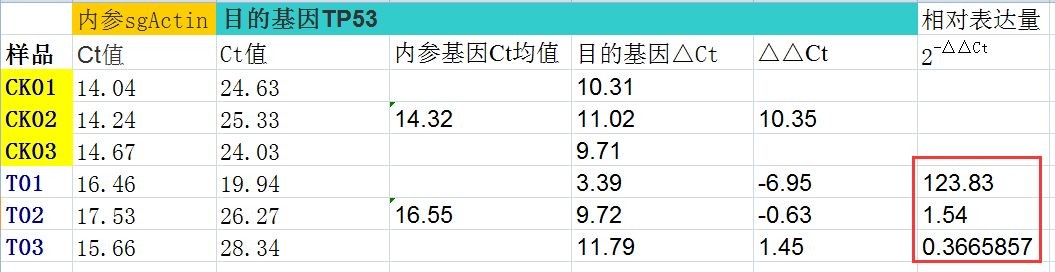
不难看出这里的-ΔΔct和我们转录组当中的log2(fold change)值是一致的,所以如果多做几个基因就可以绘制类似如下图:
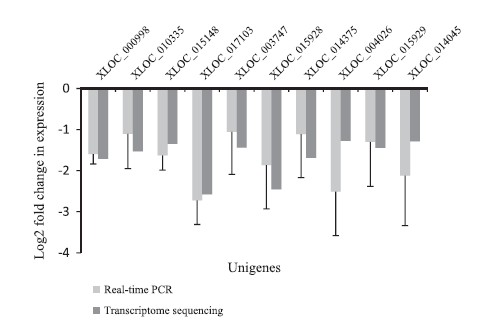
或者相关性点图:
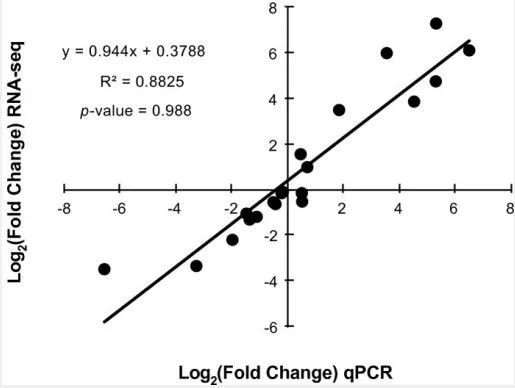
测试数据表格下载:qRT-PCR_demo_data.xlsx
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、docker搭建生物信息分析环境、实验室linux生信分析平台搭建、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高、python语言入门到精通
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,NCBI数据上传、二代fastq测序数据解读、
9,高级生物信息分析课程:重测序数据自主分析、二代测序转录组数据自主分析、微生物扩增子分析课程实操、群体遗传进化分析、GWAS全基因组关联分析
10.全部课程可点击:组学大讲堂视频课程
- 发表于 2018-09-20 20:11
- 阅读 ( 111132 )
- 分类:实验相关

