详细介绍如何利用iTOL在进化树中用颜色体现样品分组信息
前不久有小伙伴问到怎样才能在进化树中体现样品的分组,其实很简单,小编就来讲讲怎样实现吧。
使用的工具是iTOL这款在线软件,可点这里 绘一棵超酷炫的系统发育树 了解iTOL的使用方法。基础的使用方法就不再讲述了,只对配置文件进行说明。
方法一:标签颜色区分样品分组
利用标签的颜色来区分样品分组,不同颜色代表不同的分组。如下所示:
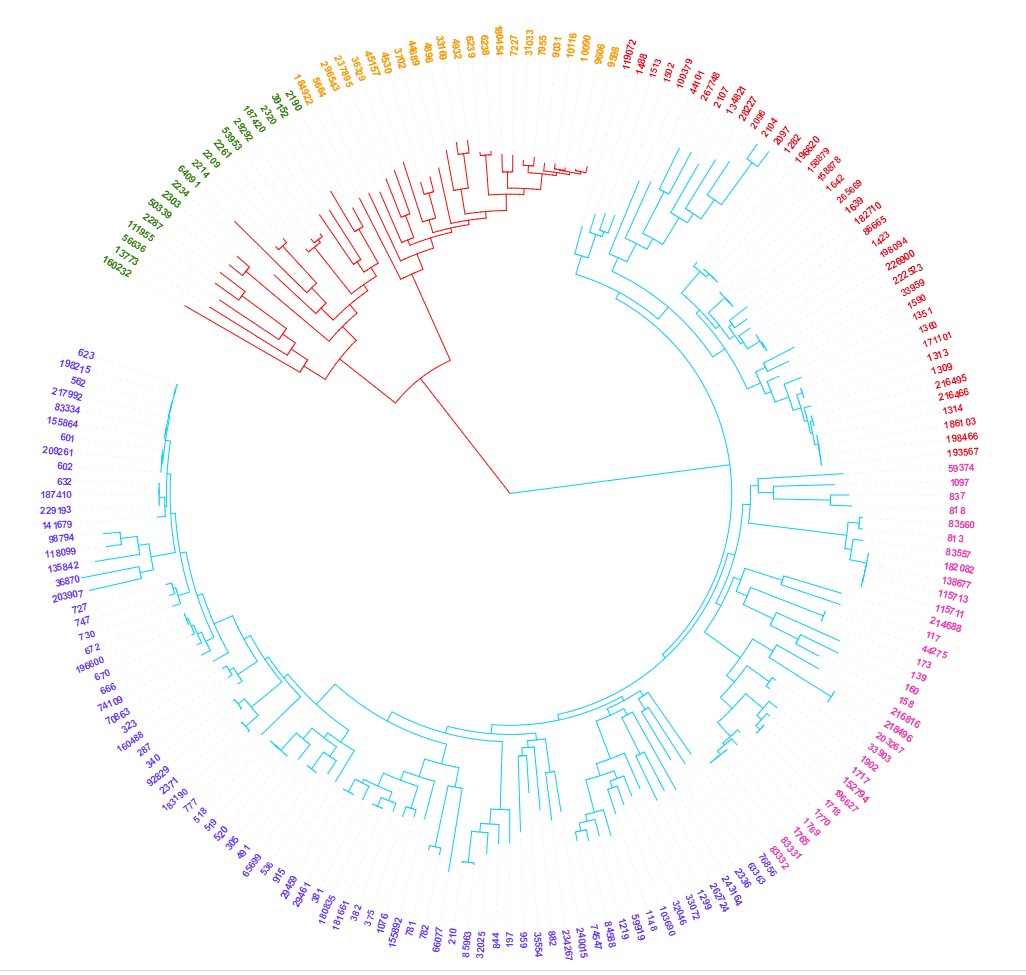
要实现上方进化树图中对每一个样品或基因名称设置不同字体颜色用以分组,需要准备以下配置文件,其结构如下:
TREE_COLORS
SEPARATOR TAB
DATA
155864 label #6633ff bold 1.5
83334 label #6633ff bold 1.5
217992 label #6633ff bold 1.5
562 label #6633ff bold 1.5
...
181661 label #6633ff bold 1.5
180835 label #6633ff bold 1.5
382 label #6633ff bold 1.5
29461 label #6633ff bold 1.5
前三行为固定格式,可不用修改,DATA后就是我们要添加的注释信息了。这里总共分5列信息,每列之间使用Tab制表符分隔(由第二行"SEPARATOR TAB"所设置):
第一列:进化树中物种、样品或基因的名称;
第二列:设置修改类型为label;
第三列:为每一样品设置对应颜色;
第四列:标签的字体,如bold表示使用粗体;
第五列:对标签字体的大小进行修改(数值越大,字体越大)。
同一分组的我们给设成同一种颜色。这样就很容易在进化树中区分不同分组了。
方法二:标签背景色区分样品分组
同样的,也可以利用标签的背景色区分样品的分组,不同颜色代表不同的分组。如下所示:
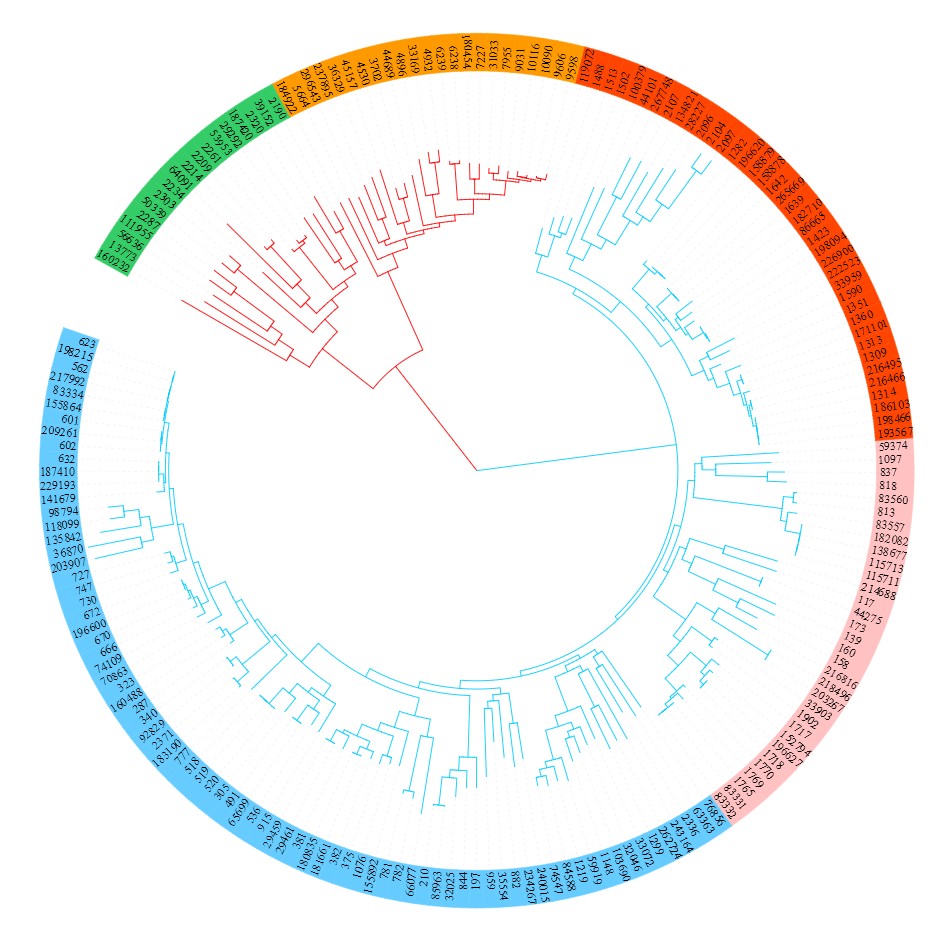
要实现上方进化树图中对每一个样品或基因名称设置不同背景颜色用以分组,需要准备以下配置文件,其结构如下:
TREE_COLORS
SEPARATOR TAB
DATA
155864 range #6633ff test1
83334 range #6633ff test1
217992 range #6633ff test1
...
181661 range #6633ff test2
180835 range #6633ff test2
382 range #6633ff test2
文件格式与之前的配置文件类似,所不同的是这里只有四列:
第一列:进化树中物种、样品或基因的名称;
第二列:设置修改类型为range(分枝上添加一个color range,即整个分枝添加背景色,但默认只显示标签背景色);
第三列:为每一样品设置对应的背景颜色;
第四列:设置color range 的标签,这里用处不大。
枝的颜色设置
此外,还可以对进化树的枝的颜色进行设置,其 配置文件 如下:
I149 clade #00ccff normal 2
I189 clade #FF0000 normal 2
第一列:进化树中节点ID。iTOL在导入nwk文件时会为每个节点分配一个Node id,可以点击进化树的节点查看,如下:
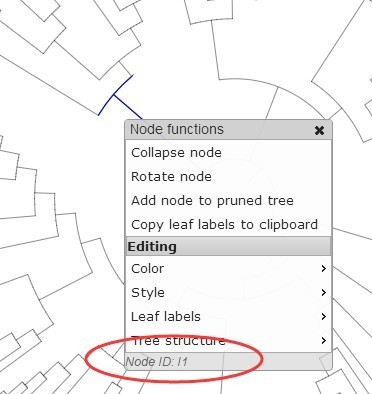
第二列:设置修改类型为clade;
第三列:设置枝的颜色;
第四列:设置线条类型,这里用的实线;
第五列:设置枝的粗细,数值越大,线条越粗。
以上操作介绍完了,怎么样?是不是很简单?有兴趣的小伙伴自己动手试试吧!
最后祝您科研愉快!
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2018-09-21 13:19
- 阅读 ( 29427 )
- 分类:软件工具
你可能感兴趣的文章
- 给进化树标定化石时间 2202 浏览
- 快速生成iTOL配置文件美化你的进化树 2412 浏览
- itol添加圆环分组和节点标签时位置调整 4911 浏览
- 如何用R语言绘制进化树+热图 10377 浏览
- 进化树构建的方法及原理详解 43076 浏览
- 如何利用iTOL绘制漂亮的进化树 17102 浏览
