ggplot2-气泡图(控制点的大小范围)
Pathway富集结果常用气泡图来显示,其基本代码如下:
案例数据:
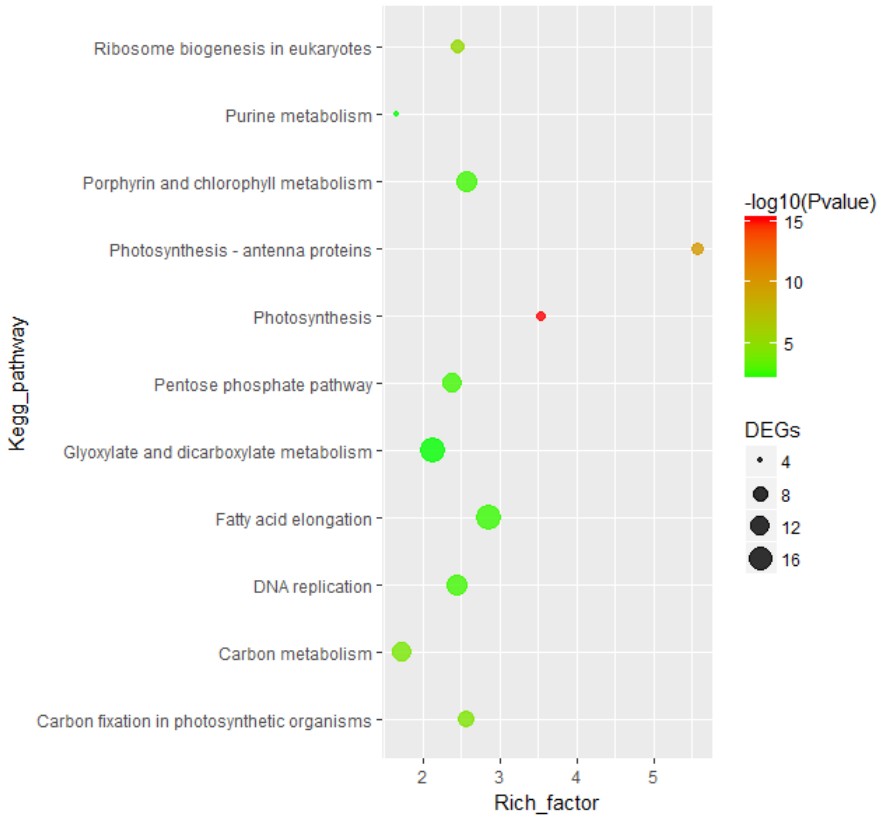
> dat ko_id Kegg_pathway Rich_factor Pvalue DEGs1 ko00195 ...
Pathway富集结果常用气泡图来显示,其基本代码如下:
案例数据:
> dat
ko_id Kegg_pathway Rich_factor Pvalue DEGs
1 ko00195 Photosynthesis 3.543363 1.000000e-15 5
2 ko00196 Photosynthesis - antenna proteins 5.575221 1.500000e-10 6
3 ko03008 Ribosome biogenesis in eukaryotes 2.464696 4.920000e-06 7
4 ko00710 Carbon fixation in photosynthetic organisms 2.567654 4.510000e-05 9
5 ko01200 Carbon metabolism 1.738592 7.350000e-05 11
6 ko03030 DNA replication 2.453097 1.772599e-03 14
7 ko00030 Pentose phosphate pathway 2.397575 1.772599e-03 12
8 ko00860 Porphyrin and chlorophyll metabolism 2.574238 1.772599e-03 14
9 ko00062 Fatty acid elongation 2.857551 2.316650e-03 18
10 ko00630 Glyoxylate and dicarboxylate metabolism 2.129425 5.299956e-03 19
11 ko00230 Purine metabolism 1.671398 5.299956e-03 4
绘图常利用geom_point几何对象函数实现气泡图绘制过程,同时实现坐标轴翻转
注意显著性大小-log10(Pvalue)映射到的点颜色上、差异基因数映射到点大小上。
kegg_point=ggplot(dat,aes(x=Kegg_pathway,y=Rich_factor))+
geom_point(aes(color=-log10(Pvalue),size=DEGs),alpha=0.8)+
coord_flip()+
scale_color_gradient(low = "green",high = "red")
kegg_point
 不过需要注意的是在部分情况下,对应的DEGs数量较小,自动匹配的点的大小较小,在图片上难以显示明显,可以利用scale_size_continuous(range)参数控制,
不过需要注意的是在部分情况下,对应的DEGs数量较小,自动匹配的点的大小较小,在图片上难以显示明显,可以利用scale_size_continuous(range)参数控制,
譬如:
kegg_point=kegg_point+scale_size_continuous(range=c(4,10))
kegg_point
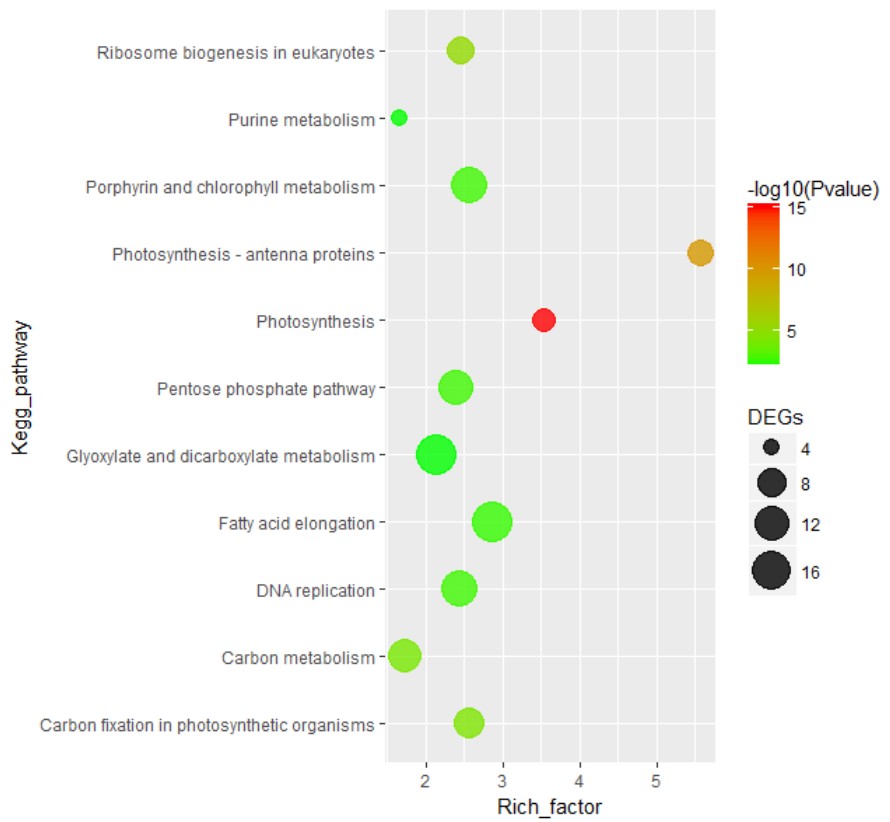
size对应的range参数可以有效控制点大小的范围。
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-09-21 15:04
- 阅读 ( 23881 )
- 分类:R
你可能感兴趣的文章
- 不均等坐标系绘制 523 浏览
- ggplot 柱状图,y轴从0 开始,最高柱子 留有余地代码: 2012 浏览
- cnv_dumbbell_plot.r 绘制哑铃图ggplot2包 2714 浏览
- 绘制展示基因在样本中表达量与数量的柱状图 4044 浏览
- ggplot中用字符窜传递变量名称列名等 2444 浏览
- ggplot2中各种主题元素修改对应方法函数速查表 4539 浏览
0 条评论
请先 登录 后评论
