ggplot2控制坐标轴截距交点位置(原点)
ggplot2绘图结果往往X轴和Y轴的截距交点往往不是原点:譬如下图y轴就不是起始于0
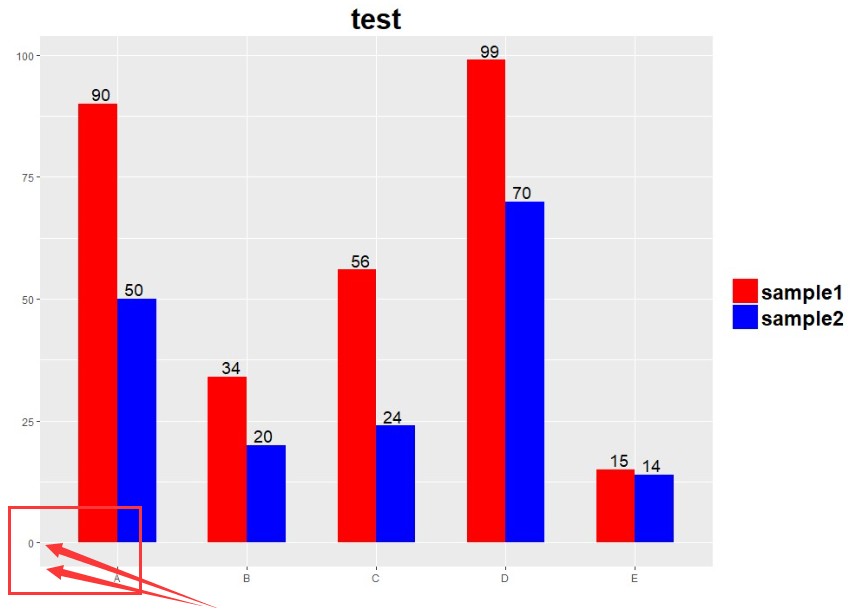 调整坐标起始位点可以利用scale_y_continuous(expand = c(0, 0))或者scale_x_continuous(expand = c(0, 0))
调整坐标起始位点可以利用scale_y_continuous(expand = c(0, 0))或者scale_x_continuous(expand = c(0, 0))
expand的解释如下
| expand | A numeric vector of length two giving multiplicative and additive expansion constants. These constants ensure that the data is placed some distance away from the axes. The defaults are c(0.05, 0) for continuous variables, and c(0, 0.6)for discrete variables. 连续性和离散型都涉及,默认值不同,离散型scale_y_discrete 或者scale_x_discrete,这里针对连续性进行介绍: |
参考画图数据和代码 https://www.omicsclass.com/article/92
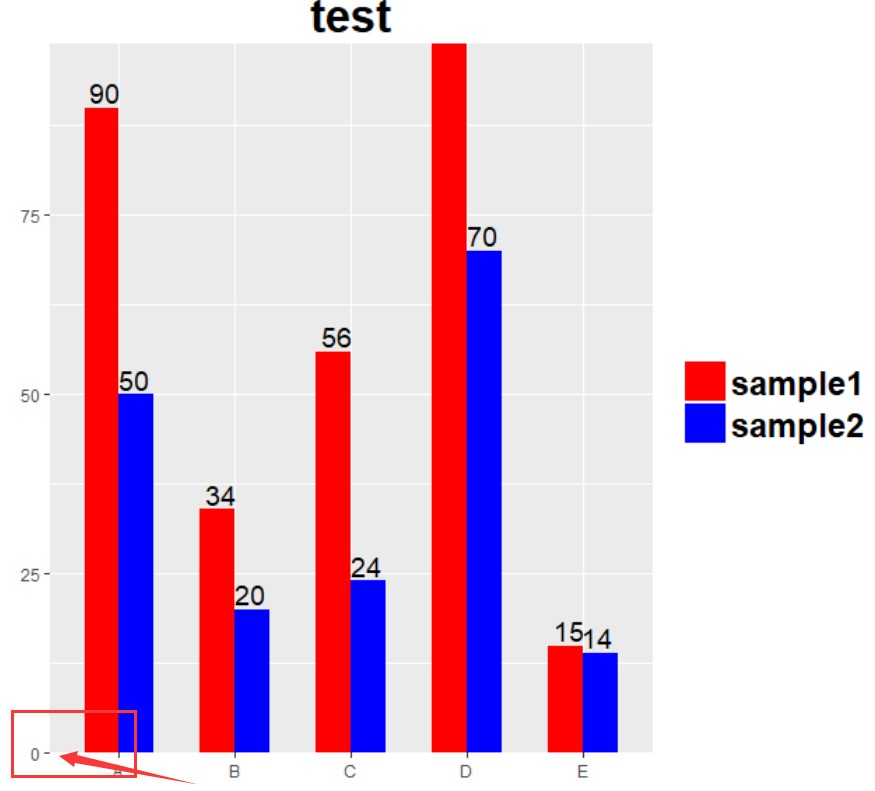
修改y轴连续性坐标起点:
Y=p+
scale_y_continuous(expand=c(0,0))
Y
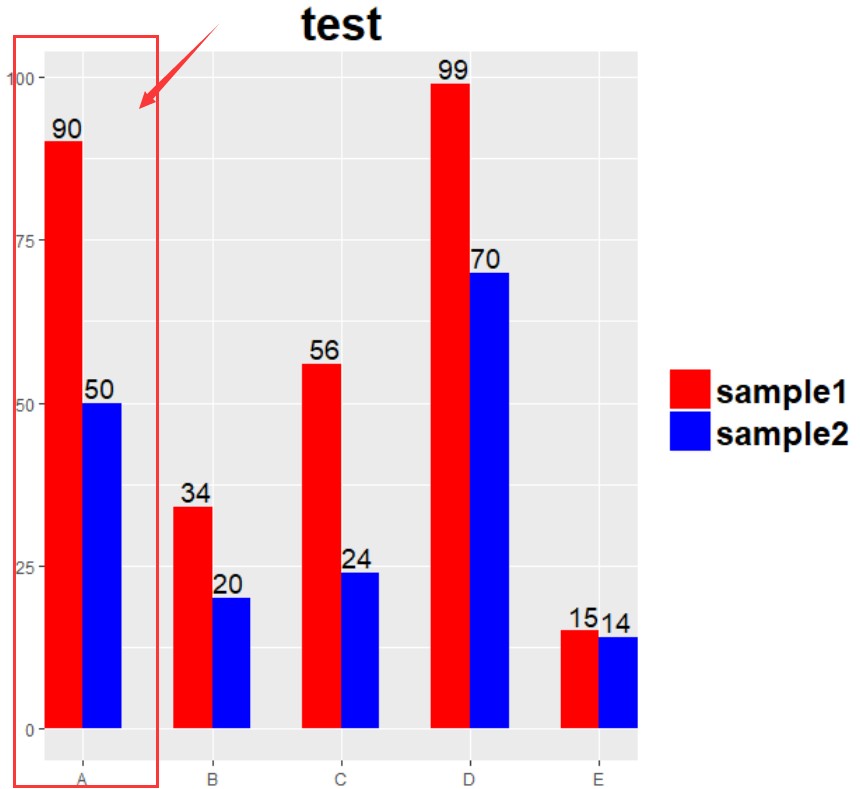
如果针对x离散型的调整呢?尝试调整数,使柱子紧贴Y轴
X=p+
scale_x_discrete(expand=c(0,0))
X
如果想提升自己的绘图技能,我们推荐:R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-09-21 17:05
- 阅读 ( 17941 )
- 分类:R
你可能感兴趣的文章
- 不均等坐标系绘制 523 浏览
- ggplot 柱状图,y轴从0 开始,最高柱子 留有余地代码: 2012 浏览
- cnv_dumbbell_plot.r 绘制哑铃图ggplot2包 2714 浏览
- 绘制展示基因在样本中表达量与数量的柱状图 4045 浏览
- ggplot中用字符窜传递变量名称列名等 2444 浏览
- ggplot2中各种主题元素修改对应方法函数速查表 4540 浏览
