全外显子测序疾病相关基因变异位点过滤标准及参考文献
1.基于跨物种位点保守性过滤
SIFT, Polyphen2, LRT, Grantham scale, Mutation Taster and PhyloP
可以用ANNOVAR一站式注释dbNSFP,各种方法的算法参见文章,使用说明见ANNOVAR官网:ljb
2.基于位点的变异频率的稀有性
filtering by MAF < 5% (ESP and 1000G),
nonsynonymous changes, splicing site alterations, inheritance modeling in a trio setting and included genes in HGMD and OMIM with phenotypic overlap
3.根据变异对基因编码蛋白的影响
这边首先利用ANNOVAR软件对变异位点所在的区域进行注释,看变异所处的位置与基因的关系:(exonic, splicing, UTR5, UTR3, intronic, ncRNA_exonic, ncRNA_intronic, ncRNA_UTR3, ncRNA_UTR5, ncRNA _splicing, upstream, downstream, intergenic)。一般会关注编码蛋白基因外显子上的非同义突变,移码突变,引起剪切改变splicing突变,stopgain,stoploss突变等
4.根据家系遗传特点
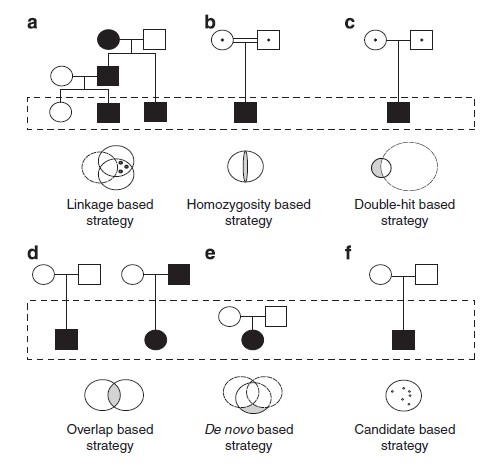
doi:10.1038/ejhg.2011.258;
5.一代sanger测序确认结果
伴随着WES临床应用的深入,对于WES结果的规范解读也有了更多要求规范以保证最终检测结论的准确可信。ACMG率先于2015年发布了针对测序位点解读注释指南(Richards, Aziz et al. 2015),该指南对变异分类、支持分类的证据以及适用范围均给出了比较明确的界定,意义重大。随后,ACMG于2016年又再次更新了次要发现(secondary finding)的处理规范(Kalia, Adelman et al. 2017),在此次规范中重新更新了59个基因,建议临床实验室一并报告检测中涉及的该基因列表中的检测情况,进一步补充和完善了相关测序结果的报告范围。
【2】VALENCIA, C. Alexander et al. CCEPAS: the creation and validation of a fast and sensitive clinical whole exome analysis pipeline based on gene and variant ranking. Journal of Translational Genetics and Genomics, [S.l.], v. 2, n. 1, p. 1, jan. 2018. ISSN 2578-5281
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.全部课程可点击:组学大讲堂视频课程
- 发表于 2018-09-25 15:31
- 阅读 ( 5515 )
- 分类:文献解读
你可能感兴趣的文章
- 外显子测序解析卵巢早衰的遗传因素 2923 浏览
