Cytoscape如何基于点的属性快速对点和相连的线进行颜色分类
在利用Cytoscape绘图的过程中如何对具有不同属性(分类)的点(以及与该点相连的线)进行颜色着色呢?
譬如下图 显示了miRNA 和靶基因的相互作用,并基于靶基因的类型对网网络图进行了颜色处理,方便显示各个分类:

绘图的原始数据基本格式如下,仅两列:
| miRNA | Target |
| ath-miR156a-3p | AT1G65440 |
| ath-miR156a-3p | AT3G06340 |
| ath-miR156a-3p | AT5G58220 |
| ath-miR156a-5p | AT1G08590 |
| ath-miR156a-5p | AT1G19800 |
| ath-miR156a-5p | AT1G72700 |
| ath-miR156a-5p | AT1G80680 |
| ath-miR156a-5p | AT2G02090 |
| ath-miR156a-5p | AT2G40316 |
| ath-miR156a-5p | AT3G58190 |
| ath-miR156a-5p | AT4G33540 |
| ath-miR156a-5p | AT5G01810 |
| ath-miR156a-5p | AT5G19610 |
| ath-miR156b-3p | AT2G38440 |
| ... | ... |
如涉及相关的属性信息,可以在数据中增加相应的列,来描述点或者线的特征。
基于最原始的数据可以获得如下图片(三角对应miRNA,圆点对应靶基因):

1、根据其他的分析,例如基于注释,将靶基因所属的pathway列举出来:
| Target | pathway |
| AT1G65440 | A |
| AT3G06340 | A |
| AT5G58220 | A |
| AT1G08590 | A |
| AT1G19800 | A |
| AT1G72700 | A |
| AT1G80680 | A |
| AT2G02090 | A |
| AT2G40316 | A |
| AT3G58190 | |
| AT4G33540 | |
| AT5G01810 | B |
| AT5G19610 | B |
| AT2G38440 | B |
| AT1G08590 | B |
| AT1G19800 | B |
| AT1G72700 | B |
| AT1G80680 | B |
| AT2G02090 | B |
| AT2G40316 | B |
| AT3G58190 | B |
| AT4G33540 | B |
| AT5G01810 | B |
| AT5G19610 | |
| AT1G73370 | |
| AT3G06340 | |
| AT3G12860 | |
| AT1G08590 | C |
| AT1G19800 | C |
| AT1G72700 | C |
| AT1G80680 | C |
| AT2G02090 | C |
| AT2G40316 | C |
| ... | ... |
获取的信息中,有一些基因能注释到特定pathway,有一些不能(可将对应的内容空置不填),基于这样的表格数据,将其作为table,导入到Cytoscape中,作为点的属性,进行绘图。譬如经过table导入后,在对应的数据栏中,显示增加了一列pathway(注意作为table导入的数据应该是去重的数据,也就是说每个靶基因只有一个分类属性):
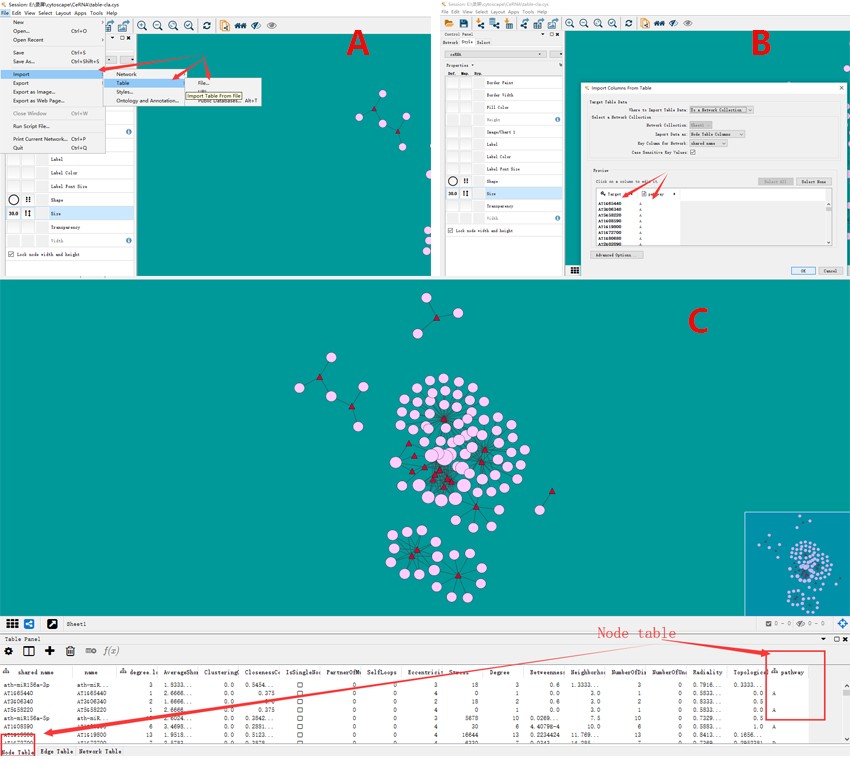
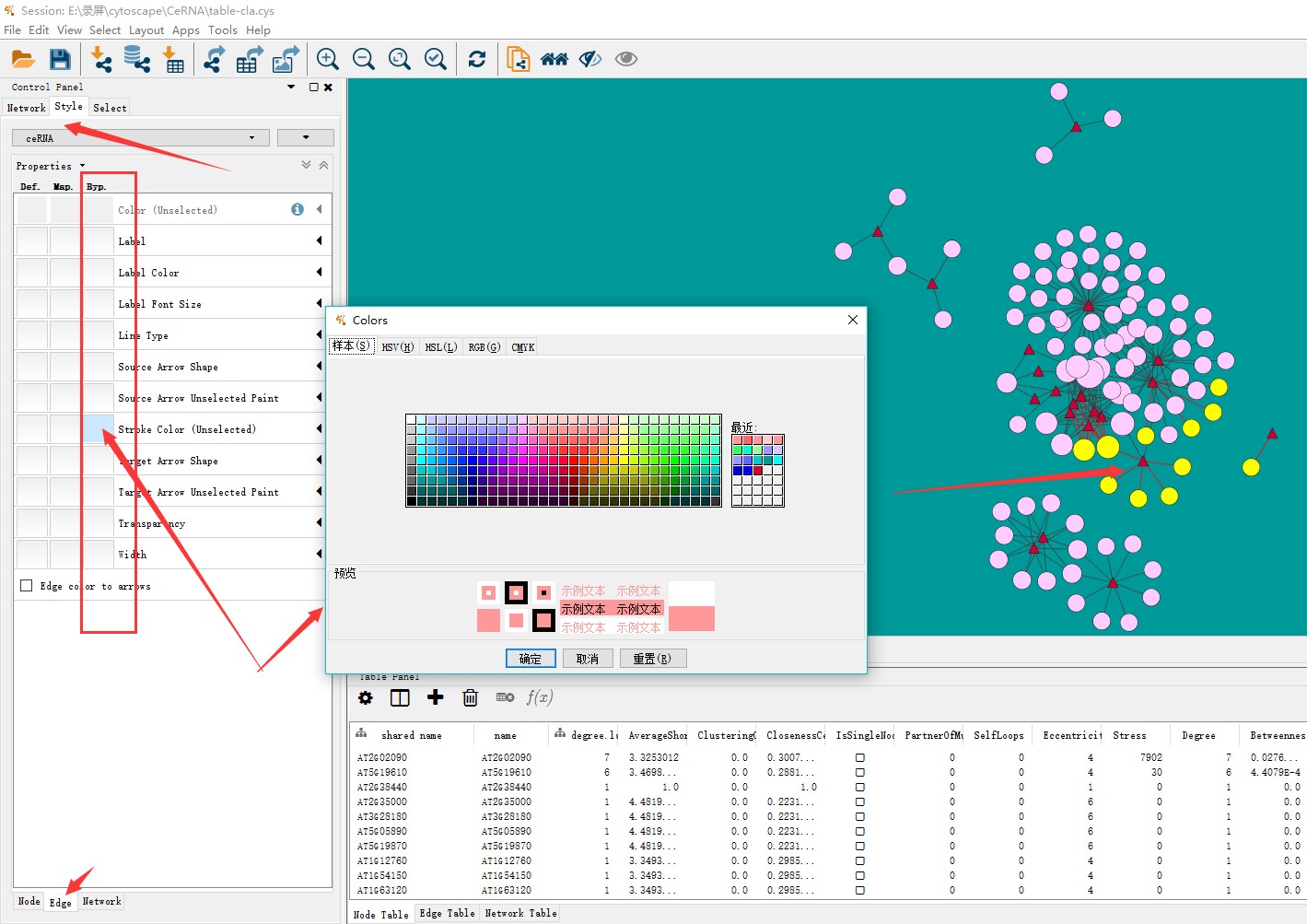
2、在控制面板中 对应的select中 设定筛选栏,以Node:pathway来(表示Node属性中pathway这一列的数据) 选择其中一个pathway B,即可将属于这一类的点选中(图中黄色点)
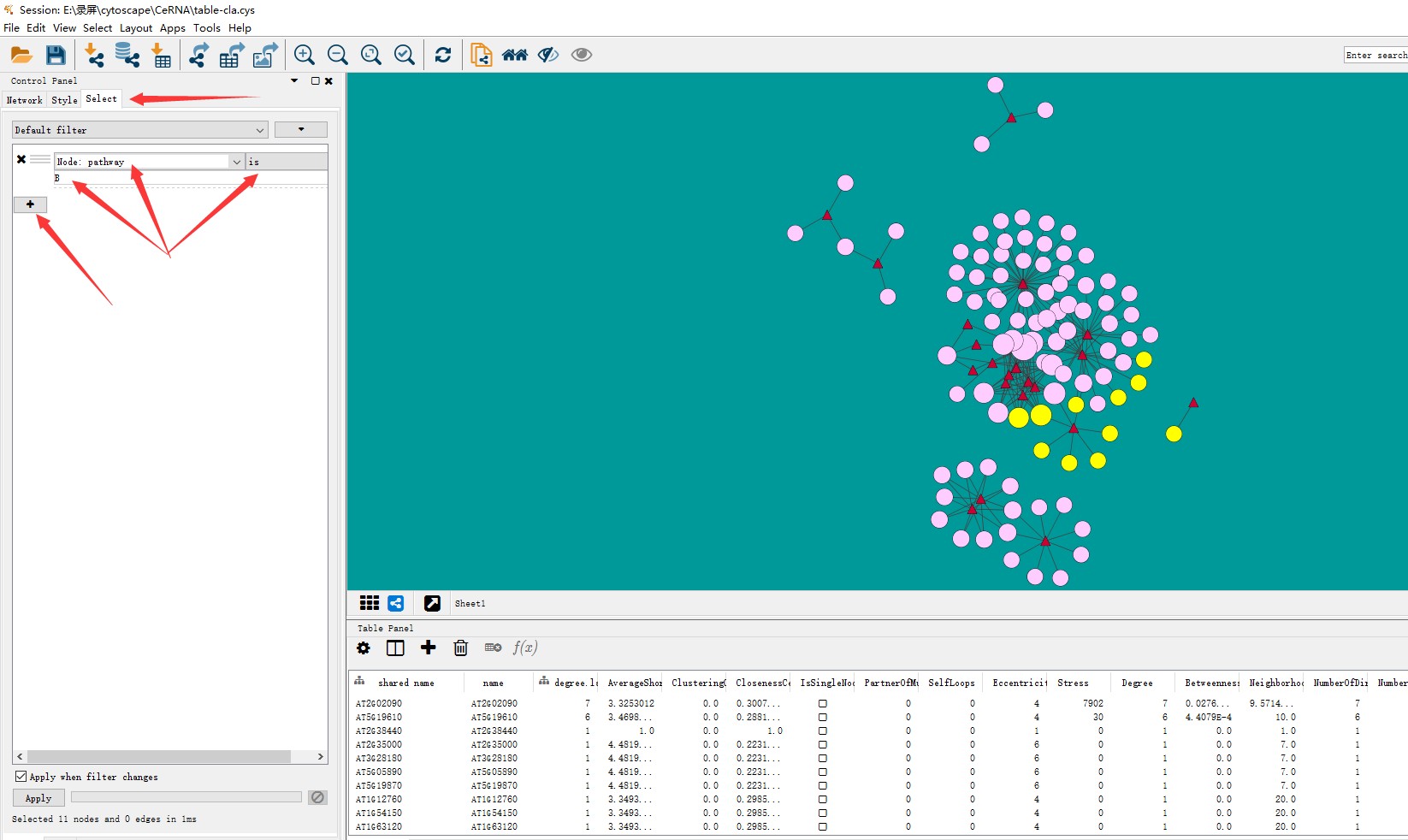 选中点之后,进入控制面板style-node 中在第三列修改点的颜色(第三列仅修改选中的点的设置)
选中点之后,进入控制面板style-node 中在第三列修改点的颜色(第三列仅修改选中的点的设置)
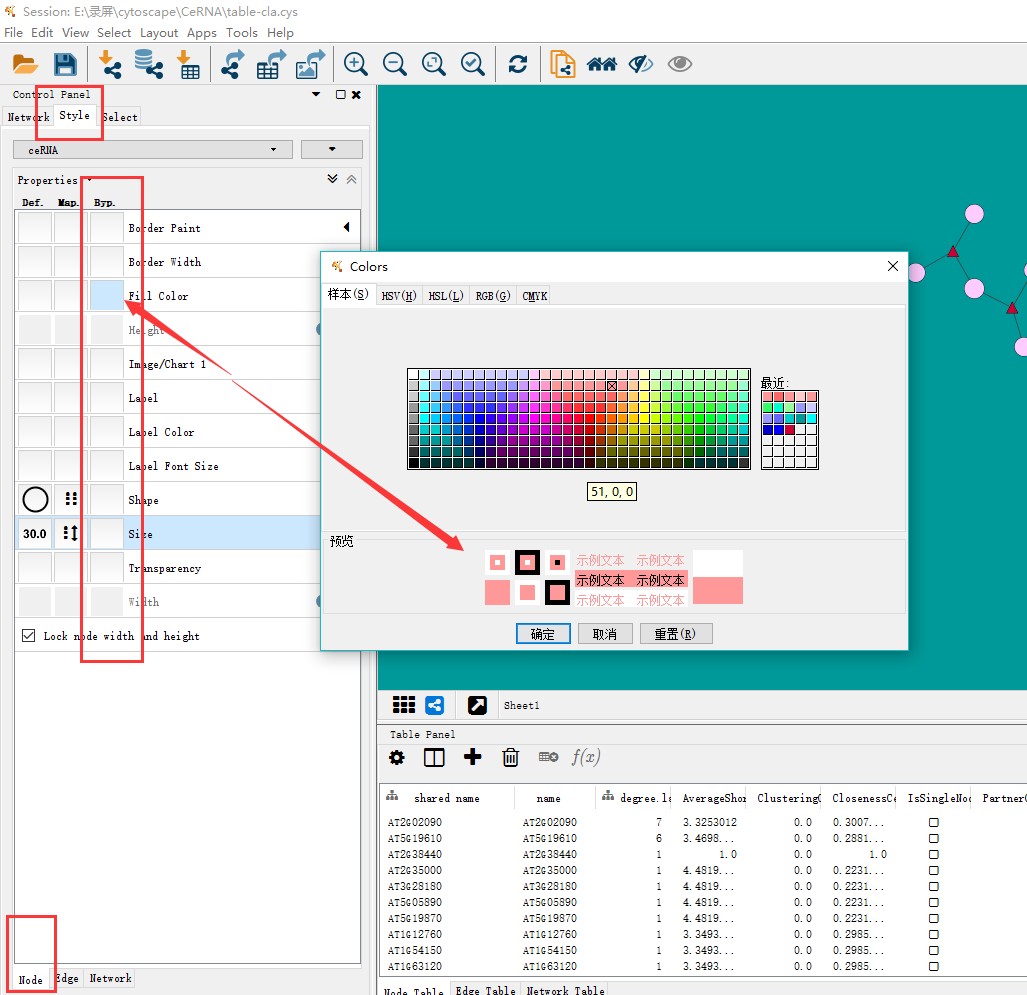 即可将点修改成指定颜色,而与该类点相连的线,可以在选中点之后,基于工具栏select 选中与点相连的线,再进行修改线的颜色。
即可将点修改成指定颜色,而与该类点相连的线,可以在选中点之后,基于工具栏select 选中与点相连的线,再进行修改线的颜色。
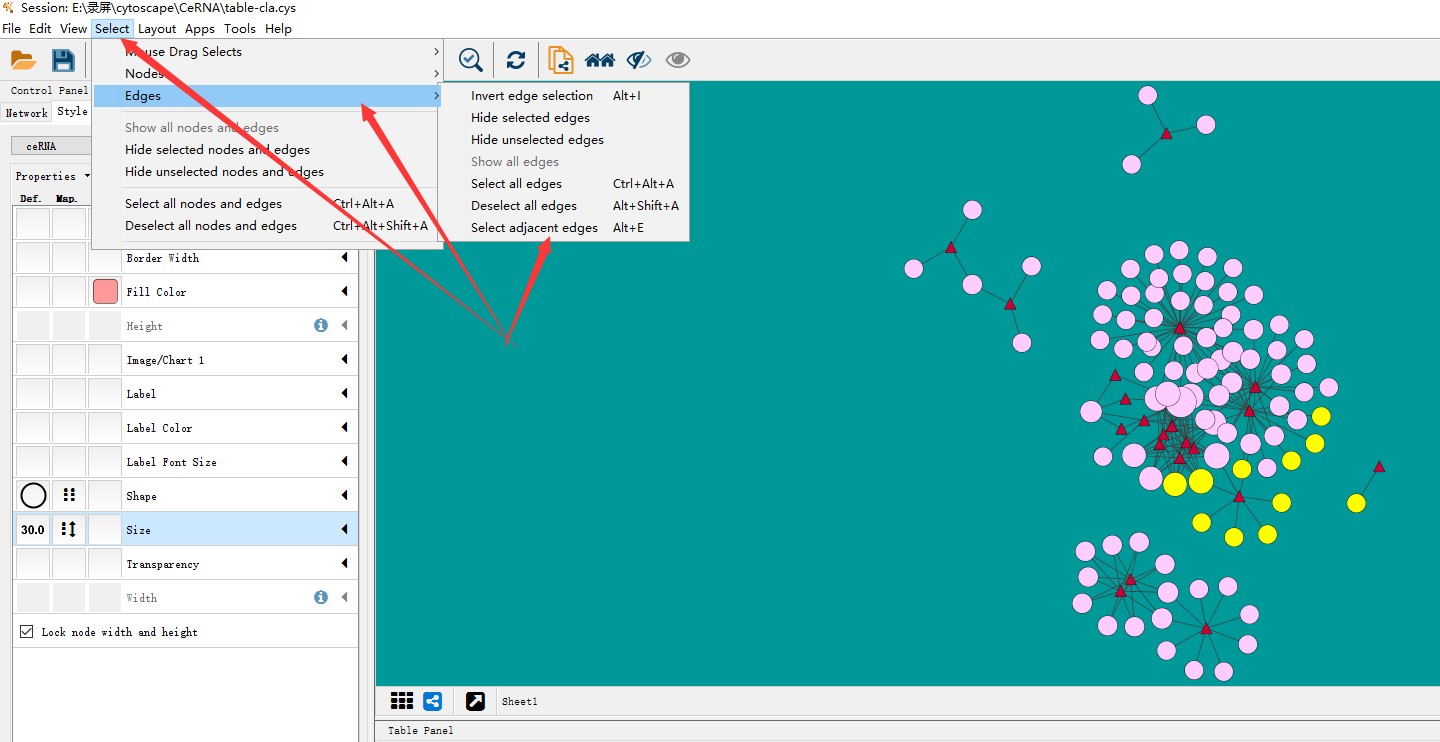 线的颜色,修改类似于点,选中之后线显示红色,在控制面板Edge中修改,注意选中的线也是第三列,修改颜色为Stroke color
线的颜色,修改类似于点,选中之后线显示红色,在控制面板Edge中修改,注意选中的线也是第三列,修改颜色为Stroke color
经过多次修改不同类型之后即可完成:

除了基于table之外,也可以对最初导入的Network文件进行信息补充,方便后续进行网络网络设置,例如:
| miRNA | Target | pathway | type |
| ath-miR156a-3p | AT1G65440 | A | A |
| ath-miR156a-3p | AT3G06340 | A | A |
| ath-miR156a-3p | AT5G58220 | A | A |
| ath-miR156a-5p | AT1G08590 | A | A |
| ath-miR156a-5p | AT1G19800 | A | A |
| ath-miR156a-5p | AT1G72700 | A | A |
| ath-miR156a-5p | AT1G80680 | A | A |
| ath-miR156a-5p | AT2G02090 | A | A |
| ath-miR156a-5p | AT2G40316 | A | A |
| ath-miR156a-5p | AT3G58190 | A | A |
| ath-miR156a-5p | AT4G33540 | A | A |
| ath-miR156a-5p | AT5G01810 | A | A |
| ath-miR156a-5p | AT5G19610 | A | A |
| ath-miR156b-3p | AT2G38440 | A | A |
| ath-miR156b-5p | AT1G08590 | A | A |
| ath-miR156b-5p | AT1G19800 | A | A |
| ath-miR156b-5p | AT1G72700 | A | A |
| ath-miR156b-5p | AT1G80680 | ||
| ath-miR156b-5p | AT2G02090 | ||
| ath-miR156b-5p | AT2G40316 | ||
| ath-miR156b-5p | AT3G58190 | ||
| ath-miR156b-5p | AT4G33540 | B | B |
| ath-miR156b-5p | AT5G01810 | B | B |
| ath-miR156b-5p | AT5G19610 | B | B |
| ath-miR156c-3p | AT1G73370 | B | B |
| ath-miR156c-3p | AT3G06340 | B | B |
| ath-miR156c-3p | AT3G12860 | B | B |
| ath-miR156c-5p | AT1G08590 | B | B |
| ath-miR156c-5p | AT1G19800 | B | B |
| ath-miR156c-5p | AT1G72700 | B | B |
| ...... | ... | ... |
在导入网络时,注意将pathway 设置成靶点属性,将type设置成线属性,之后再于控制面板中中设置筛选栏,基于Node:pathway 或者Edge:type去选中点或者线,从而去修改相关的设置也可以达到相同效果。
如果想提升自己的绘图技能,我们推荐:Cytoscape与网络图绘制课程、R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-10-12 11:28
- 阅读 ( 17162 )
- 分类:科研作图
你可能感兴趣的文章
- cytoscape软件的安装与使用_v3.10.2 4888 浏览
- cytoscape-cytoHubba插件的安装与使用 22003 浏览
- cytoscape软件安装与使用-绘制蛋白互作网络图 7805 浏览
- 网络概念以及Cytoscape导入数据格式理解(ceRNA等多元关系网络数据) 12167 浏览
- cytoscape插件BinGO安装以及GO富集分析和网络可视化 292166 浏览
