phytozome植物基因组下载网站
Phytozome 作为专门收录植物基因组的网站,在基因组数据的下载、查询、可视化浏览等方面做的也很不错,也是一个不错的基因组数据下载数据库。
今天主要介绍一下该网站基因组如何下载,如何通过blast查询同源基因,以及根据基因相关功能结构域批量下载相关基因的CDS,PEP等序列信息;
基因组下载
该网站需要注册登陆之后才能下载基因组数据:注册也非常简单,这里就不详细说明了:注册完账号登陆就可以:https://phytozome.jgi.doe.gov
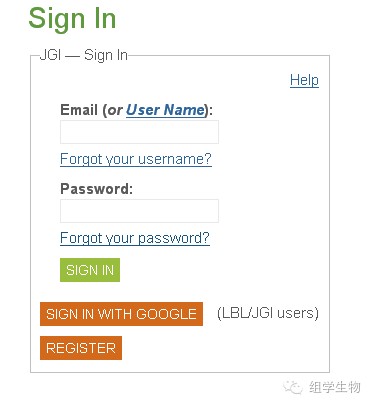
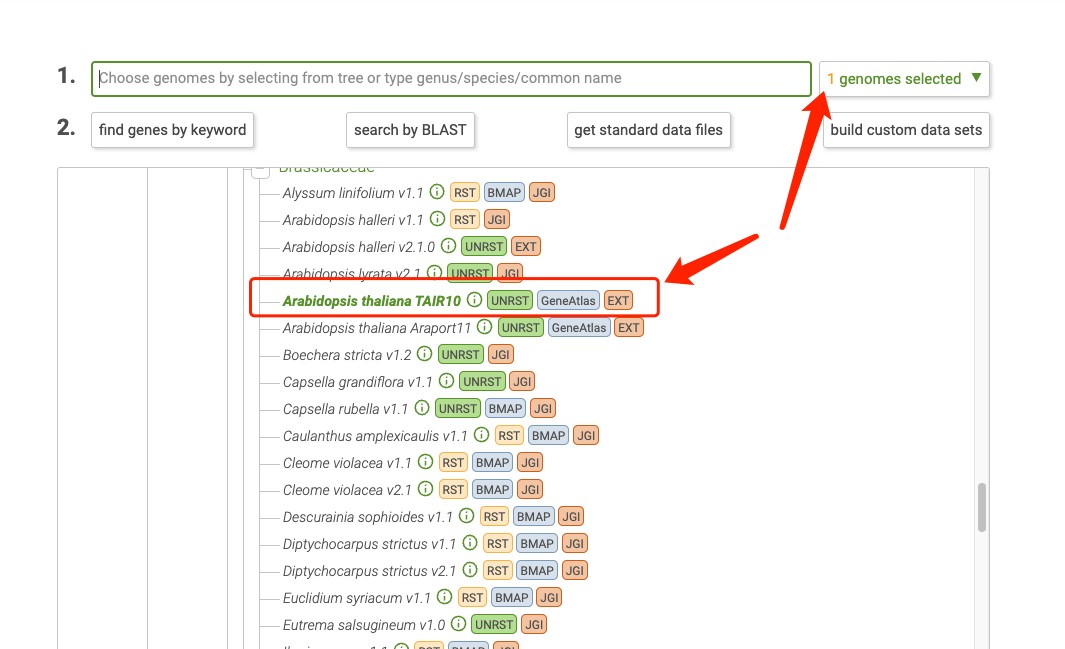
登陆之后就可以去下载参考基因组,输入自己物种的拉丁名搜索,之后点击添加到genome selected:
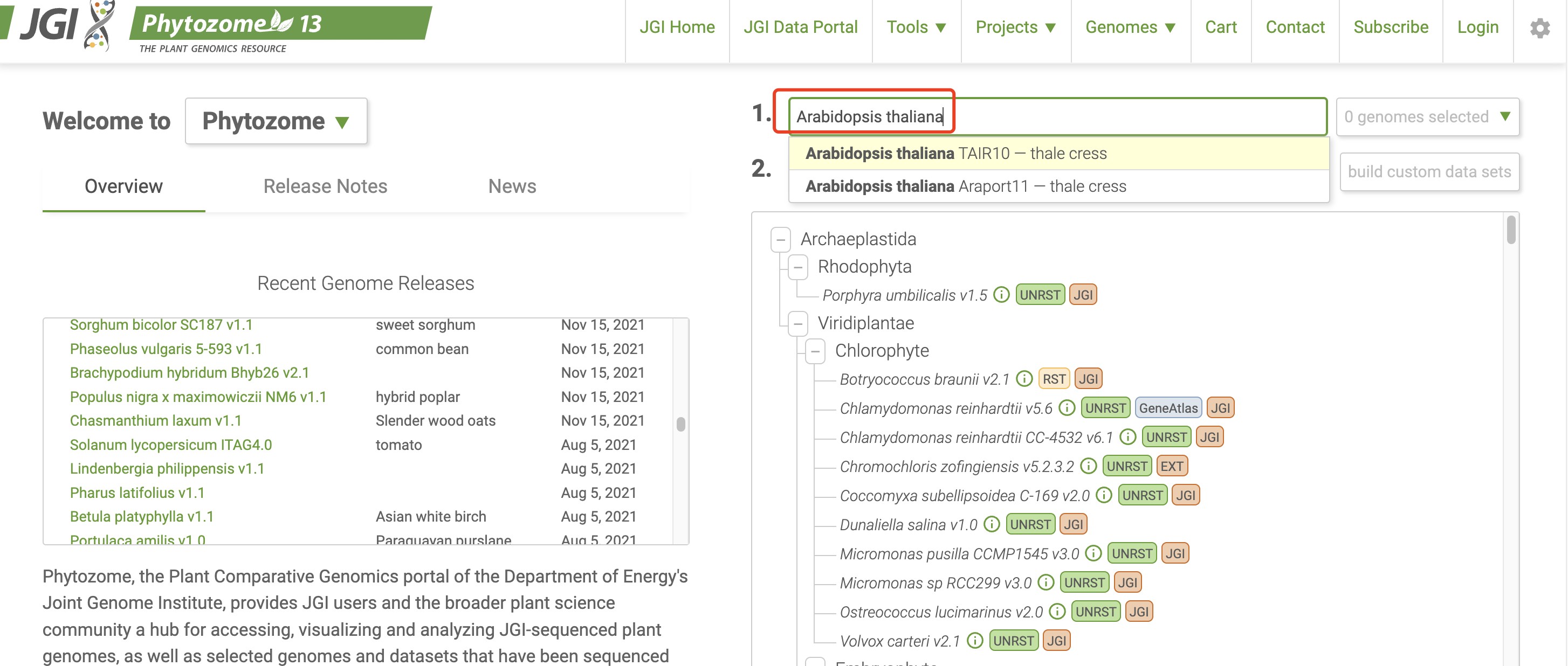
点击物种名称,进入物种介绍页面:
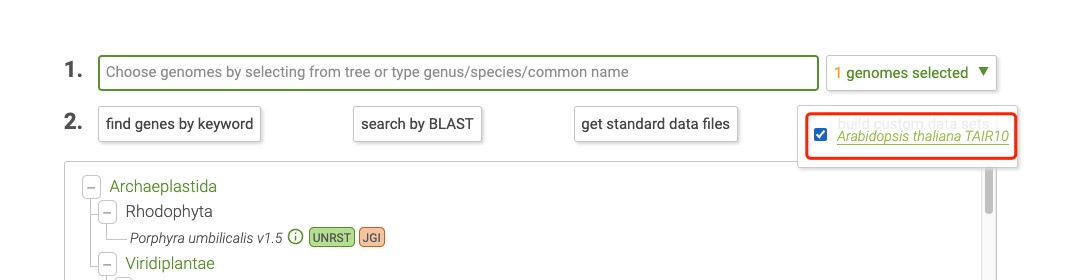
基因组介绍页面,可点击download页面下载: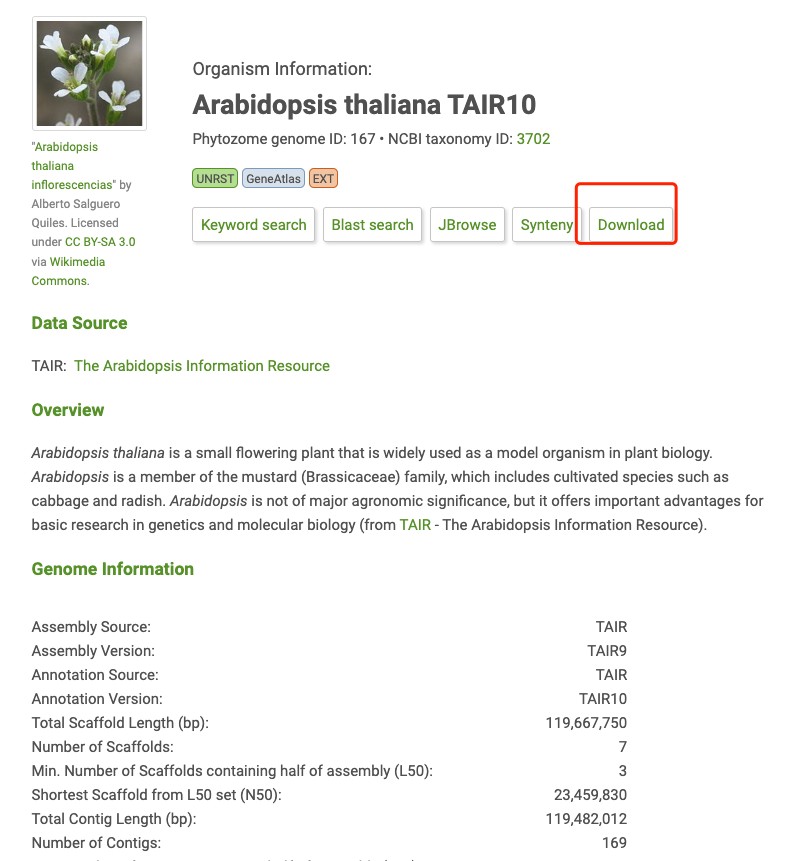
勾选自己想要的数据,最后到购物车中下载即可:
查找相似功能或结构域的基因序列,并批量下载:
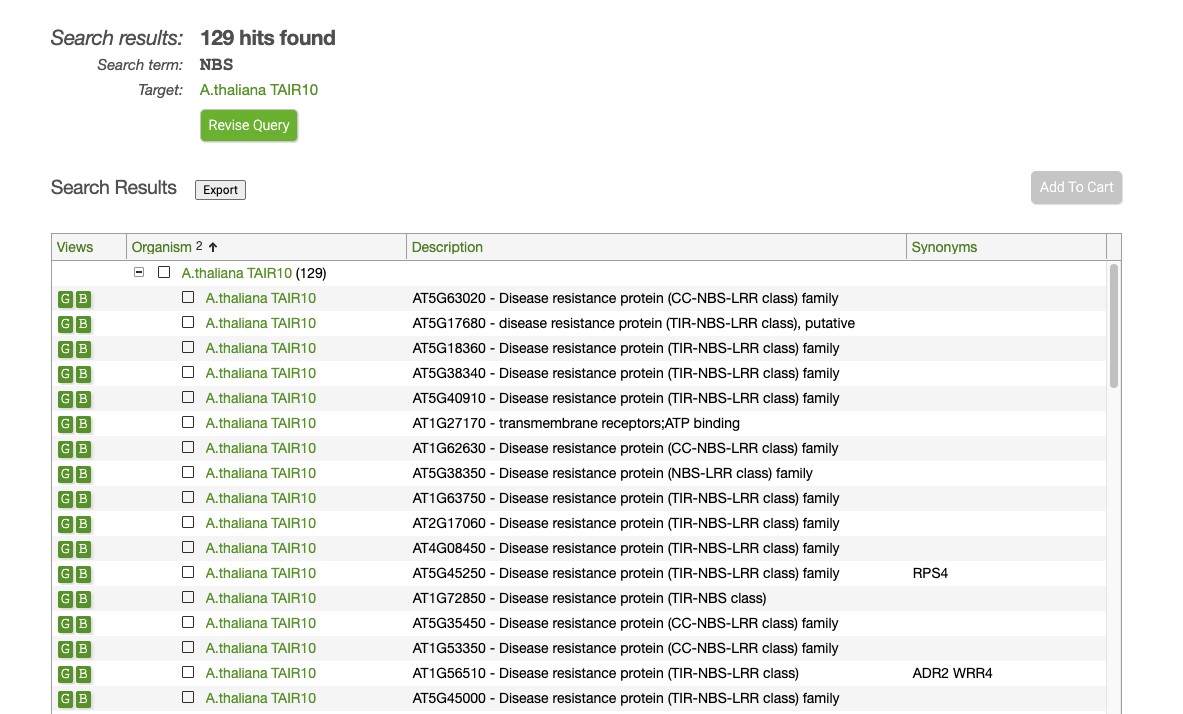
以拟南芥基因组为例,查找Nucleotide binding site,NBS结构域相关的基因,并批量下载其CDS等序列,以便用于做后续分析;如定量,设计引物,同源比对等等;
首先,回到首页,1.点击想要搜索的物种, 2 选择find genes by keywords
得到很多关于NBS相关的基因,共有128个基因如下:
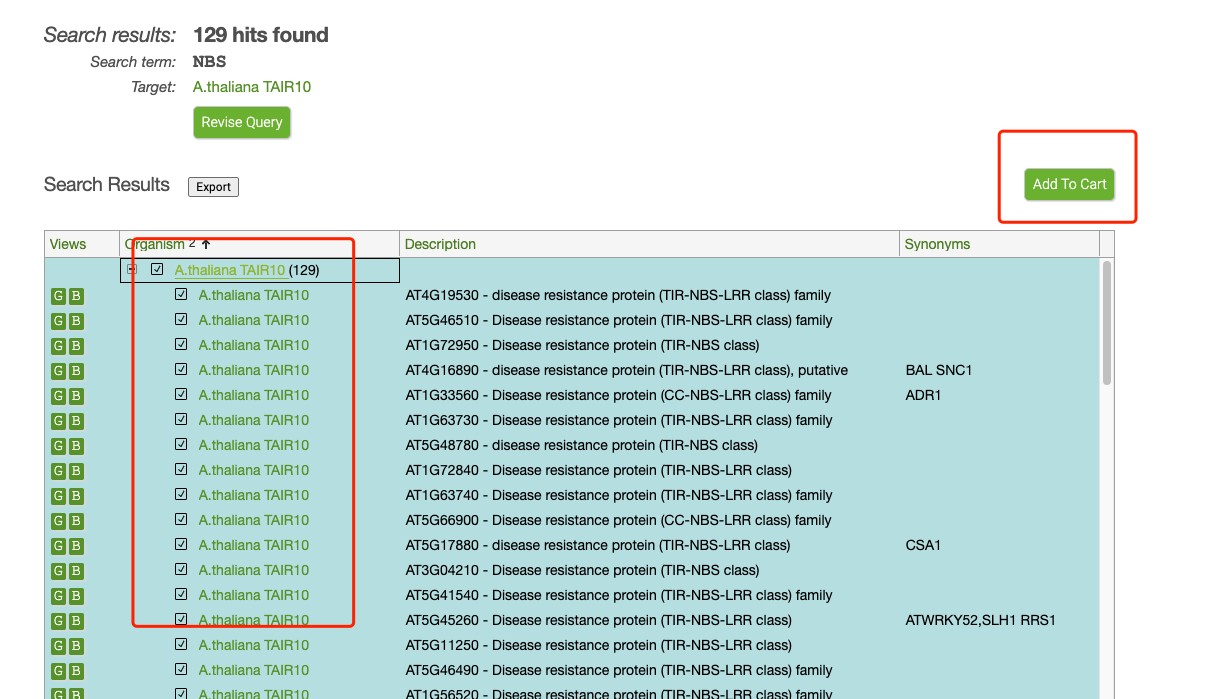
批量下载这些基因的序列:先勾选所有基因,注意有翻页,记得一起勾选上,再add一下:
然后就可以按下面的步骤下载想要的序列了,非常的方便:
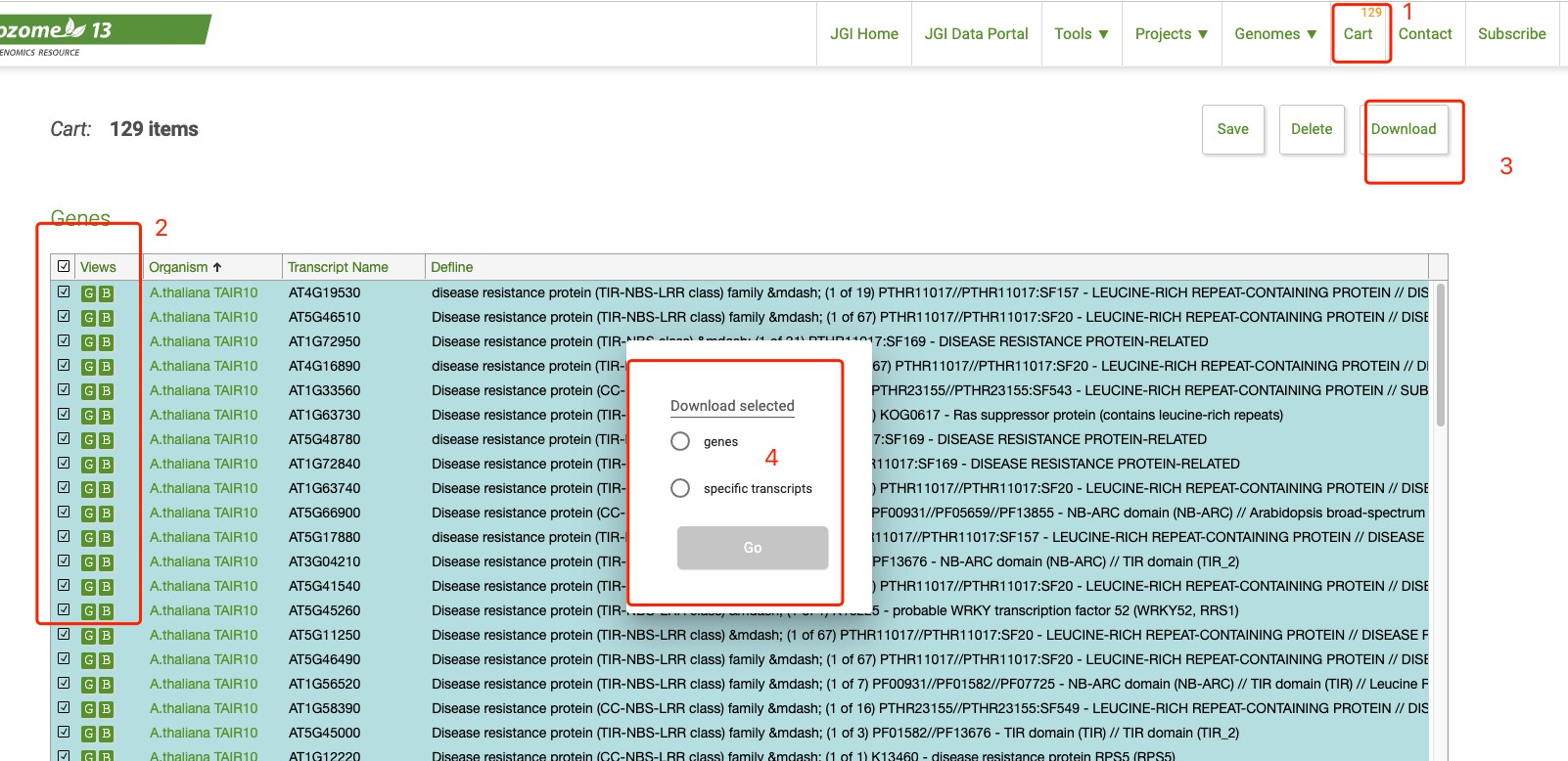
当然了,如果想看其中一个感兴趣的基因具体的信息也非常的方便:只要点击GB 可视化方法就可以:
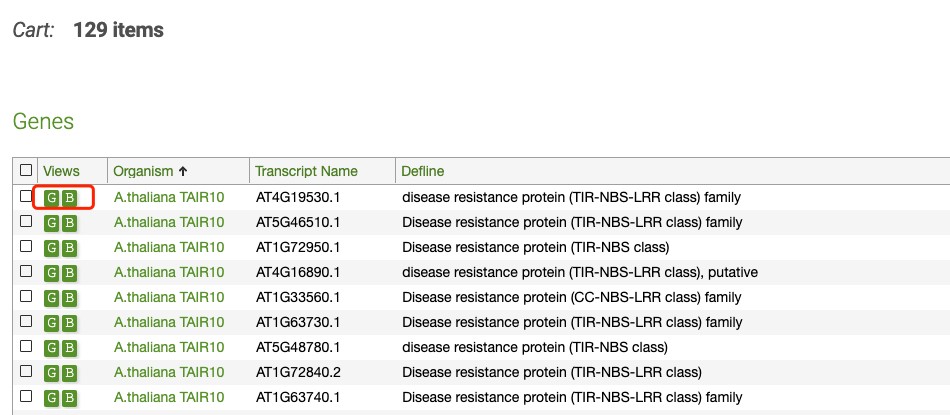
点击B显示的效果:
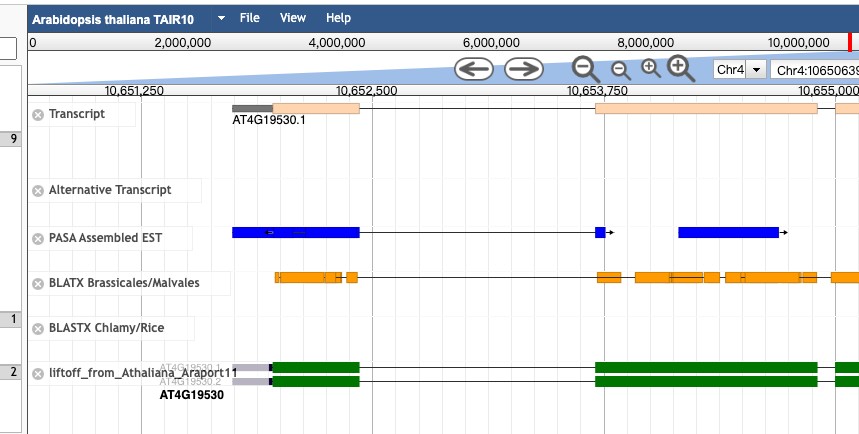
点击G显示的效果,具体序列直接可以得到:
关于该基因的序列,功能,基因外显子内含子,同源蛋白,表达量等信息应有尽有:
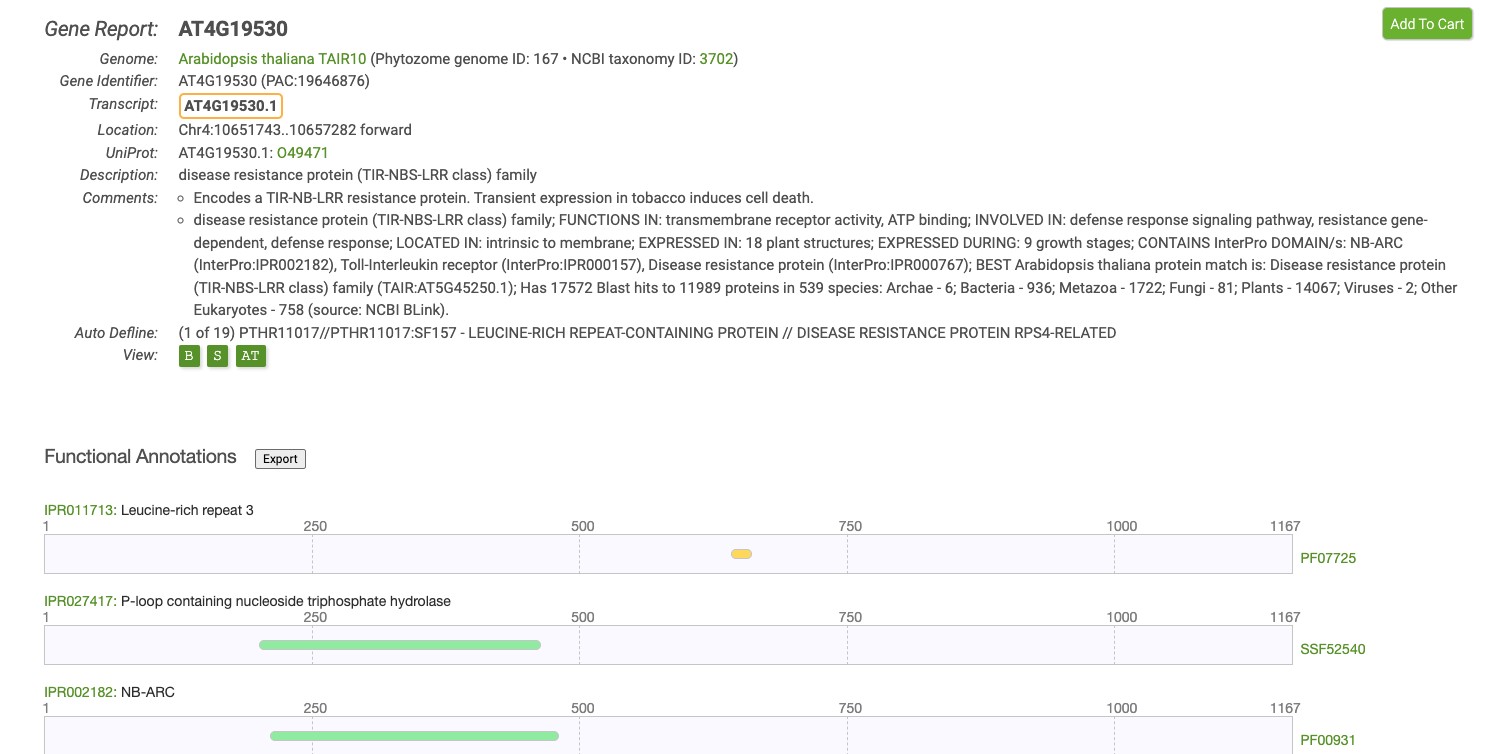
blast 比对查找同源序列
进入首页 ,多勾选几个感兴趣物种,
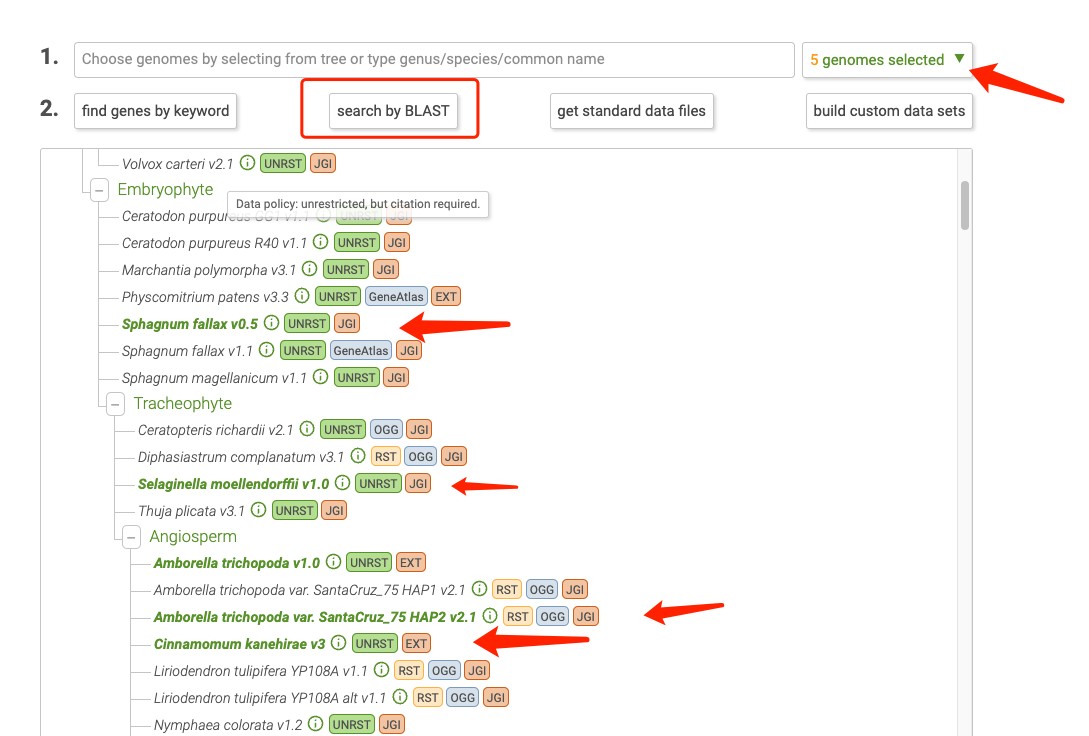
然后点击 search blast, 输入自己的序列:
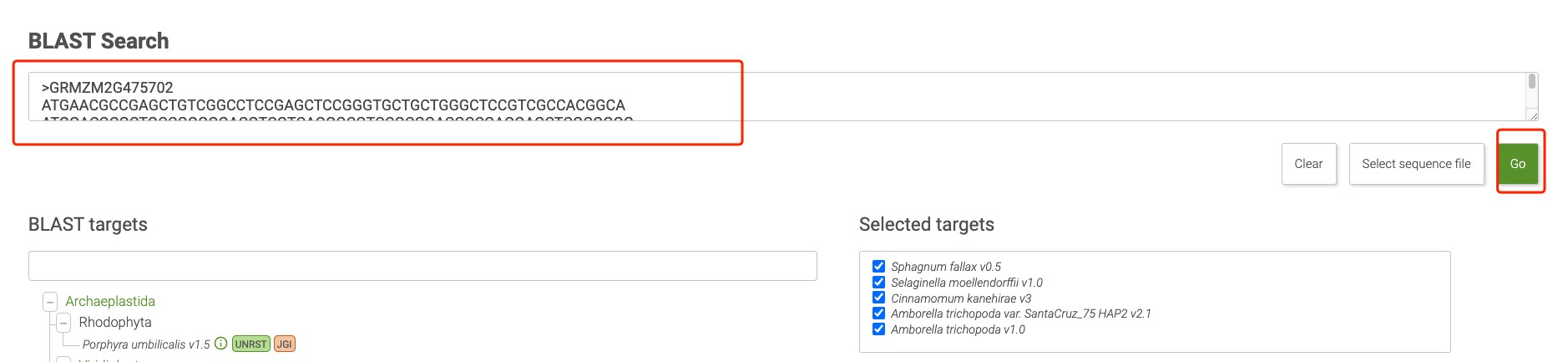
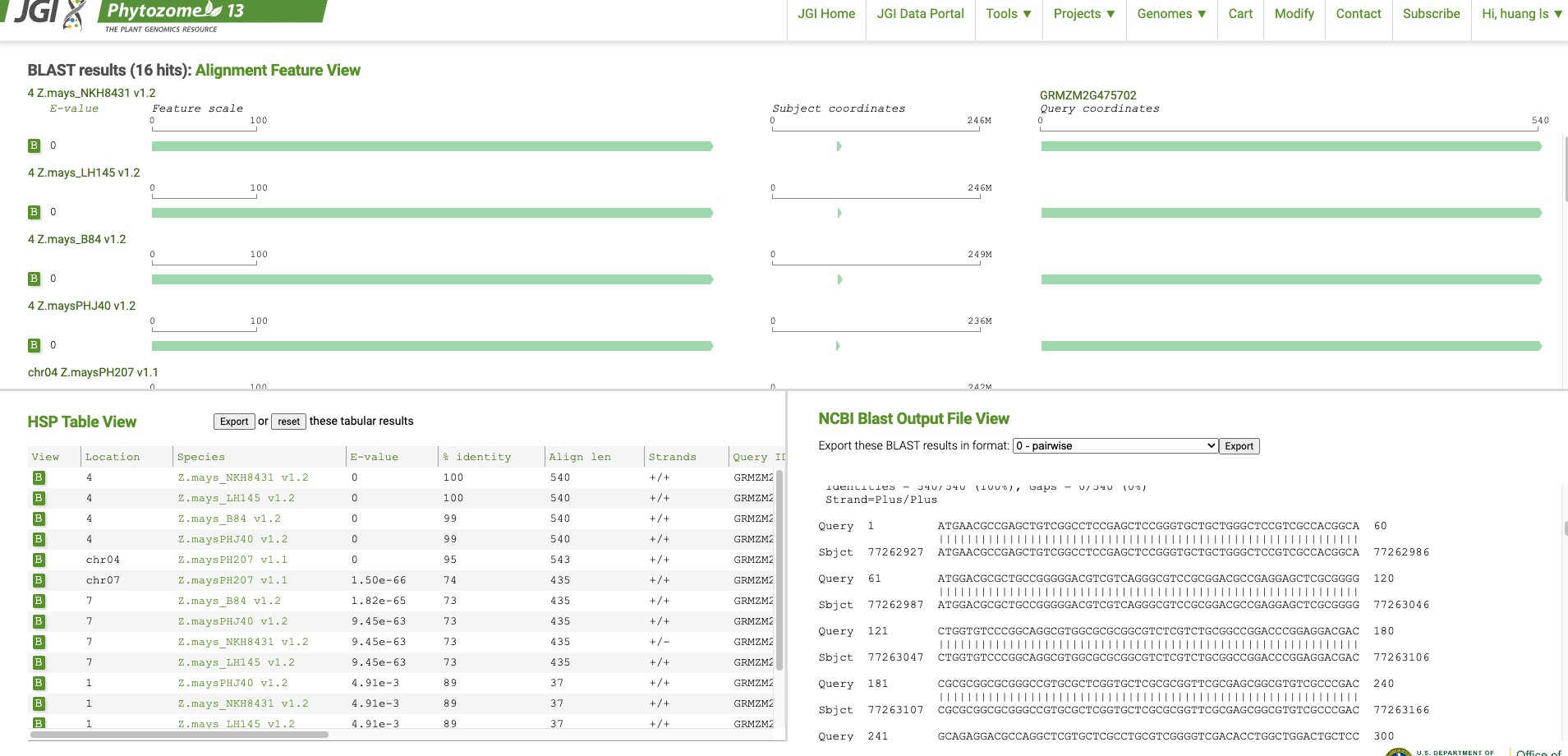
搜索结果如下,也可以批量导出,便于后续分析,是不是功能很强大?
更多生物信息课程:https://study.omicsclass.com/index
- 发表于 2018-04-22 14:21
- 阅读 ( 44424 )
- 分类:软件工具
