从NCBI当中SRA数据库中下载高通量测序数据
用别人的数据,发自己的文章
由于大多数杂志在文章发表前要求公开数据,所以随着测序文章的爆发,NCBI的SRA数据库当中积累了海量的测序数据。我们可以利用这些数据重新做数据挖掘,发表新的文章。
官方下载方法不太稳
要利用数据,首先得下载得到数据,虽然SRA数据库提供的SRA Toolkit 工具包里的prefetch可以下载,但是用这个方法下载数据需要经过复杂的设置,而且经常莫名奇妙的下不了,总的来说体验很差。
其实老朋友迅雷也是可以下载的
下面通过一个例子介绍如何用迅雷下载SRA数据:
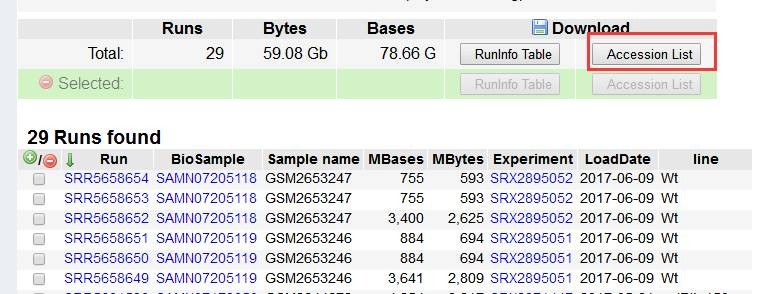
例如,我们要下载SRP108428(阅读文献可以找到公开数据的project号)下的所有数据,打开NCBI网址:https://www.ncbi.nlm.nih.gov/Traces/study/?acc=SRP108428(此处为project号),点击"Accession List"键,下载得到SRR List 储存在sra.txt文件中。
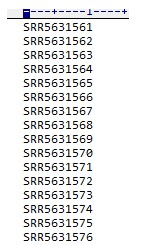
打开sra.txt文件:
那么我们就可过下载地址规律生成所有样品的ftp的下载地址:
ftp://ftp-trace.ncbi.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR563/SRR5631562/SRR5631562.sra
注:头部都一样(黑色字),后面地址分别为SRR,SRR+前三个数、SRR号、SSR号.sra
得到如下结果:

最后,将链接粘贴到迅雷下载即可, 是不是很方便?
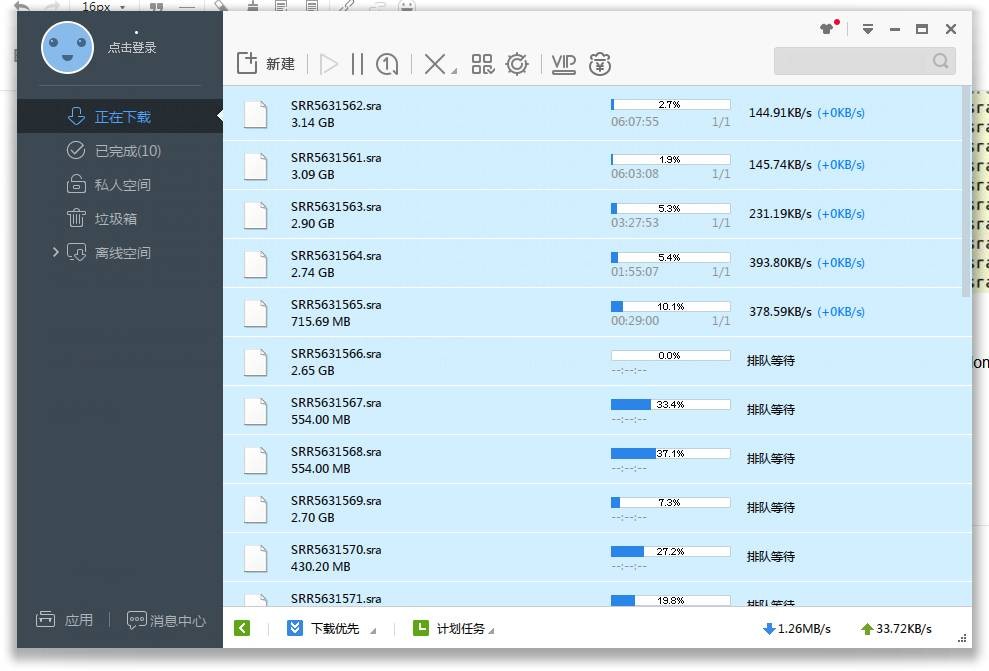
Tips:
如果下载上百个样品,手动粘贴就太累了。在Linux下可以实现一键自动产生下载链接,输入文件为sra.txt, 给出命令:
cat SRR_Acc_List.txt|while read a ;do t1=${a:0:3};t2=${a:0:6};echo "ftp://ftp-trace.ncbi.nih.gov/sra/sra-instant/reads/ByRun/sra/$t1/$t2/$a/$a.sra";done
更多生物信息课程:https://study.omicsclass.com/index
- 发表于 2018-04-22 14:37
- 阅读 ( 21138 )
- 分类:软件工具
你可能感兴趣的文章
- 探索基因大数据的利器——SRA Explorer 2563 浏览
- NCBI 基因组文件下载——datasets 2495 浏览
- 使用NCBI——Batch Entrez批量下载蛋白序列 12151 浏览
- NCBI的SRA Lite和SRA Normalized数据有什么区别 7792 浏览
- NCBI下载SRR并转换为fastq文件 12786 浏览
- NCBI下载物种叶绿体或线粒体基因组fasta,gff3,gtf,gb文件等 10788 浏览
相关问题
- SRA下载 1 回答
- SRA数据下载 2 回答
- R语言下载GSE数据一直在报错 0 回答
- 老师,下载数据是出现图片这个错误,我该怎么办? 1 回答
- 求助:用Rstudio下载数据,出现remaining后不动了,是不能下载了吗? 2 回答
- 请问老师怎么将NCBI里的gbff文件转化为gff格式的文件,因为里面找不到gff文件 0 回答