再挖TCGA,发篇SCI
今天再一次给大家介绍一篇不用做实验,只需要进行TCGA数据库挖掘,就能发的SCI的金思路。接下来以一篇2018年7月30日发表在Cellular Physiology and Biochemistry(IF=5.5)上题为Genome-Wide Analysis of Prognostic lncRNAs, miRNAs, and mRNAs Forming a Competing Endogenous RNA Network in Hepatocellular Carcinoma 的文章为例,详细介绍下该类文章的写作思路!
该文章思路清晰明了,分别针对癌症和癌旁的lncRNA,miRNA,mRNA进行差异分析,生存分析,并构建ceRNA调控网络,再对网络中的分子进行功能分析;整个分析过程一气呵成,值得学习和参考。
1. 数据来源
从TCGA数据库中下载肝癌的374份癌症组织和50份癌旁组织的基因和lncRNA表达数据。从UCSC Xena 下载相应肝癌的miRNA数据。
2. 差异表达分析
基于374份肝癌样本和50份癌旁样本的基因,lncRNA,和miRNA表达数据,进行差异表达分析,筛选差异表达的分子。
差异表达的火山图如下:
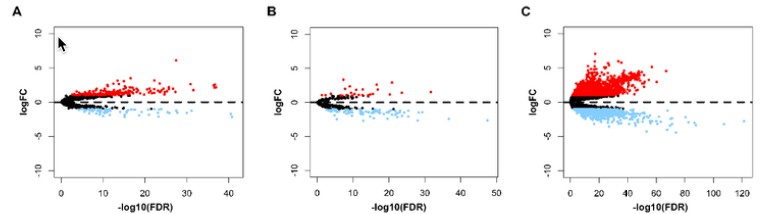
差异表达聚类热图如下:

3. 生存分析
3.1 单因素生存分析
基于差异表达的结果,分别针对差异表达的分子与临床数据中的总生存时间进行单因素的COX生存分析,最终得到77个lncRNA, 29个 miRNA, 1014个mRNA与生存显著相关。
生存相关lncRNA, miRNA,mRNA的风险比例深林图如下:
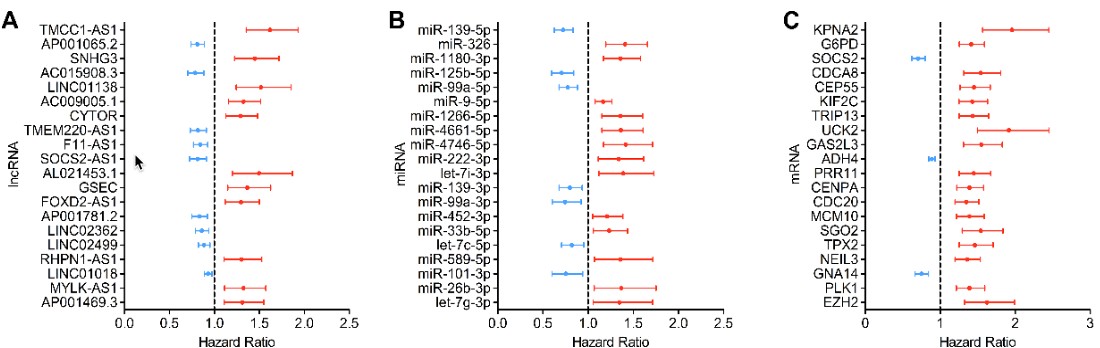
3.2 多因素生存分析
基于单因素的分析结果,进一步采用多因素的COX回归模型进行分析,最终分别筛选了3个分子构建了预后指数。
基于预后指数,参考中位数,可以将病人分成两组,分别是高风险组和低风险组。
针对两个分组,进一步采用K-M生存分析,得到预后指数的分类效果。从P值的大小来看,分类效果很好。
基于预后指数进行KM分析,获得的生存曲线图如下:
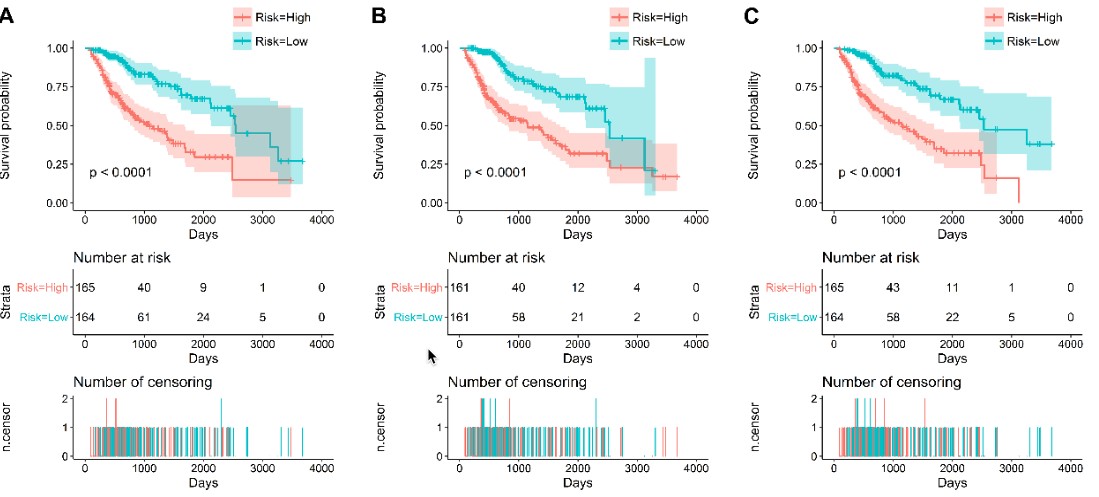
基于预后指数进行预测效果检验,绘制检测效果的ROC曲线如下:
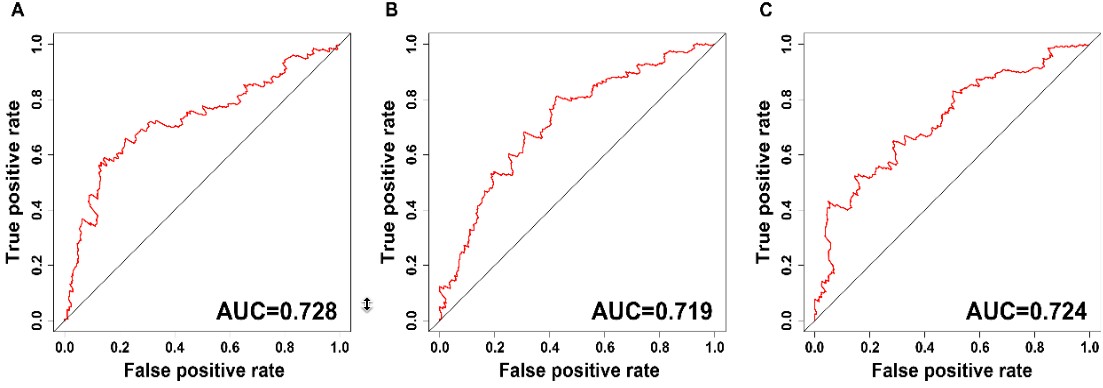
从ROC曲线的AUC值来看,分类效果还不错,都在0.7 以上。
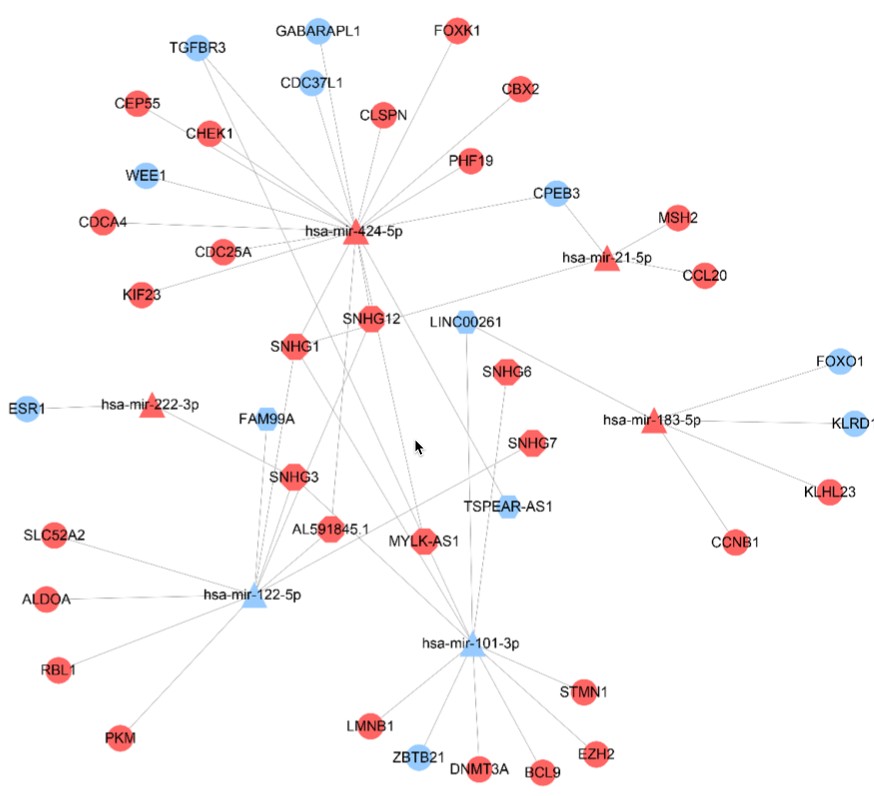
4. ceRNA网络构建
基于生存分析筛选出来的显著相关lncRNA, miRNA, mRNA 构建ceRNA网络。网络包括两部分,分别是:
1. lncRNA-miRNA 相互作用: 采用miRcode 网站进行预测;
2. miRNA-mRNA的相互作用: 基于TargetScan,miRTarBase,miRDB 等软件和数据库进行预测。
构建的ceRNA网络如下:
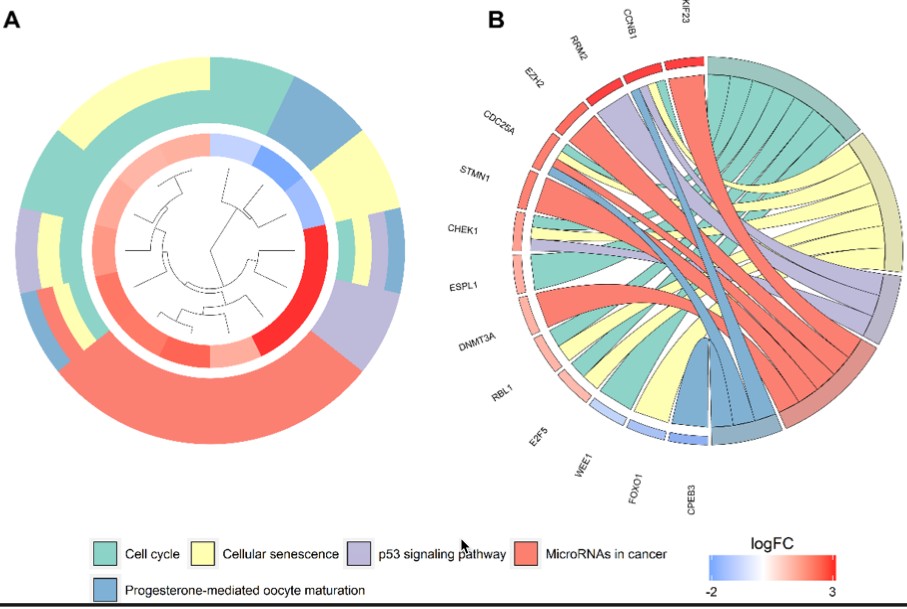
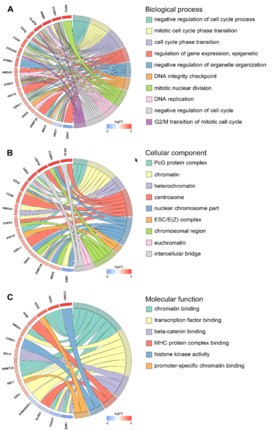
5. 基因功能分析
针对ceRNA网络中的mRNA进行GO,KEGG的功能注释和分析。从中可以发现一些基因跟细胞周期,P53 信号途径,癌症相关miRNA等通路相关。
KEGG 代谢通路注释如下:
GO功能注释如下:
6. 文章亮点
文章的思路简单明了:从差异分析到生存分析,ceRNA以及功能分析,一步步紧扣,一气呵成。
分析对象系统而全面:从lncRNA, miRNA,mRNA等多个层面去分析,并构建相互作用的调控网络,使得分析结果更加具有说服力。
图表简洁美观: 文章中的图表非常多,而且简洁美观。
参考文献:Genome-Wide Analysis of Prognostic lncRNAs, miRNAs, and mRNAs Forming a Competing Endogenous RNA Network in Hepatocellular Carcinoma. Cell Physiol Biochem,2018; 48: 1953-1967.
- 发表于 2018-11-09 17:40
- 阅读 ( 5577 )
- 分类:TCGA
你可能感兴趣的文章
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- TCGA下载 docker报错 0 回答
- TCGA差异分析如何根据临床信息分组? 1 回答
- TCGA用R数据下载错误 1 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- TCGA的RNA-seq count annotation过滤,有些不知道是否需要删除 0 回答
