ValueError: invalid literal for int() with base 10: ''
python 版本mcscan 分析报错
python 版本的mcscan分析最后绘制共线性绘图时报错如下:
运行命令如下:
/biosoft/miniconda/miniconda2/bin/python -m jcvi.graphics.karyotype --format=pdf --figsize=15x5 mcscan_seqid mcscan_layout
报错信息如下:
markers', 'networkx.algorithms.community.__future__', 'networkx.generators.atlas', 'networkx.algorithms.connectivity.edge_kcomponents', 'pkg_resources._vendor.types', 'scipy.sparse._matrix_io', 'time', 'jcvi.apps.socket', 'networkx.algorithms.flow.capacityscaling', 'networkx.algorithms.boundary', 'scipy.spatial.kdtree']
12:43:28 [__init__] CACHEDIR=/home/manager/.cache/matplotlib
12:43:28 [font_manager] Using fontManager instance from /home/manager/.cache/matplotlib/fontList.json
12:43:28 [__init__] backend agg version v2.2
12:43:28 [base] Load file `mcscan_layout`
12:43:28 [base] Load file `HVI.bed`
12:43:29 [base] Load file `OSI.bed`
Traceback (most recent call last):
File "/biosoft/miniconda/miniconda2/lib/python2.7/runpy.py", line 174, in _run_module_as_main
"__main__", fname, loader, pkg_name)
File "/biosoft/miniconda/miniconda2/lib/python2.7/runpy.py", line 72, in _run_code
exec code in run_globals
File "/biosoft/miniconda/miniconda2/lib/python2.7/site-packages/jcvi/graphics/karyotype.py", line 330, in <module>
main()
File "/biosoft/miniconda/miniconda2/lib/python2.7/site-packages/jcvi/graphics/karyotype.py", line 318, in main
Karyotype(fig, root, seqidsfile, layoutfile)
File "/biosoft/miniconda/miniconda2/lib/python2.7/site-packages/jcvi/graphics/karyotype.py", line 300, in __init__
tr.draw(roundrect=roundrect, plot_label=plot_label) # this time for real
File "/biosoft/miniconda/miniconda2/lib/python2.7/site-packages/jcvi/graphics/karyotype.py", line 173, in draw
si = str(int(si))
ValueError: invalid literal for int() with base 10: ''
这个错误我推测是mcscan_seqid文件中的染色体编号中必须带数字,所以我删除最后一条染色体,运行成功;
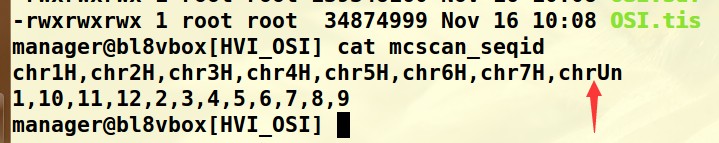
但是如果要显示不带数字的染色体,怎么办呢:可以手动修改染色体编号加上数字(mcscan_seqid),另外别忘了对应bed文件中的染色体编号也修改一下;
图片绘制出来之后可以用illustrator修改一下;
其他生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读、
- 发表于 2018-11-17 20:49
- 阅读 ( 10131 )
- 分类:软件工具
你可能感兴趣的文章
- duplicate_gene_classifier统计全基因组不同类型复制事件的数目 4350 浏览
- mcscanX基因组内共线分析输出结果解读 13169 浏览
- AttributeError: 'LayoutLine' object has no attribute 'color' 7935 浏览
- python版本MCScanX分析方法 10787 浏览
- mcsan结果文件说明 4297 浏览
相关问题
0 条评论
请先 登录 后评论
