比较3种差别表达方法对TCGA转录组数据的分析
比较3种差别表达方法对TCGA转录组数据的分析
在我们的《TCGA-基因差异表达分析》课程和《TCGA-ceRNA网络分析》的课程中,我们都介绍了针对TCGA的转录组数据,进行基因的差异表达分析。当时我们介绍的都是采用edgeR 软件包进行的分析, 有学员问我,采用DESeq2 和limma 的方法也可以进行差异表达的分析。当然,这两个软件也是可以进行差异表达分析,关键点是,这三款软件对同样的数据进行差异表达分析,差别有多大。
我们采用“TCGA-CHOL” 癌症的转录组数据进行分析,该数据集有45份样本,其中36份癌症组装,9份癌旁组织。分析采用edgeR, DESeq2, limma 进行差异分析。
1. 上调基因绘制韦恩图
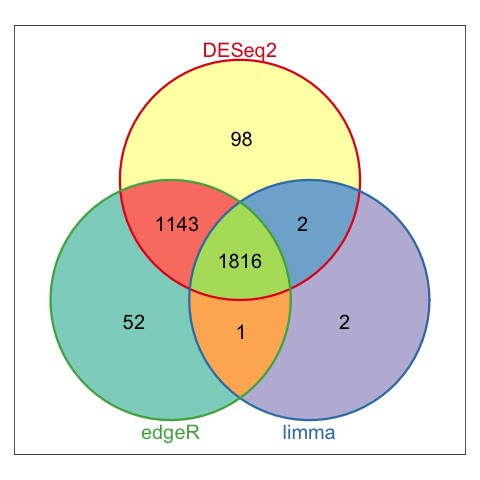
2 下调基因绘制韦恩图
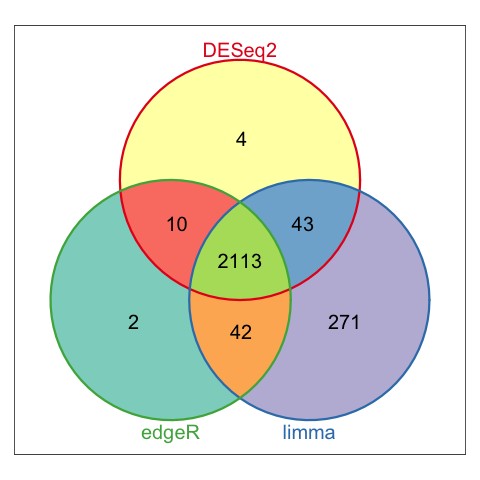
3. 差异表达基因绘制韦恩图
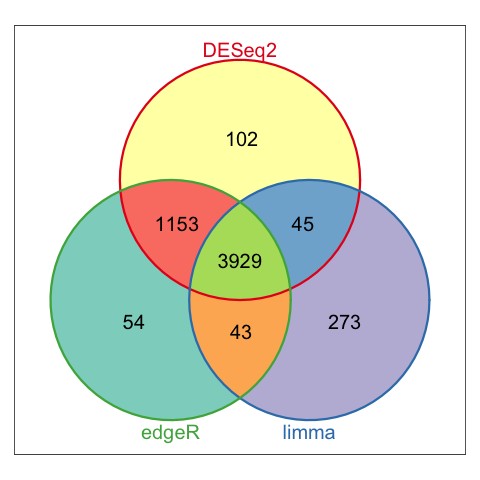
从韦恩图的分析结果来看,三款软件的分析结果,差别还是有的,其中DESeq2和edgeR的结果比较相近。所以在差异表达分析时,如果差异结果不满意,可以换一个软件试试。
- 发表于 2018-11-23 14:32
- 阅读 ( 6163 )
- 分类:TCGA
你可能感兴趣的文章
- IF:8.8|TCGA+ceRNA+肝癌文献解析 3079 浏览
- 你还在为基因芯片表达差异分析发愁吗?这个方法带你飞! 5711 浏览
- TCGA癌症和正常组织中的基因表达差异图 14976 浏览
- 计算lncRNA 与基因的表达相关性 9691 浏览
- 一个植物ceRNA数据库-pceRBase 3856 浏览
相关问题
0 条评论
请先 登录 后评论
