4个样品重测序发PlosOne
小投入高产出
全基因组重测序,大多做群体遗传进化,或者全基因组关联分析(GWAS)的确能发高分文章,很多发表在Nature,Nature genetic等国际著名期刊上。但是,对于哪些大牛的文章,动辄测几十上百甚至上千的样本量,对于经费紧张的老师也只能望而却步。然而,测少量样品能发好文章吗?回答是肯定的,可以。今天小编就来分享一篇小投入高产出的文献;4个甜瓜重测序绘制抗白粉变异图谱[1]发PLoS ONE;
文献解读
作者对白粉病不同抗性的4个甜瓜品种进行高深度重测序,四个品种中三个高抗白粉病( Edisto, MR-1, and PMR5: resistant),一个易感白粉病(SCNU1154: susceptible;),得到测序reads与参考基因组比对,查找SNP,Indel,SV等变异信息,并对变异信息进行质控过滤,变异信息在染色体上的分布比较,统计信息;这些都是常规重测序变异检测分析内容;
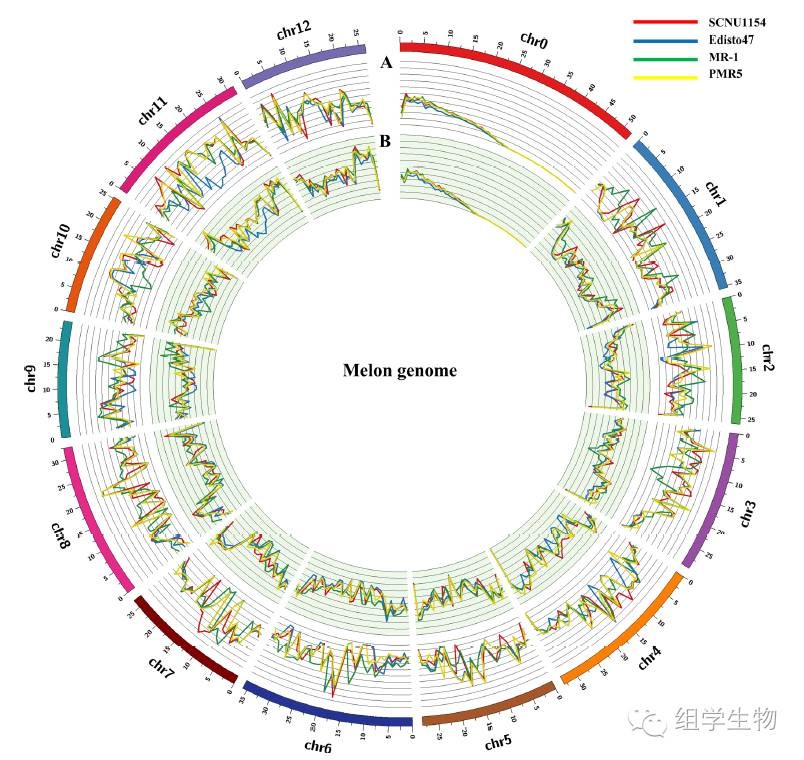
重要的分析内容, 也是文章的亮点在于,作者额外鉴定了甜瓜基因组中一类疾病抗性基因家族(NBS),NBS基因家族在植物中广泛存在,常见的有
CNL [CC-NB-LRR, proteins containing coiled-coil (CC), nucleotide-binding site (NBS), and leucine-rich repeat (LRR) domains], TNL [TIR-NB-LRR, Toll-interleukin receptor-like, nucleotide binding site (NB) and LRR domains], RLP (receptor serine/threonine kinase-like domain), and an extracellular LRR (ser/thr-LRR); 这些基因往往和植物抗病性相关,因此,找到抗性品种与感病品种在这些基因上不同的变异信息非常重要,因为这些变异位点很可能与植株的抗病性获得息息相关,作者通过感抗品种变异信息的对比和筛选,绘制了抗性位点的变异图谱,并用不同的颜色标注变异与抗性的相关性,为后续的研究提供了研究方向和基础;

参考文献如下:
[1] Natarajan S, Kim H-T, Thamilarasan SK, Veerappan K, Park J-I, Nou I-S. Whole Genome Re-Sequencing and Characterization of Powdery Mildew Disease-Associated Allelic Variation in Melon. Gent D, ed. PLoS ONE. 2016;11(6):e0157524. doi:10.1371/journal.pone.0157524.
- 发表于 2018-04-22 15:04
- 阅读 ( 3321 )
- 分类:文献解读
你可能感兴趣的文章
- 基因组重测序docker镜像更新到2.0 2307 浏览
- 简述一代、二代、三代测序技术在应用上的区别 11913 浏览
- 性价比超高5个重测序发IF=6遗传进化文章 3745 浏览
相关问题
- 重测序 1 回答
- picard处理后的bam文件被gatk识别为多个样本怎么办? 2 回答
- docker下载镜像时出现问题 1 回答
- 重测序 1 回答
- 重测序 1 回答
- 关于使用nohup vcftools过滤vcf文件的问题 1 回答
