计算lncRNA 与基因的表达相关性
计算lncRNA 与基因的表达相关性
在构建ceRNA 网络时,需要计算lncRNA 与 蛋白编码gene (pc gene) 间的表达相关性,一般采用皮尔逊相关系数。具体如何做呢?
1. 首先获得lncRNA 的表达矩阵
> head(lncRNA)
lncRNA1 lncRNA2 lncRNA3 lncRNA4 lncRNA5
sam1 7.80 5.81 6.40 6.11 4.87
sam2 1.47 1.75 2.49 2.20 2.02
sam3 0.00 0.00 1.28 0.00 0.00
sam4 0.00 0.00 1.74 0.00 0.00
sam5 1.83 0.99 0.72 0.69 0.50
sam6 0.00 4.04 0.00 0.00 0.00
> dim(lncRNA)
[1] 100 5
从lncRNA 的表达数据来看,是5个lncRNA 在100个样本中的表达量矩阵。
2. 获得 gene 的表达矩阵
> head(gene)
gene 1 gene 2 gene 3 gene 4
sam1 7.66 7.19 7.30 6.98
sam2 1.34 0.29 1.77 1.61
sam3 0.00 0.00 0.00 0.00
sam4 0.14 0.41 0.56 0.77
sam5 0.50 0.48 0.73 0.27
sam6 0.00 0.00 0.00 0.00
> dim(gene)
[1] 100 4
从基因的表达矩阵来看,是4个基因在100个样本的表达量矩阵。
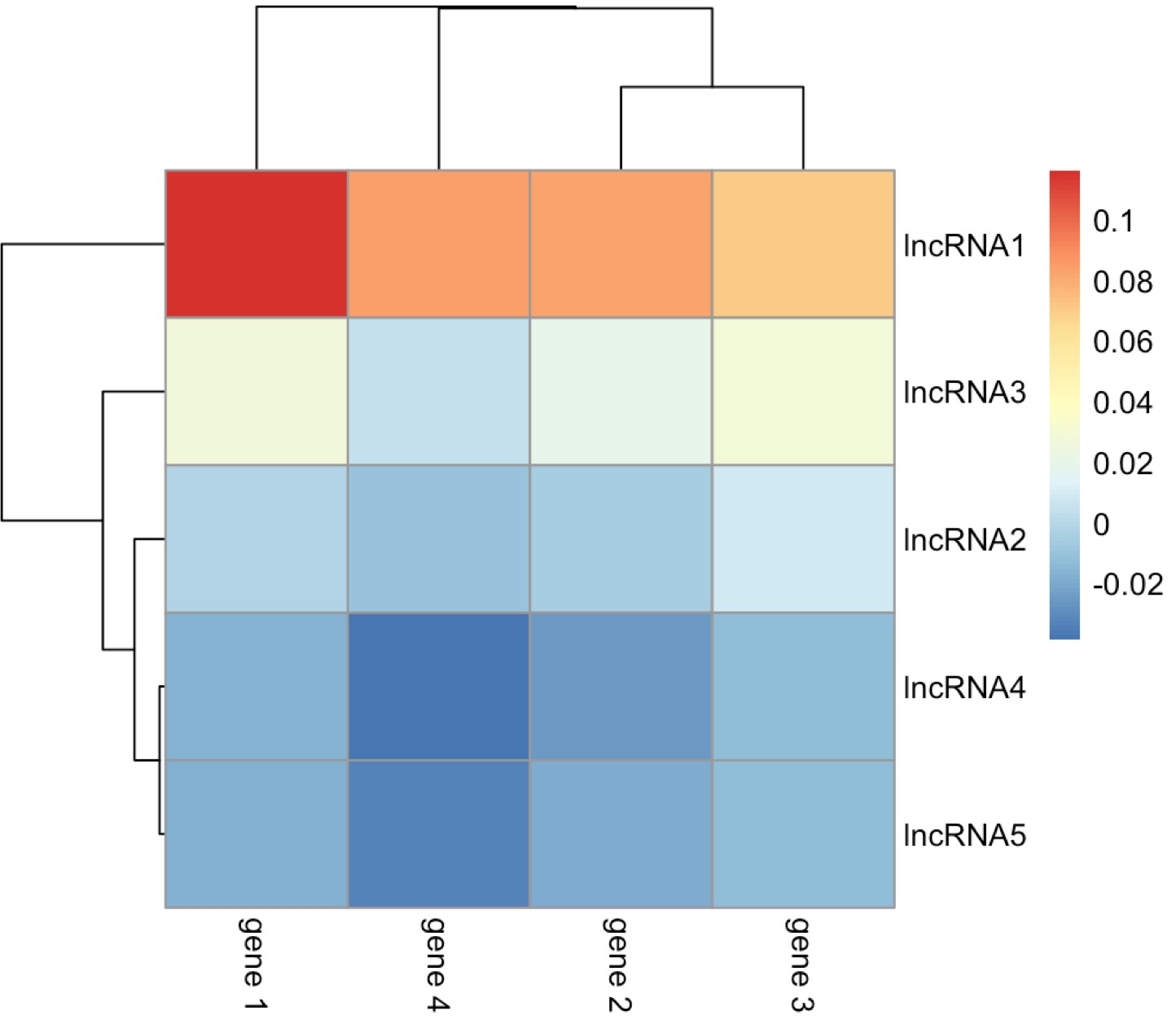
3. 计算lncRNA 与gene 的表达相关性
# 相关性计算,就这么简单
> cor_matrix <- cor(lncRNA,gene)
> cor_matrix
gene 1 gene 2 gene 3 gene 4
lncRNA1 0.116393943 0.082464228 0.070419800 0.08509885
lncRNA2 -0.001455028 -0.004848818 0.008653588 -0.01047322
lncRNA3 0.027655655 0.019760497 0.029196236 0.00464662
lncRNA4 -0.016414621 -0.024633893 -0.012498862 -0.03835430
lncRNA5 -0.018116788 -0.018668500 -0.013198682 -0.03287980
> pheatmap(cor_matrix)
采用cor 函数计算相关性,并绘制热图,如下:
如果你想学习lncRNA与基因表达的相互调控,可以学习我的ceRNA课程。
- 发表于 2018-12-07 10:00
- 阅读 ( 10257 )
- 分类:TCGA
你可能感兴趣的文章
- R语言计算两组数据相关性及对应p值的方法——psych包和Hmisc包 23087 浏览
- IF:8.8|TCGA+ceRNA+肝癌文献解析 3868 浏览
- 蛋白质组和转录组相关性图绘制 6398 浏览
- 比较3种差别表达方法对TCGA转录组数据的分析 6873 浏览
- 一个植物ceRNA数据库-pceRBase 4291 浏览
相关问题
0 条评论
请先 登录 后评论
