进化树中如何添加分组圆环
进化树中如何添加分组圆环
最近有小伙伴问小编怎样才能在进化树外层加圆环,以体现样品的分组(如下图所示),其实很简单,小编就来讲讲怎样实现吧。
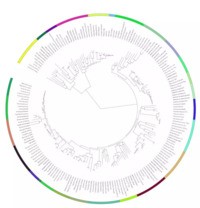
使用的工具是iTOL这款在线软件,可点这里 绘一棵超酷炫的系统发育树 了解iTOL的使用方法。基础的使用方法就不再讲述了,只对配置文件进行说明。
注释文件准备
首先来看一下所需要的注释文件的格式:
DATASET_COLORSTRIP
#lines starting with a hash are comments and ignored during parsing
SEPARATOR TAB
DATASET_LABEL color_strip2
STRIP_WIDTH 25
MARGIN 0
BORDER_WIDTH 1
BORDER_COLOR #000
SHOW_INTERNAL 0
DATA
160232 #efff22
13773 #efff22
56636 #efff22
111955 #efff22
2287 #efff22
50339 #efff22
2303 #efff22
2234 #efff22
64091 #efff22
2214 #efff22
2209 #117355
2261 #117355
格式说明:
注释文件中以"#"开头的行是注释行,只起说明作用,iTOL不会读取该行的内容。
DATASET_COLORSTRIP:注释文件数据类型声明,这里声明数据集是为进化树外层添加彩色圆环。
SEPARATOR TAB:指定数据分隔符,即数据列与列之间使用Tab分隔。也可指定分隔符为空格,需将"TAB"改为"SPACE"。
DATASET_LABEL color_strip2:指定数据集标签,标签可任意。
STRIP_WIDTH 25:指定圆环的宽度。
MARGIN 0:指定圆环与进化树之间的距离,可为负数,会导致与进化树重叠。
BORDER_WIDTH 1:指定圆环边框的宽度。
BORDER_COLOR #000:指定圆环边框的颜色。
如果没有特殊设置,以上部分通常不需要改,直接拷贝使用即可。
DATA 之后的行就是所有的分组数据信息。数据分为两列,第一列为进化树叶节点ID,第二列是叶节点关联的颜色,每种颜色代表一个分组,不同分组的样品对应相应的颜色。可以用十六进制(#开头的颜色值),RGB表示法指定颜色。
注释文件美化
按照以上格式准备好注释文件,准备好文件后直接将其拖到iTOL进化树展示所在的位置,就能自动给进化树外层加上圆环了。之后也可以在Datasets中对圆环位置、宽度以及圆环边框的粗细、颜色等进行微调。
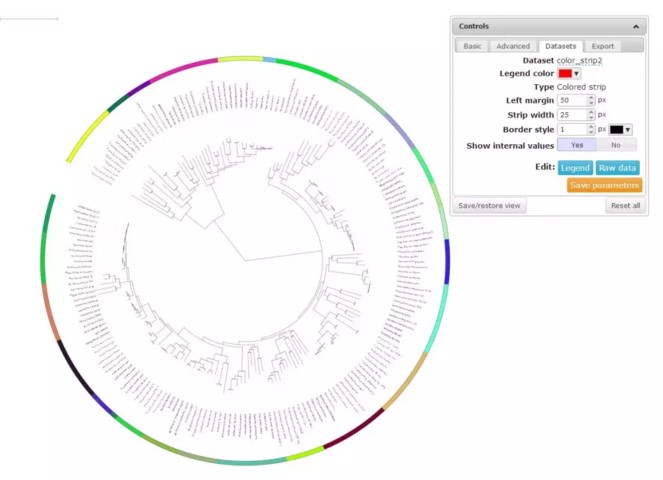
以上操作介绍完了,怎么样?是不是很简单?有兴趣的小伙伴自己动手试试吧!
最后祝您科研愉快!
- 发表于 2018-12-14 13:55
- 阅读 ( 12032 )
- 分类:软件工具
你可能感兴趣的文章
- 给进化树标定化石时间 1720 浏览
- 快速生成iTOL配置文件美化你的进化树 2017 浏览
- itol添加圆环分组和节点标签时位置调整 4497 浏览
- 如何用R语言绘制进化树+热图 9900 浏览
- 进化树构建的方法及原理详解 41122 浏览
- 如何利用iTOL绘制漂亮的进化树 16417 浏览
0 条评论
请先 登录 后评论
