circos-基因组圈图绘制软件基础绘图
Circos(官网链接:http://circos.ca/)是一款非常nice的基因组圈图绘制软件,它所绘制的图片,emmm……你想有多漂亮就能有多漂亮。它提供了部分在线绘图的功能,可以戳这里了解一下:教你绘制circos图。但是基于对图片的要求不同,掌握了命令行的方式进行绘图,能够获得的结果也就越完善。这里就从最简单的入手(如下图),来教大家掌握绘图方法!
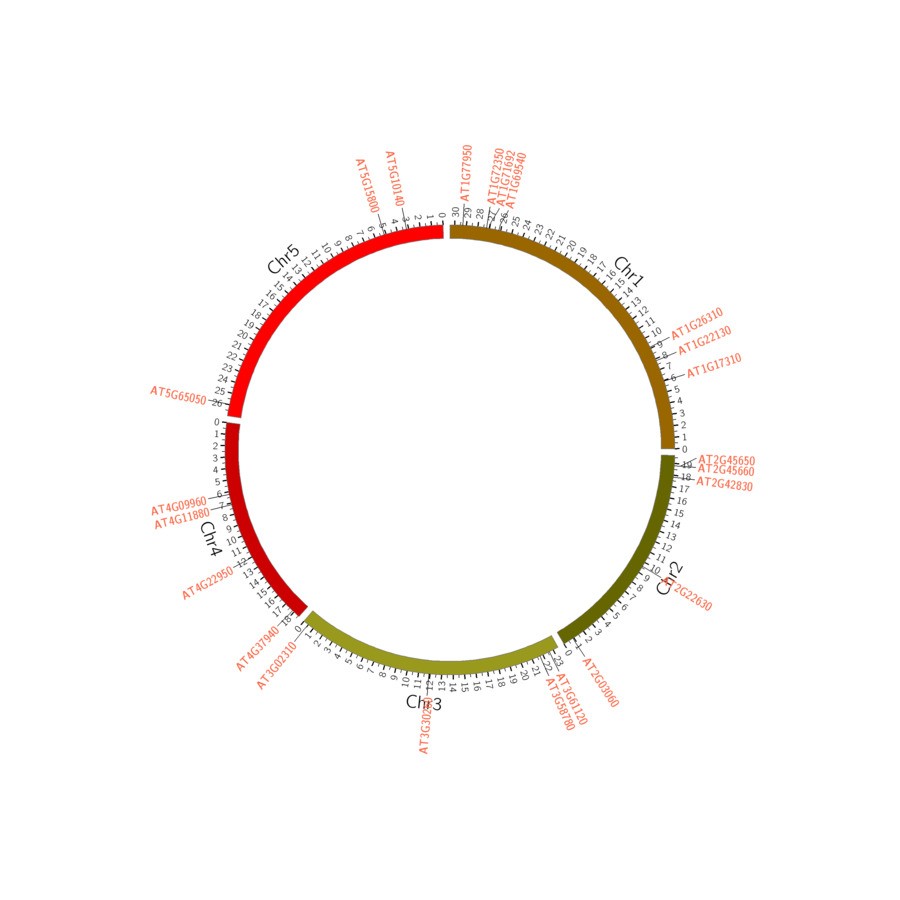
数据准备
参考上图,绘制需要两项基本内容,其一是染色体信息文件(核型文件),主要目的是在图片上显示出染色体,并进行颜色等相关设置;其二是标签文字文件(文本text文件),主要目的是在染色体上具体的位置将基因作为label显示。
核型文件(chr.info)
格式如下所示:
chr - 1 Chr1 0 30427671 chr1
chr - 2 Chr2 0 19698289 chr2
chr - 3 Chr3 0 23459830 chr3
chr - 4 Chr4 0 18585056 chr4
chr - 5 Chr5 0 26975502 chr5
文件以TAB分隔,前两列可以不变,而需要注意第三列到最后一列的信息,第三列为染色体实际编号,第四列是图片上进行显示的编号,第五列表示染色体长度的起始,第六列对应染色体的终止,也就是最终长度,最后一列实际上控制的是染色体的颜色,chr1至chr5都对应不同的颜色。
text文件(text.txt)
文件格式如下(全部内容共24行,此处仅显示部分):
5 25982254 25986435 AT5G65050
4 17835217 17838969 AT4G37940
4 12023824 12028004 AT4G22950
3 22618259 22620529 AT3G61120
1 25982294 25986349 AT1G69120
文件依然以TAB分隔,第一列表示标签文字(基因)出现的染色体实际编号,第二列第三列表示基因位于该染色体上的起止坐标位置,第四列是该基因的ID即在染色体上显示的文字。
绘图配置
进行circos绘图,需要依据最终目标整理配置文件,也就是对图片进行各种细节设置,以目标图片为例,配置文件(config.txt)如下:
chromosomes_units=100000 #染色体单位长度
#chromosomes_reverse=/[12345]/ #是否对匹配到的染色体进行反转
#####针对染色体显示进行设置
<ideogram>
fill=yes #是否进行颜色填充
label_font=default #染色体标签字体
label_parallel=yes #字体方向易于浏览
label_radius=1.1r #标签位置
label_size=45 #标签字体大小
radius=0.5r #染色体位置
show_label=yes #是否显示染色体标签
###针对染色体之间空隙大小进行设置
<spacing>
default=0.005r
</spacing>
stroke_color=dgrey #染色体外边框轮廓颜色
stroke_thickness=2p #轮廓厚度
thickness=0.03r #染色体厚度
</ideogram>
karyotype=/share/circos/result/chr.info ##核型文件位置
#######此处仅显示针对标签text进行设置
<plots>
<plot>
type=text #限定plot为文本标签
color=red #限定文字标签颜色
file=/share/circos/result/text.txt ##text文件位置
label_font=condensed #文字字体类型
label_size=30 #文字字体大小
label_snuggle=yes #如多个文本文字距离过近,避免重叠 参考:https://www.omicsclass.com/article/678
show_links=yes #显示标签和染色体之间的连接
link_color=black #连接线颜色
link_dims=0p,20p,50p,2p,2p #连接线细分五部分,各部分长度
link_thickness=2p #连接线厚度
r0=1r #标签位置,其r0和r1大小能控制标签在内或在外
r1=1r+200p
rpadding=5p #文字边缘大小
</plot>
</plots>
#######针对染色体刻度开始进行设置
show_tick_labels=yes #是否显示刻度标签
show_ticks=yes #是否显示刻度
<ticks>
color=black #刻度线颜色
multiplier=1e-6 #刻度标签显示值的计算,显示刻度的位置对应该值的大小
radius=1r #显示位置
thickness=2p #刻度线厚度
##设置每5u的刻度并进行细节设置
<tick>
size=10p #刻度线大小
spacing=5u #每5个单元长度出现一个刻度
</tick>
##设置每10u的刻度并进行细节设置
<tick>
color=black #刻度线颜色
format=%d #标签数字格式,%d表示整数,%f浮点数
label_offset=10p #标签和刻度之间的距离
label_size=25p #标签大小
show_label=yes #显示标签,标签值需按照multiplier计算
size=15p
spacing=10u #每10u出现一个刻度
thickness=4p
</tick>
</ticks>
###其他基础设置
<colors>
<<include etc/colors.conf>>
<<include etc/brewer.conf>>
</colors>
<fonts>
<<include etc/fonts.conf>>
</fonts>
<image>
<<include etc/image.conf>>
</image>
<<include etc/housekeeping.conf>>
命令行绘图
在安装软件之后,运行绘图命令,即可获得基础图片:
/share/work/biosoft/circos/circos-0.69/bin/circos -conf config.txt --outputdir ./ -outputfile test
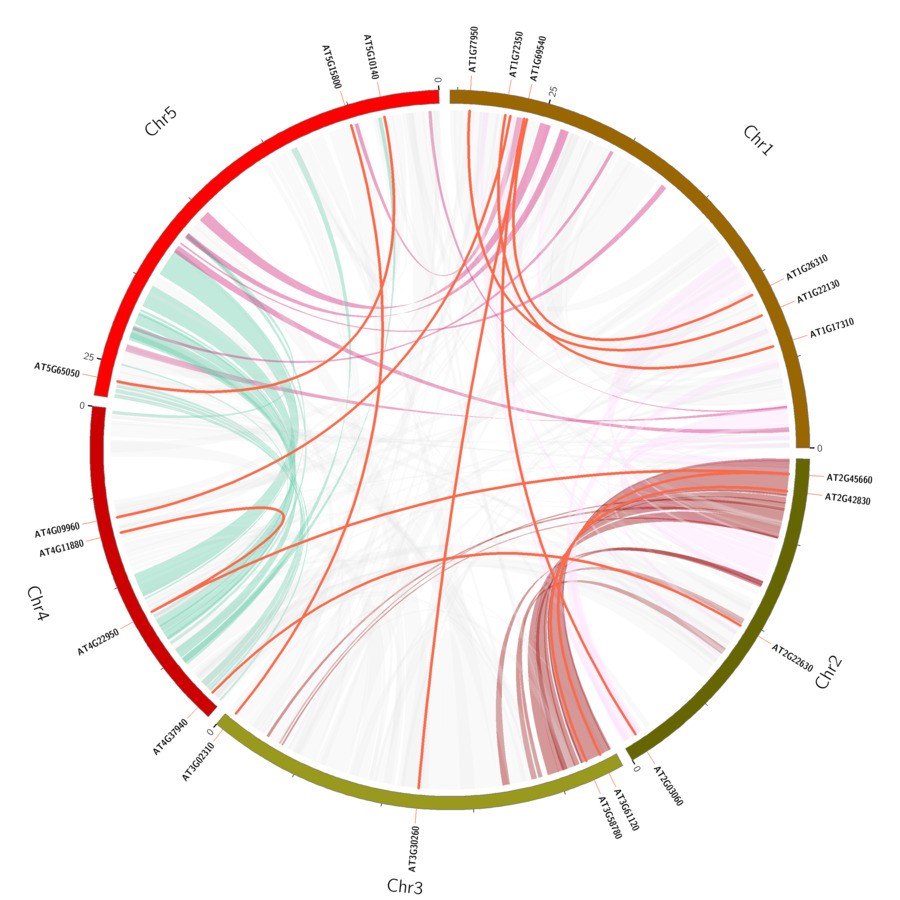
当然实际绘图配置文件更复杂,涉及的参数也更多,譬如基因组共现线圈图(见下图),感兴趣的同学可以在circos官网学习更细节的设置,或者点击下方“相关课程”学习相关的绘图方法。
相关课程:
基因家族分析实操课程、基因家族文献思路解读
R语言绘图基础(ggplot2)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-12-21 13:24
- 阅读 ( 18377 )
- 分类:科研作图
你可能感兴趣的文章
- circos改变某两条染色体之间的距离 1583 浏览
- circos|修改不同染色体之间区块block及连线link的颜色 2438 浏览
- circos当中的颜色介绍 11241 浏览
- circos-基因组圈图调整标签文字重叠 10030 浏览
- circos-调整基因组圈图的图片大小 6390 浏览
