vcftools过滤变异类型
vcftools过滤变异类型
vcf文件中可能会同时是包含snp以及indel两种变异类型,如果想将其分开,可利用vcftools实现。
具体用法如下:
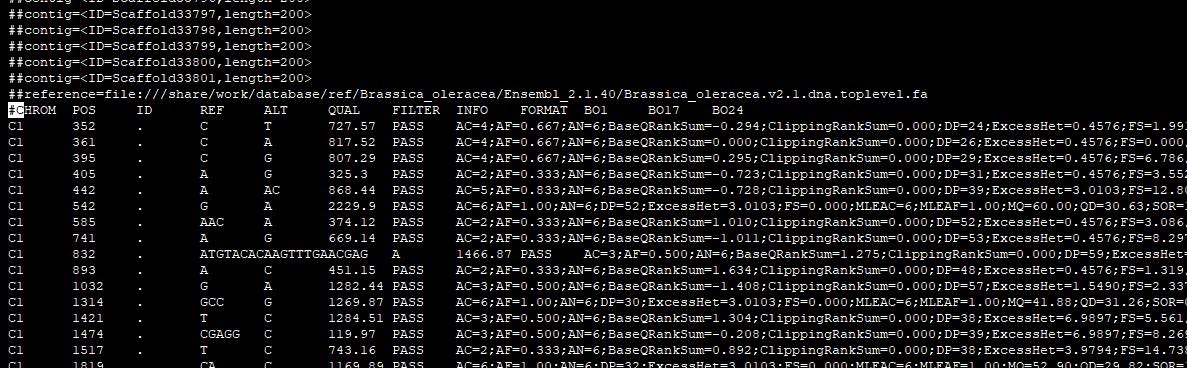
下图为原始vcf文件(raw.vcf),可以看到包含indel以及snp。
过滤掉indel,只保留snp --remove-indels。
执行以下命令:
vcftools --remove-indels --recode --recode-INFO-all --vcf raw.vcf --stdout >raw.snp.vcf
过滤掉snp,只保留indel --keep-only-indels。
执行以下命令:
/vcftools --keep-only-indels --recode --recode-INFO-all --vcf raw.vcf --stdout >raw.indel.vcf
这样,就可以分别得到只包含snp和indel的vcf文件了。
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2019-01-04 14:27
- 阅读 ( 7931 )
- 分类:软件工具
你可能感兴趣的文章
- 使用vcftools对vcf文件做滑窗处理 2082 浏览
- 多款软件进行vcf合并--gatk、vcftools、bcftools 8099 浏览
- vcf格式文件基础知识与编辑操作详解 17701 浏览
- vcftools使用 8547 浏览
- vcftools过滤指定缺失率的变异位点 10891 浏览
相关问题
- 老师 就是 我在过滤数据的时候 下面截屏中利用vcfutils.pl进行过滤的时候 生成的文件还有7.2 Gb,但是用vcftools进行过滤后还剩764Mb的文件,这个是不是过滤掉的太多啦呀 麻烦您重新帮我设置一个过滤参数嘛 谢谢我重测序的个体才36个 1 回答
- 重测序遗传变异过滤 0 回答
- 老师,在第三步vcf文件过滤时,得到的all.clean.vcf文件太小了,宽松设置调整数值后,依旧很小,怎么办啊,有没有其他的方法,可以让最后的数据大一些啊 1 回答
- 怎么过滤vcf文件中INFO列中的DP参数? 1 回答
- 怎么过滤vcf文件中INFO列中的DP参数? 2 回答
- 变异结果质控过滤,去掉低质量变异结果时报错i 1 回答
0 条评论
请先 登录 后评论