circos-基因组圈图调整标签文字重叠
利用circos绘制基因组圈图(可参考:https://www.omicsclass.com/article/644),对部分位置进行文字标签标注,由于位置较近,很容易导致文字重叠,在绘图时可以利用plot中对应的参数进行控制:
(具体请结合参考:http://circos.ca/tutorials/lessons/2d_tracks/text_labels1/images 和 http://circos.ca/documentation/tutorials/2d_tracks/text_labels1/lesson )
label_snuggle=yes #如多个文本文字距离过近,避免重叠
show_links=yes #显示标签和染色体之间的连接
link_color=black #连接线颜色
link_dims=0p,20p,50p,2p,2p #连接线细分五部分,各部分长度
link_thickness=2p #连接线厚度
r0=1r #标签位置,其r0和r1大小能控制标签在内或在外
r1=1r+200p
如上所述,选择label_snuggle为yes ,同时结合link_dims五个值进行控制,譬如五部分值为,可以获得如下图结果:
link_dims=10p,10p,50p,20p,10p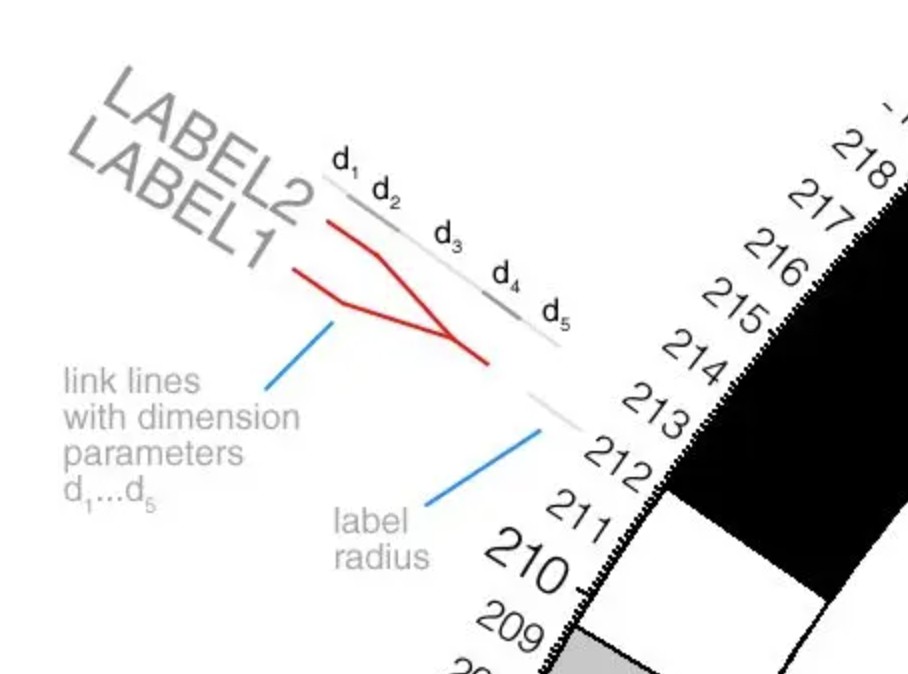
C和D还是在位置上重叠,只不过径向上一前一后进行移动了。
但是当标签文字过长还是可能会出现重叠问题,或者隐藏,譬如:
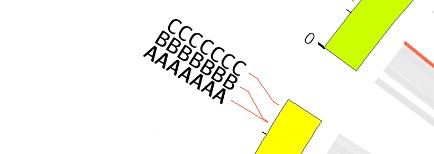
此时可以进一步调整的参数 r0=1r和 r1=1r+200p
将r1位置调大,譬如修改如下可获得结果如下图:
link_dims=10p,10p,50p,20p,10p
r0=1r
r1=1r+500p
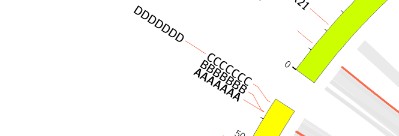
综上其实可以发现,当标签超过r0和r1的位置时,其实这个标签不会进行绘制的。 所以即使此刻度只有一个标签,当其名称过长,r0与r1控制的位置小,也不会绘制这个标签。

除了控制r0和r1之外,还可以对两个参数进行控制,控制文字位置,分别为
rpadding ###径向控制文字位置(可正可负)
padding ###角度上控制文字
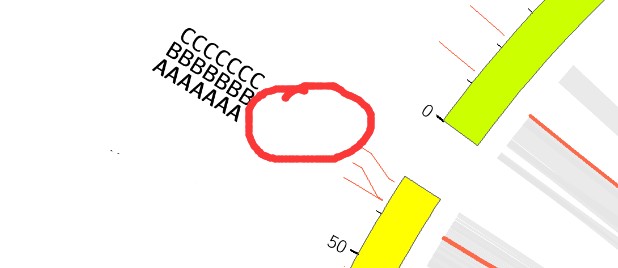
1、rpading会导致整体移动,并没有对重叠问题有帮助,除非同时调整r0和r1
link_dims=10p,10p,50p,20p,10p
r0=1r
r1=1r+200p
rpadding=200p
2、padding,在角度上调整,会调整标签重叠,数值可正可负,譬如正值结果活负值结果不同:
link_dims=10p,10p,50p,20p,10p
r0=1r
r1=1r+200p
padding=0.25r
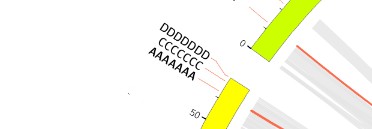 很神奇的隐藏了B,而修改为负值:
很神奇的隐藏了B,而修改为负值:
link_dims=10p,10p,50p,20p,10p
r0=1r
r1=1r+200p
padding=-0.25r
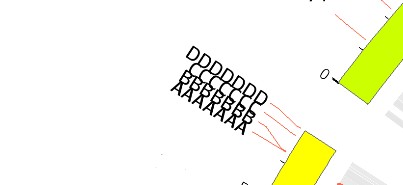 四个标签都存在,比较拥挤,之后简单调整一下link_dims即可
四个标签都存在,比较拥挤,之后简单调整一下link_dims即可
更多生物信息课程:https://study.omicsclass.com/index
- 发表于 2019-01-17 17:39
- 阅读 ( 9372 )
- 分类:软件工具
你可能感兴趣的文章
- circos改变某两条染色体之间的距离 981 浏览
- circos|修改不同染色体之间区块block及连线link的颜色 1913 浏览
- circos当中的颜色介绍 10619 浏览
- 部分基础绘图函数隐藏坐标轴(并重绘) 4722 浏览
- circos-调整基因组圈图的图片大小 5955 浏览
