进化树中如何添加热图?
之前小编写了很多iTOL绘制进化树的文章,今天小编再介绍一下进化树中添加热图(如下图所示)的方法。
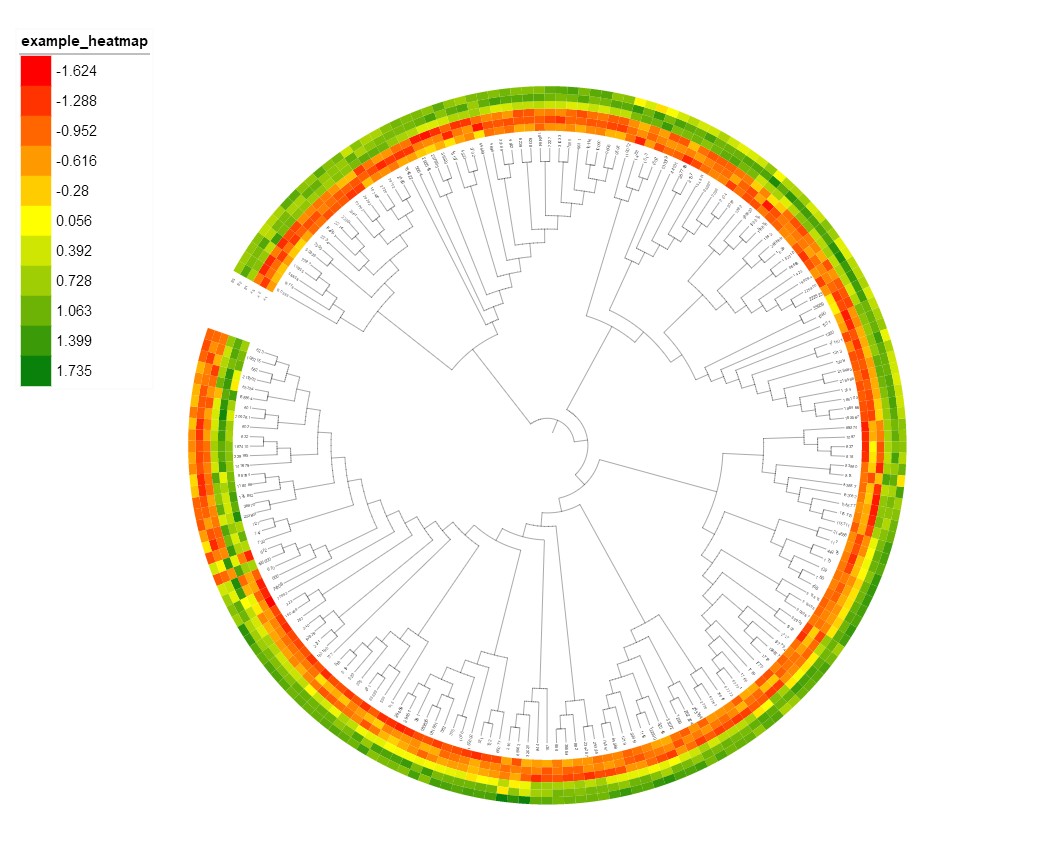
使用的工具依然是iTOL这款在线软件,可点这里 绘一棵超酷炫的系统发育树 了解iTOL的使用方法。基础的使用方法就不再讲述了,只对配置文件进行说明。
配置文件准备
首先来看一下所需要的文件的格式:
DATASET_HEATMAP
#SEPARATOR TAB
SEPARATOR SPACE
DATASET_LABEL example_heatmap
COLOR #ff0000
#define labels for each individual field column
FIELD_LABELS A1 A2 A3 B1 B2 B3
MARGIN 0
STRIP_WIDTH 25
COLOR_MIN #ff0000
COLOR_MAX #0000ff
USE_MID_COLOR 1
COLOR_MID #ffff00
DATA
155864 1.33118766397716 1.17857919899514 -0.409551070843932 -0.630306286554169 -1.04604589954572 -0.423863606028482
83334 -0.0506076910870376 1.3902493708319 1.06059120726857 -0.886946488704782 -0.820282432690725 -0.693003965617928
217992 0.215479234585023 1.4989464081584 0.688218558910058 -1.21085219033218 -0.764324705926616 -0.427467305394685
562 1.06564649867261 0.961867445609222 0.597829776500456 -0.461509960030431 -1.19966321785905 -0.964170542892806
198215 0.96900544380798 1.21939609883181 0.426791627672821 -0.838548688929931 -0.653211840822719 -1.12343264055996
623 0.727282455309188 1.17101033530621 0.806443403628944 -0.820220618604714 -0.920091135497558 -0.964424440142074
209261 0.34017826857092 1.66855815466279 0.357643216125776 -0.545542360482732 -1.00245964906156 -0.818377629815193
601 0.693183645180168 1.46683923827496 0.279576953582569 -1.04280232644501 -1.03511634861385 -0.36168116197883
602 0.70273801067879 1.37079662629187 0.366781728164418 -0.773552511347515 -1.33593177597361 -0.330832077813954
229193 0.877365674482512 1.19322948550412 0.554384448895058 -0.958849032442236 -1.14266481591487 -0.523465760524581
格式说明:
配置文件中以"#"开头的行是注释行,只起说明作用,iTOL不会读取该行的内容。
DATASET_HEATMAP:配置文件数据类型声明,这里声明数据集是对进化树添加热图信息。
SEPARATOR SPACE:指定数据分隔符,即数据列与列之间使用空格分隔。
COLOR #ff00ff:指定数据集初始颜色,可在后面进行更改。
FIELD_LABELS A1 A2 A3 B1 B2 B3:为DATA中的每一列数据定义标签(指定样品名称)。
MARGIN 0:指定热图与进化树之间的距离,可为负数,会导致与进化树重叠。
STRIP_WIDTH 25:指定热图中每一格的宽度。
COLOR_MIN #ff0000:指定热图中最小值的颜色。
COLOR_MAX #0000ff:指定热图中最大值的颜色。
USE_MID_COLOR 1:指定是否设置中间值颜色,0代表不设置,1代表设置。
COLOR_MID #ffff00:指定热图中中间值的颜色。
如果没有特殊设置,以上部分通常不需要改,直接拷贝使用即可。
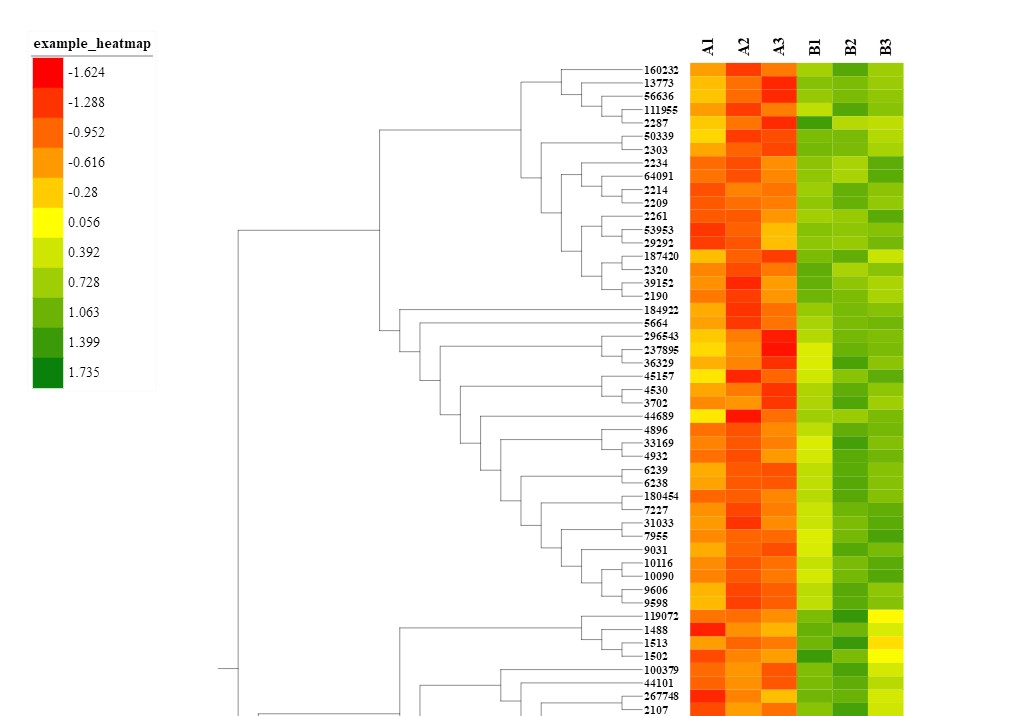
DATA 之后的行就是所有的基因在各样品中的表达量数据,这里我用的数据是基因在样品中的表达量水平log2(FPKM)进行归一化转换(scale number)后的值。数据分为七列,第一列为进化树枝节点ID(基因ID),第二到第七列为基因在各样品中的表达量数据,注意数据要与 FIELD_LABELS 所指定样品名称的数量及顺序保持一致。
注释文件美化
按照以上格式准备好注释文件后,直接将其拖到iTOL进化树展示所在的位置,就能自动在进化树中添加热图了。如果对图片的效果不满意,还也可以在Datasets中对热图的颜色、宽度及位置进行调整。
以上操作介绍完了,怎么样?是不是很简单?有兴趣的小伙伴自己动手试试吧!
祝您科研愉快!
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2019-01-18 09:28
- 阅读 ( 9579 )
- 分类:软件工具
你可能感兴趣的文章
- 给进化树标定化石时间 2299 浏览
- 快速生成iTOL配置文件美化你的进化树 2484 浏览
- 如何用R语言绘制进化树+热图 10449 浏览
- 进化树构建的方法及原理详解 43440 浏览
- 多种进化树编辑处理软件 4349 浏览
- Evolview绘制进化树时添加热图 4821 浏览
