OTU互作网络分析用MENA
前言
生物之间相互作用比如竞争、合作、共生等;由于进化生物之间存在某种系统发育关系,这些联系都能描述为生态网络,在生态网络中,每一个元素(生物或基因)可以描述成网络中的一个节点,他们之间的关系能描述成网络中的边。随着高通量测序、基因芯片等技术的发展,研究人员可以得到了空前庞大的数据量,而利用微生物16S测序数据得到的OTU丰度矩阵也可以进行网络分析,探究不同操作单元之间的互作关系。
因此,今天要给大家推荐一个网站:MENA,一个可以利用OTU数据进行在线分子生态网络分析的网站。
准备数据
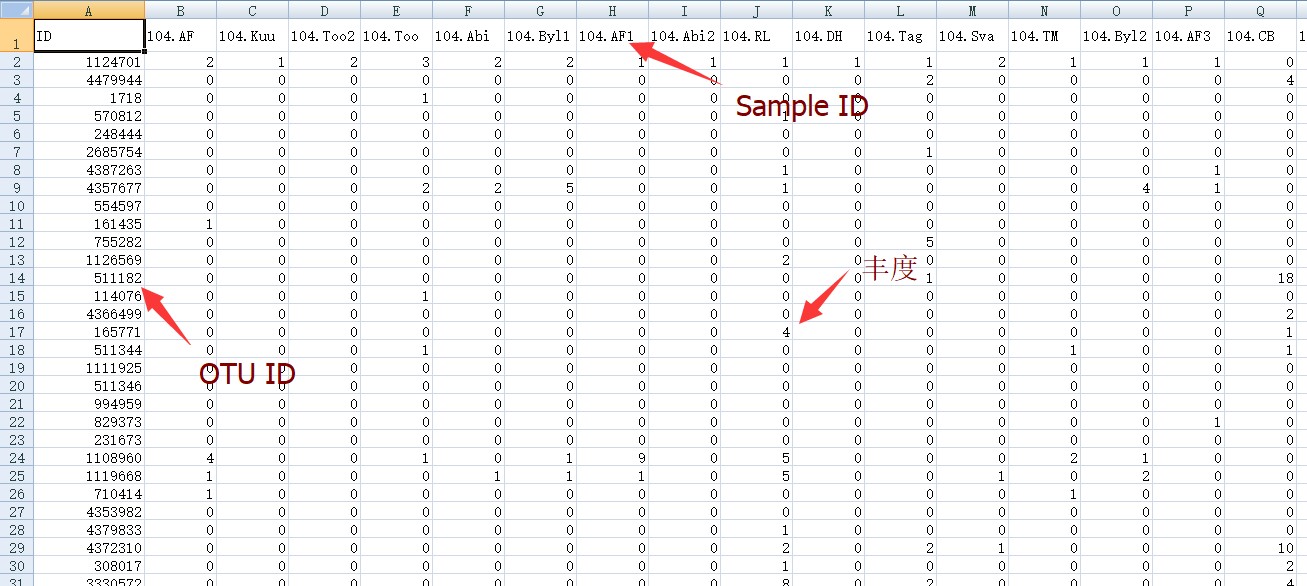
网络分析的输入数据为OTU丰度矩阵,每一行对应一个OTU,每一列对应OTU在不同样品中的丰度,具体格式如下图
网站登陆注册
网站地址: http://ieg4.rccc.ou.edu/MENA/,(个别情况下,网站可能无法访问,正常等待几天再尝试),网站登陆界面如下,如无账号可以进行注册并登陆,注册只需要邮箱等信息即可。

登陆后界面的主要操作内容为前四部份,涉及上传数据、构建网络、分析网络以及查看此前上传和分析的网络结果。
上传数据
点击主页左侧Upload your dataset选项后,选择对应的文件并且对数据进行命名后提交,提交过程中注意网站对文件的要求,包括文件大小,格式等等(下图)。提交成功后会自动显示上传数据的大致信息,包括样本数量、OTU数量等等。
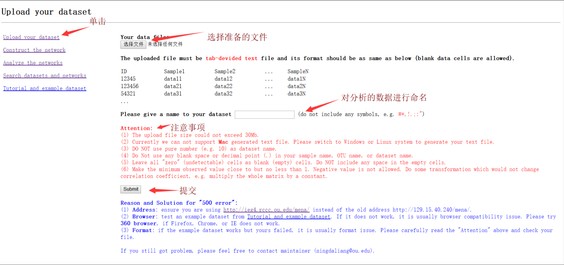
提交成功后会自动显示上传数据的大致信息,包括样本数量、OTU数量等等。
构建网络
点击左侧Construct the network,跳转到Select Dataset页面,选择需要进行网络分析的数据进行下一步,提交之后进入参数设置页面(如下图),具体涉及了数据过滤,相似性计算算法,RMT(随机矩阵理论)模型计算,等等(理论和应用文章见末尾文献引用),提交后跳转至等待页面。

注意:基于RMT 模型计算的过程耗时可能非常久,可以直接进入主页后进行等待,在几个小时候后再继续后续分析。
从主页Search datasets and networks,查看分析的结果,计算完成后,会显示状态为"Done",单击对应的结果,进行网络构建。
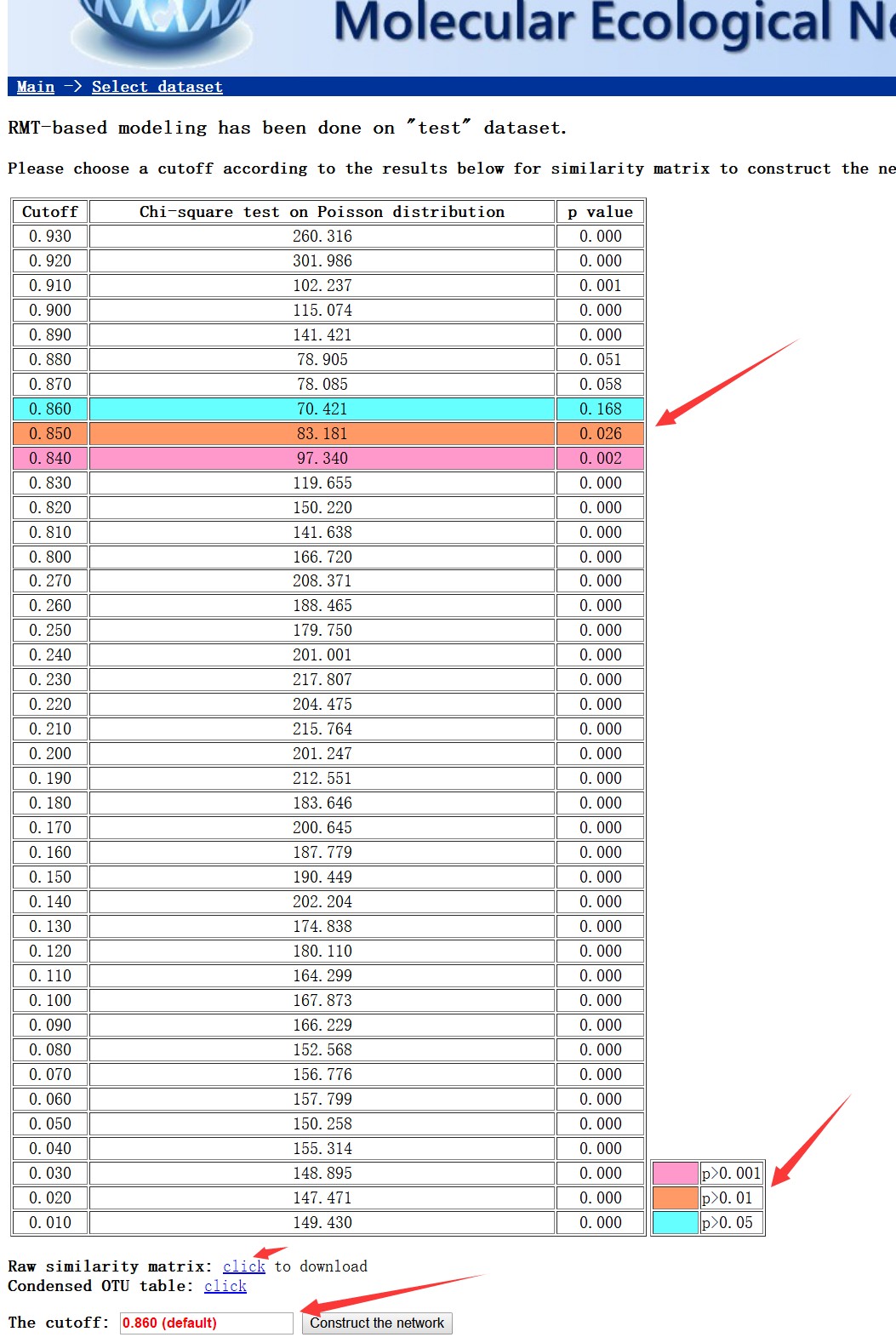
 RMT计算结果,选择合适的相关性阈值构建网络(如下图)。网站会自动计算构建的网络中节点数量,连接数量,平均连通性等网络的基本特征值,并显示。
RMT计算结果,选择合适的相关性阈值构建网络(如下图)。网站会自动计算构建的网络中节点数量,连接数量,平均连通性等网络的基本特征值,并显示。
网络分析
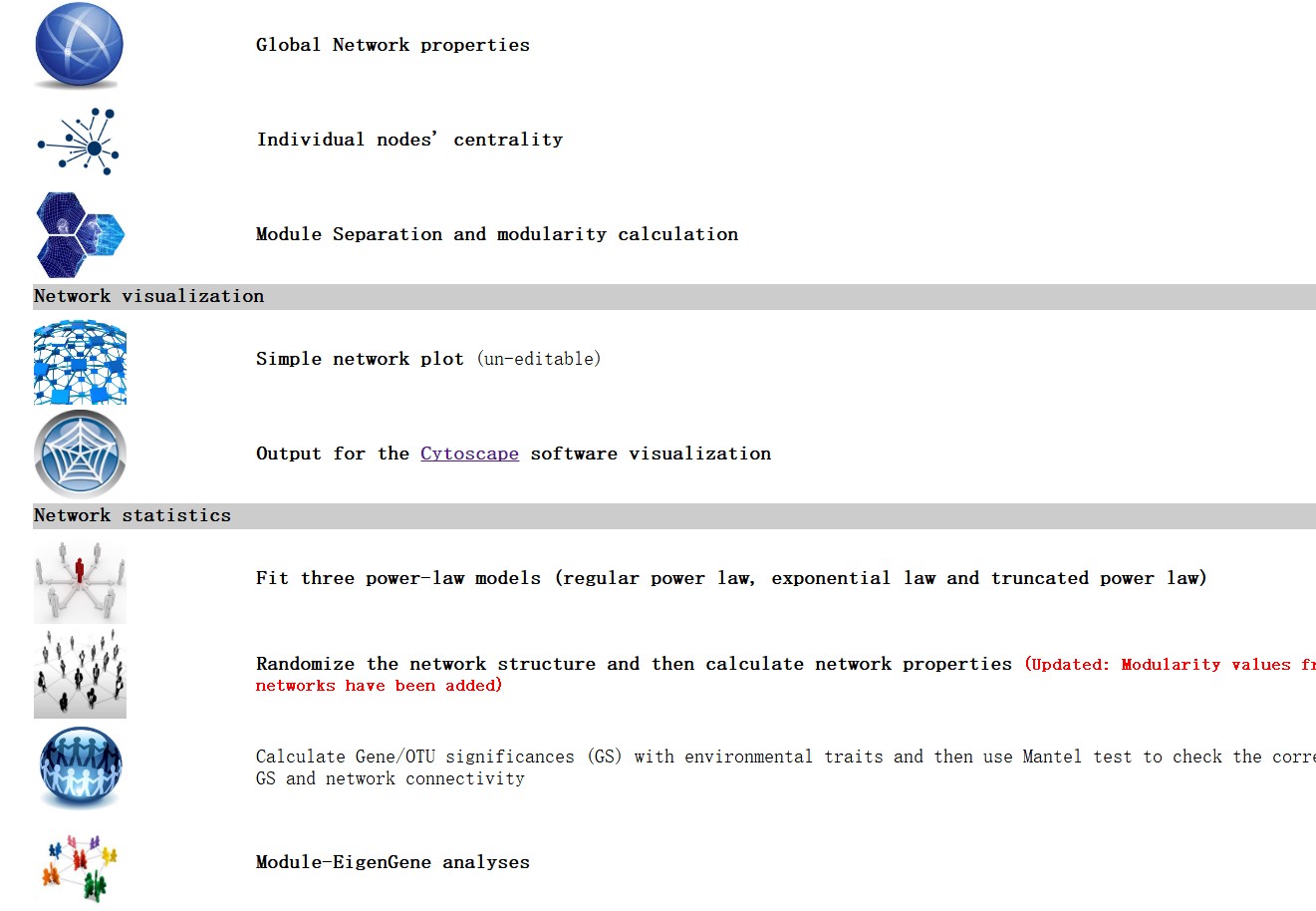
完成网络构建之后,可由主页选择Analyze the networks,进行网络分析,主要分析项目如下图,涉及网络整体特征,模块性分析,网络可视化等等,部分分析还涉及不同的算法和参数的设置,分析结果可以下载进行保存。其中网络可视化,网站可以绘制简单的网络图,也可以将网络结果导出为tif格式 ,并利用Cytoscape等软件进行设置和处理,同时利用Cytoscape也可进行网络的基本分析。
Show Time
下图利用的OTU数据分析获得的网络,利用了可视化软件为Cytoscape,并结合MENA的模块分析部分的结果。祝大家学习快乐~~
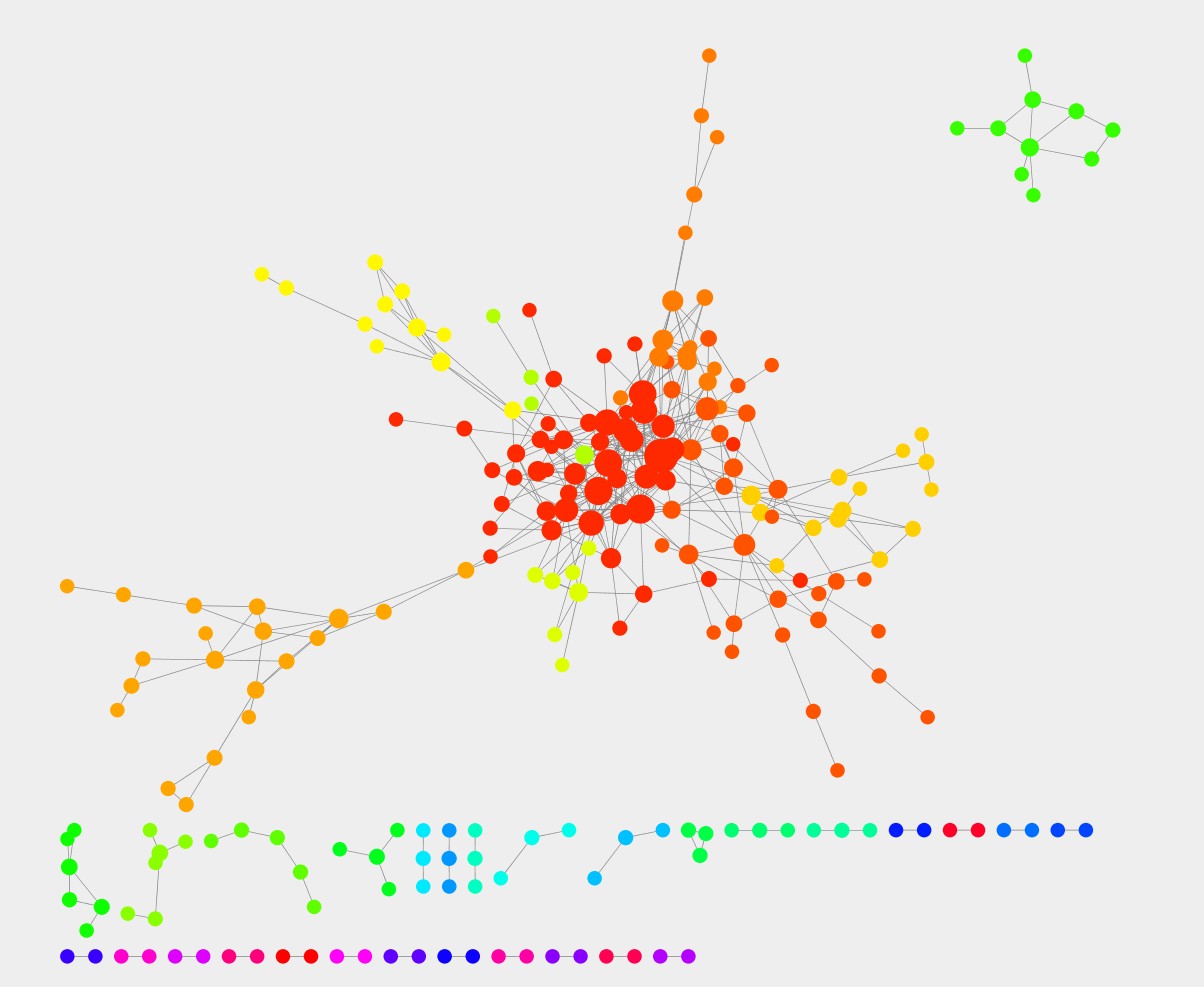
参考文献:
Deng, Y., Jiang, Y. H., Yang, Y., He, Z., Luo, F., & Zhou, J. (2012). Molecular ecological network analyses. Bmc Bioinformatics, 13(1), 113.
沈烽, 黄睿, 曾巾,等. 分子生态网络分析研究进展[J]. 环境科学与技术, 2016(s1):94-98.
相关推荐:微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制
课程推荐:微生物扩增子分析课程实操 微生物16S/ITS/18S分析原理及结果解读
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2018-04-22 21:19
- 阅读 ( 23353 )
- 分类:宏基因组
你可能感兴趣的文章
- MENA如何结合OTU共现性网络与环境因子 14355 浏览
