可变剪切在癌症中的研究
之前给大家分享过:下载TCGA数据库内转录组/LncRNA等数据,通过筛选差异基因进行生存分析,构建预后模型的文章思路,感兴趣的可以点下以下三个链接查看详细内容。
3. 再挖TCGA,发篇SCI
那么除了筛选差异基因进行此类分析外,还有其他好的思路吗?答案是肯定的;可变剪切是基因转录时,通过组合不同的外显子,形成不同转录本的过程,在生命的信息传递过程中发挥着重要的作用;今天就给大家分享一篇利用TCGA数据库癌症转录组数据可变剪切事件进行生存分析,构建预后模型分析的文章,目前可变剪切在癌症的研究里还不是很多,所以此类选材较为新颖,是一个很不错的选材思路!
此篇文章是2018年11月发表在医学2区期刊Frontiers in Oncology(IF=4.416)上,作者共下载了330个结肠癌转录组数据,深入分析了与结肠癌预后相关的关键可变剪切事件!

研究思路
从TCGA数据库下载转录组数据,采用SpliceSeq进行可变剪切分析
针对可变剪切事件,进行单因素的Cox生存分析
针对显著的单因素可变剪接事件,进行多因素的Cox生存分析
构建预后预测模型,并进行性能评估
剪切因子和剪切事件进行关联分析,筛选出重要的剪切因子
可变剪切分析
采用SplicSeq软件对TCGA中的转录组数据进行可变剪切分析,分别统计可变受体位点(AA)、可变供体位点(AD)、可变启动子(AP)、可变终止子(AT)、内含子保留(RI)、外显子跳跃(ES)、外显子互斥(ME)等7种可变剪切形式的可变剪切事件。其中外显子跳跃类型的可变剪切事件最多。统计图如下: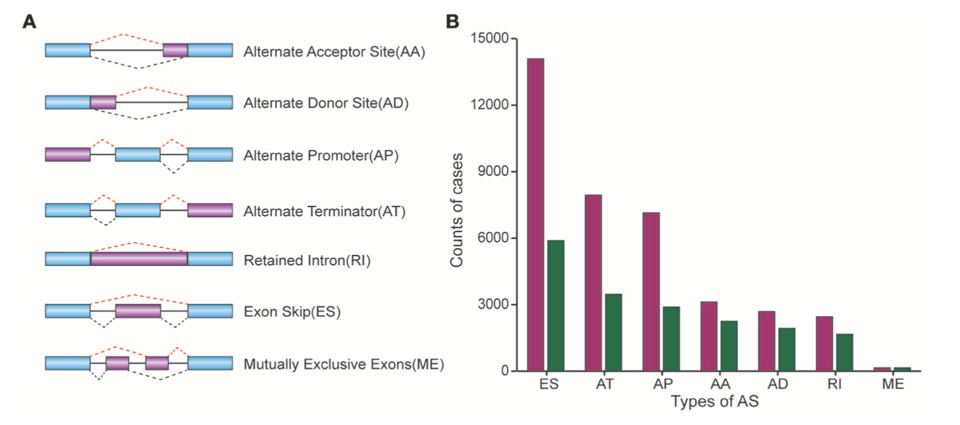
单因素生存分析
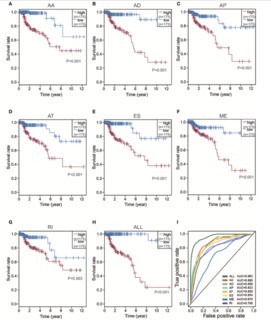
7种类型的可变剪切事件,分别进行单因素的Cox生存分析,筛选出显著相关的可变剪切事件,并将发生该可变剪切事件的基因筛选出来。下图为7类可变剪切事件对应的风险比率。

多因素生存分析
针对这7类可变剪切事件,分别基于该类中显著的可变剪切事件,构建预后预测模型,再基于中位数划分,进行KM生存分析。
为了评估预后模型的性能,采用ROC曲线进行比较分析;基于所有可变剪切事件构建的预测模型最优。
可变剪切显著相关基因的互作分析
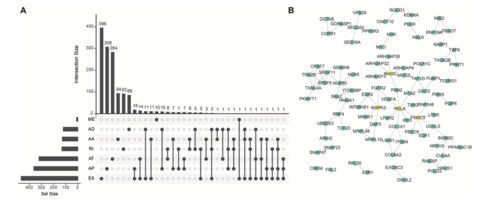
由于一个基因可能存在多种类型的可变剪切形式,分别统计含有不同类型可变剪切形式基因的数量。
基于这些生存显著相关可变剪接事件对应的基因,进行互作网络分析,一些关键基因与其他的基因存在相互作用,可能存在重要的作用。
 可变剪切因子分析
可变剪切因子分析
针对显著的可变剪切事件,与剪切因子的表达量进行相关性分析。一些剪切因子与高风险的剪切事件相关,如:HNRNPAB,一些则与低风险比例的剪切事件相关,如:HSPA7。

文章亮点
本文从可变剪切入手,进行生存分析,构建预后模型,其研究对象具有一定的新颖性。在可变剪切分析中,进一步研究其上游调控的剪切因子,下游的蛋白互作,让整个的分析内容比较深入。而且本文完全是生物信息的数据挖掘,具有很强的可模仿性,值得其他类癌症研究借鉴!
如果您想学习TCGA数据挖掘,请学习的我TCGA系列课程:
- 发表于 2019-01-29 17:09
- 阅读 ( 5483 )
- 分类:TCGA
