kaks和dnds是什么?
Ka/Ks分析是生物信息学中最常见的分析之一,它在研究核酸分子进化方面有重要应用,如果你还不知道的话——咳咳!注意听讲了!
01
“什么是Ka/Ks?”
在遗传学中,Ka/Ks表示的是两个蛋白编码基因的非同义替换率(Ka)和同义替换率(Ks)之间的比例。这个比例可以判断是否有选择压力作用于这个蛋白质编码基因。
让我们从最基本的道理开始讲起。如果你手头有两个不同物种的同一个基因的序列,比如人和小鼠的p53基因,然后把这两个基因的序列进行比对,你会发现这两段序列有差异(进化!)。再仔细观察,你会发现有些碱基的变化导致了编码氨基酸的变化(非同义替换),有些没有导致编码氨基酸的变化(同义替换)。这是由密码子的简并性造成的,因为3个碱基决定1个氨基酸,所以64种碱基组合决定20种氨基酸,会有冗余出现。一般情况下,第三个碱基变化会造成同义替换,而第一二个碱基的变化会造成非同义替换。
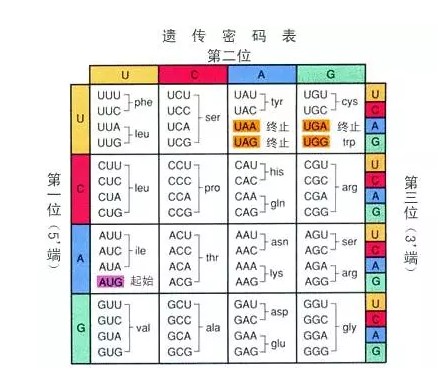
Ka和Ks的计算公式:
Ka=发生非同义替换的SNP数 或者 非同义替换位点数
Ks=发生同义替换的SNP数 或者 同义替换位点数
其中非同义替换位点数就是会造成氨基酸变化的位点数的总和,比如编码丝氨酸(ser)的第一二位碱基。而同义替换位点数就是不会造成氨基酸变化的位点数的总和,比如编码丝氨酸的第三位碱基。对于像第一个方框里面的苯丙氨酸(phe)和亮氨酸(leu)这种情况,第三位碱基的变化只是有可能造成氨基酸的变化,也就是两种不同的氨基酸各占了两个碱基的位点,则在计算非同义替换位点数和同义替换位点数时,各加上1/2,相当于两者平分了这个位点。另外,计算Ka/Ks时,不考虑start codon和stop codon。现在我们回到上面的人和小鼠的p53基因的例子。我们只需要数一下两个序列发生(非)同义替换的SNP位点数,再除以(非)同义替换位点数,就得到了Ka和Ks值,然后就可以计算Ka/Ks ratio。
上面的计算方法并没有考虑不同碱基之间发生替换的速率的不同,比如,嘌呤之间替换的概率(A=>G)要高于嘌呤替换为嘧啶的概率(A=>C或T),也就是说转换(transition,嘌呤变嘌呤,嘧啶变嘧啶)发生的概率要高于颠换(transversion,嘌呤变嘧啶,嘧啶变嘌呤)发生的概率。很多计算方法都会考虑到这些替换发生概率的不同。
另外,两个物种分化时间的长短也会影响到Ka/Ks的比值。比如有一个位点,原来是A,后来变成了T,再后来又变成了C,虽然发生了两次替换,但最后仅有一次替换被用于计算替换率。再比如有一个位点,原来是A,后来变成T,但同时与它相对应的另一个序列的位点,也发生了A到T的替换,那么我们也是无法用上面的方法来计算替换率。对于这种复杂的情况,我们可以用最大可能性算法来计算最可能的替换率,这里不再详述。
02
“那么Ka/Ks和进化有什么关系?”
细心的小伙伴可能已经看出来了,在上面的密码子表中,同义替换位点比较少。实际上一个基因中大概只有25%的位点是同义替换位点。假设这个基因没有受到自然选择压力,那么根据中性选择理论,非同义替换率和同义替换率应该是相同的。
一般来讲,因为非同义替换会造成氨基酸变化,可能会改变蛋白质的构象和功能,因此会造成适应性的变化,从而带来自然选择的优势或劣势(一般是劣势)。而同义替换没有改变蛋白质的组成,因此不受自然选择的影响(当然这里我们忽略密码子偏好性的影响),那么Ks就能反映进化过程的背景碱基替换率。Ka/Ks的比值就能说明这个基因是受到了何种选择。

长颈鹿的长脖子基因肯定是受到了正选择...........
一般情况下,在某个个体中偶然发生的一个碱基替换(突变),如果没有额外的好处或者坏处的话,慢慢地也就消失了。但是自然选择中会有很多巧合,某些突变就是很幸运地被保留了下来,并且被固定了(突变频率由极小变为100%)。一个这样的突变在一个二倍体种群中被固定的可能性为1/2N,其中N是种群大小。
对于一个没有受到自然选择压力的基因来说,我们可以计算得到这样的结果:Ka/Ks=1。但实际情况下,这个比值都是远小于1的:Ka/Ks<<1,因为一般非同义替换带来的都是有害的性状,只有极少数情况下会造成进化上的优势。
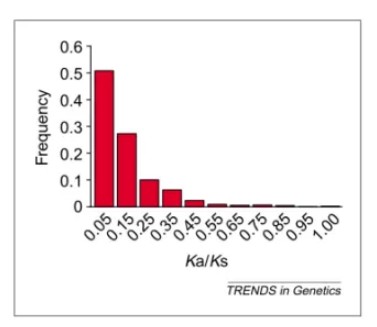
小鼠和大鼠的835个直系同源基因的Ka/Ks比值分布,横坐标的数值代表每个分类区间的中间值,如0.05代表0-0.1的区间。
于是,我们有了下面的分类:
Ka>>Ks或者Ka/Ks >> 1,基因受正选择(positive selection)
Ka=Ks或者Ka/Ks=1,基因中性进化(neutral evolution)
Ka<<Ks或者Ka/Ks << 1,基因受纯化选择(purify selection)
03
"Ka/Ks能用来做啥?"
人和黑猩猩的基因组差异只有约1.09%,染色体的数目及形态也几乎没有变化,但是仔细去看的话,会发现很多染色体片段的重排,也就是重新洗牌。2003年发表在Science的一篇研究发现,发生重排部位的基因,其Ka/Ks>1的比例,即受到正选择的比例要显著高于没有发生重排部位的基因。这说明染色体重排与基因功能的分化密切相关,与人和黑猩猩的分化密切相关。
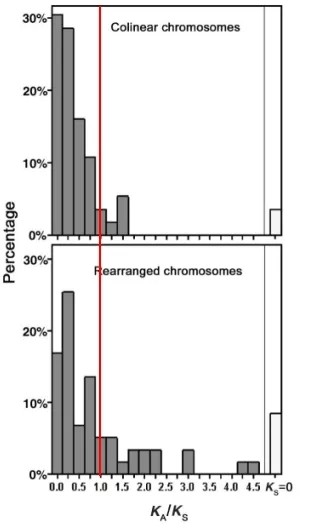
人和黑猩猩染色体非重排部位(上图)和重排部位(下图)基因的Ka/Ks分布,红色线条为Ka/Ks=1的分界线。重排部位受到正选择的基因比例更高。
当Ka/Ks>>1时,基因受到强烈正选择,这样的基因即为近期正在快速进化的基因,对于物种的进化有着非常重要的意义。我们可以根据Ka/Ks ratio筛选部分基因,然后做后期的功能研究,这种方法已经普遍的被应用到分子进化领域,已有多篇文章在Nature,Science等杂志发表。
除了查找快速进化基因,Ka/Ks还能检测基因的功能性,因为假基因(pseudogene)的Ka/Ks比值通常比功能基因更高(大家想想为什么?)。此外,Ks代表了进化过程的背景碱基替换率,因此可以用Ks来反推事件发生的时间,如全基因组多倍化的时间,这在探究物种起源方面有重要应用。在以后的高级篇中,我们会深入探讨这些问题。
Hurst L D. The Ka/Ks ratio: diagnosing the form of sequence evolution[J]. TRENDS in Genetics, 2002, 18(9): 486-487.
Navarro A, Barton N H. Chromosomal speciation and molecular divergence--accelerated evolution in rearranged chromosomes[J]. Science, 2003, 300(5617): 321-324.
北大教授顾红雅老师的公开课:生物演化http://www.chinesemooc.org/live/611219
更多生物信息课程:https://study.omicsclass.com/index
- 发表于 2019-01-30 23:58
- 阅读 ( 25567 )
- 分类:其他
你可能感兴趣的文章
- 基因家族分析课程更新-批量进行kaks计算 5215 浏览
- 计算Kaks时批量提取多对基因的序列 4996 浏览
- Ka/Ks与进化选择压力 10291 浏览
- 计算串联重复基因KaKs的时候,aln文件一定要通过脚本转换为axt文件么?这两种文件格式是否可以直接通过修改扩展名来实现? 4859 浏览
- Ka/Ks与进化选择压力 15542 浏览
