bioperl批量计算蛋白质分子量等电点pI
bioperl批量计算蛋白质分子量等电点pI
做基因家族分析,有时候我们想知道蛋白质的分子量和等电点信息还有基因的长度信息,这个时候我们一般会利用一个在线网站只要输入蛋白序列就可以:蛋白分子量分析:ExPASy (http://web.expasy.org/protparam/)来计算:
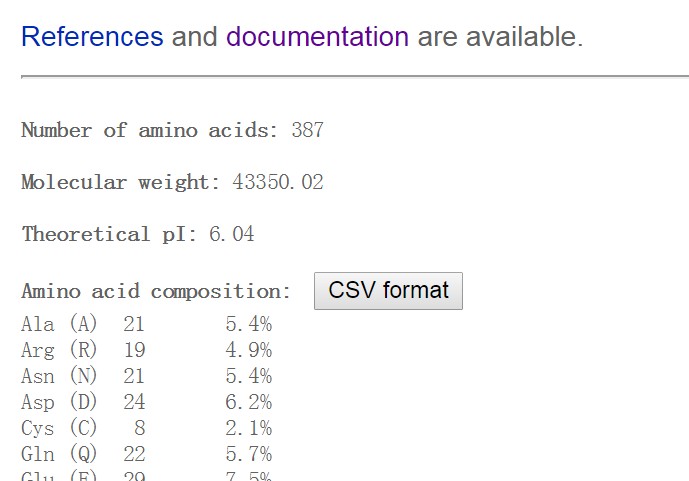
但是这个网站,只能一条一条的提交序列然后计算,如果后很多条序列要提交,其不支持批量提交,所有功能有限,这里我写了个perl脚本,利用bioperl包里面的方法,批量计算蛋白序列的长度,分子量,等电点信息,(有些值和上面网站的结果会有些许差异,可能计算的方法有些不同)如下:
die "perl $0 <in> <out>" unless(@ARGV==2);
use Bio::SeqIO;
use Bio::Seq;
use Bio::Tools::SeqStats;
use Bio::Tools::pICalculator;
use Data::Dumper;
#读入序列
my $in = Bio::SeqIO->new(
-file => "$ARGV[0]",
-format => 'Fasta'
);
open OUT,">$ARGV[1]" or die "$!";
print OUT "#ID\tlength\tMV(Da)\tpI\n";
my $calc = Bio::Tools::pICalculator->new(-places => 2,-pKset => 'EMBOSS');
#逐条读取序列
while ( my $seq = $in->next_seq() ) {
my ( $id, $sequence, $desc ) = ( $seq->id, $seq->seq, $seq->desc );
my $weight = Bio::Tools::SeqStats ->get_mol_wt($seq);
$calc->seq($seq);
my $iep = $calc->iep;
print OUT sprintf("%s\t%s\t%s\t%s\n",
$seq->id,
$seq->length,
"$weight->[0]",
$iep);
}
$in->close();
close(OUT);
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读、
- 发表于 2019-02-21 09:03
- 阅读 ( 7888 )
- 分类:perl
你可能感兴趣的文章
3 条评论
请先 登录 后评论
