如何计算蛋白序列的长度、分子量、等电点等信息
如何计算蛋白序列的长度、分子量、等电点等信息
做基因家族等分析时,有时候我们想知道蛋白质的分子量、等电点信息以及序列长度等信息。今天,小编教大家如何获取这些信息。
ExPASy ProtParam在线获取
ExPASy ProtParam 是一款在线蛋白质分析软件,它可以计算一个蛋白质序列的各种理化参数,例如氨基酸序列长度、等电点、分子量等等。其用法如下:
输入一条蛋白质序列:
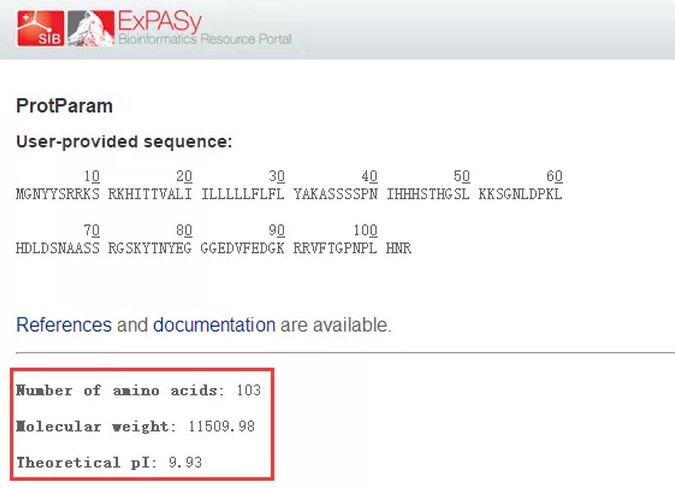
提交后,系统会计算蛋白质序列的各种理化参数并显示出来,而我们需要的信息如下图所示:
perl脚本批量计算
ProtParam 网站一次只能提交一条序列,如果我们的蛋白质序列较少还可以使用,但是序列多的话就不适用了。这就需要一种批量处理的方法,为此我们专门写了一个perl脚本,利用bioperl包里面的方法,批量计算蛋白序列的长度、分子量、等电点信息。
使用方法:
perl stat_protein_fa.pl pep.fa pep.stat.xls
pep.fa :是输入的蛋白质序列;
pep.stat.xls :为输出文件。
perl脚本代码如下:
#北京组学生物科技有限公司
#email: huangls@biomics.com.cn
die "perl $0 <in> <out>" unless(@ARGV==2);
use Bio::SeqIO;
use Bio::Seq;
use Bio::Tools::SeqStats;
use Bio::Tools::pICalculator;
use Data::Dumper;
#读入序列
my $in = Bio::SeqIO->new(
-file => "$ARGV[0]",
-format => 'Fasta'
);
open OUT,">$ARGV[1]" or die "$!";
print OUT "#ID\tlength\tMV(Da)\tpI\n";
my $calc = Bio::Tools::pICalculator->new(-places => 2,-pKset => 'EMBOSS');
#逐条读取序列
while ( my $seq = $in->next_seq() ) {
my ( $id, $sequence, $desc ) = ( $seq->id, $seq->seq, $seq->desc );
my $weight = Bio::Tools::SeqStats ->get_mol_wt($seq);
$calc->seq($seq);
my $iep = $calc->iep;
print OUT sprintf("%s\t%s\t%s\t%s\n",
$seq->id,
$seq->length,
"$weight->[0]",
$iep);
}
$in->close();
close(OUT);
最后祝您科研愉快!
此外,我们在网易云课堂上有各种教学视频,有兴趣可以了解一下:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘
6. 更多学习内容:linux、perl、R语言画图,更多免费课程请点击以下链接:
- 发表于 2019-03-15 16:36
- 阅读 ( 11729 )
- 分类:软件工具
你可能感兴趣的文章
- 基因组中的字符都代表什么 3068 浏览
相关问题
0 条评论
请先 登录 后评论
