6分SCI(医学类)-按这个思路很好发!
我们最近推出了一门重现6分医学文章分析内容的课程《TCGA-甲基化生存分析》,下面通过一篇6分SCI文章跟大家介绍一下该课程内容。
该文章于2018年11月发表在Clin Epigenetics(IF=6.0)上,题目是《A five DNA methylation signature act as a novel prognostic biomarker inpatients with ovarian serous cystadenocarcinoma》。
文章思路
首先看一下文章的分析思路: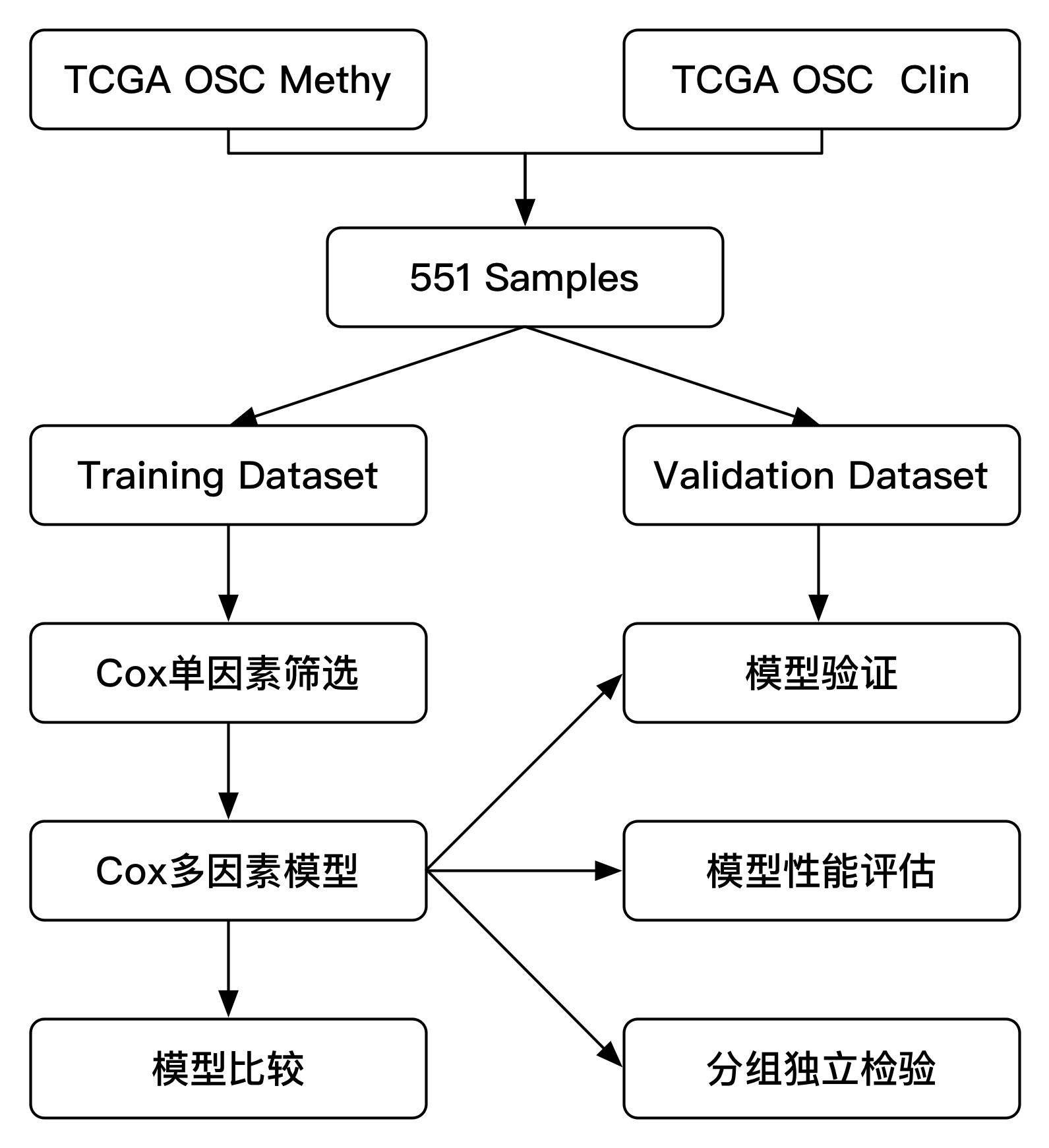
数据
551例卵巢癌患者的DNA甲基化数据和临床数据都来自于公开的癌症数据库TCGA。为了构建和检验预后预测模型,将551例数据随机划分为训练数据集(368例)和检验数据集(183例)。在训练数据集上构建生存分析模型,在检验数据集上检验模型。
数据集的临床信息统计如下: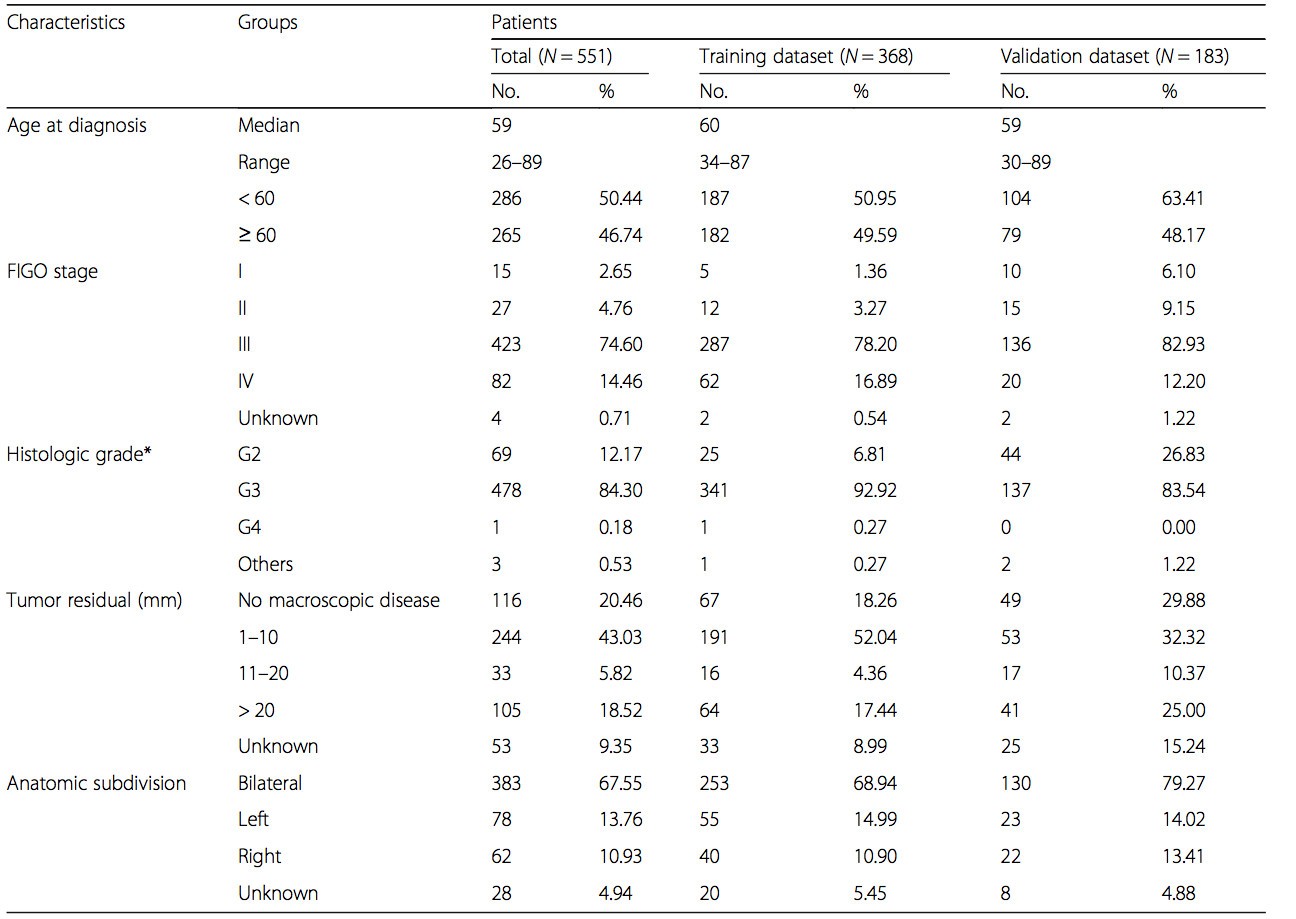
构建预后预测模型
在训练集上,以患者的DNA甲基化位点进行单因素Cox分析和多因素的Cox分析,最终构建了一个含有5个甲基化位点的预后预测模型。5个甲基化位点的模型信息如下: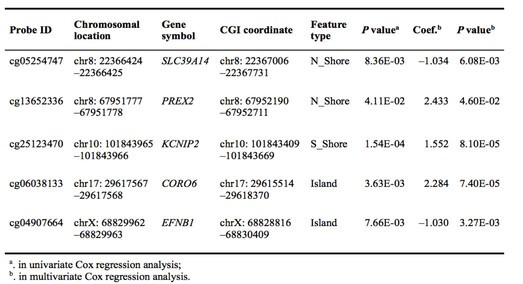
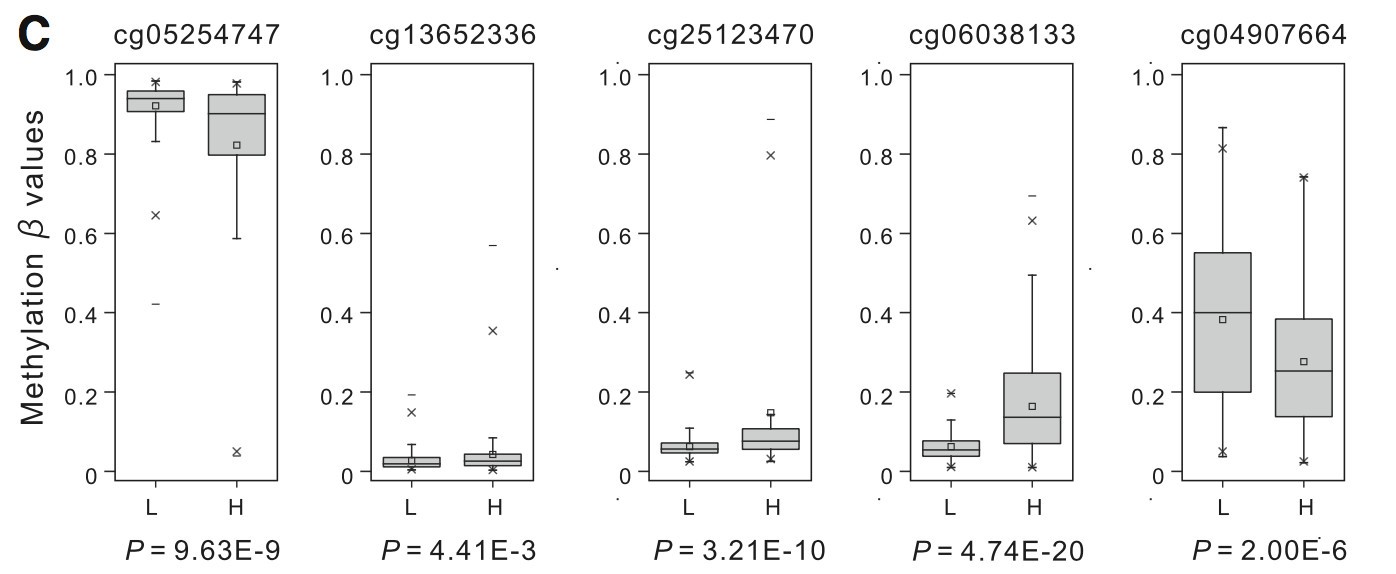
从模型中可见cg13652336, cg25123470和cg06038133为风险因子,而cg05254747 and cg04907664为保护因子。
模型性能评估
为了评估预后预测模型的性能,以预测模型在数据集上预测值的中位值为阀值将样本分成高风险和低风险两组,两组样本采用KM分析,统计两组样本是否存在生存时间的显著差异。从分析结果来看,分组样本的生存时间在训练数据集和验证数据集上都有较好的显著性差异。
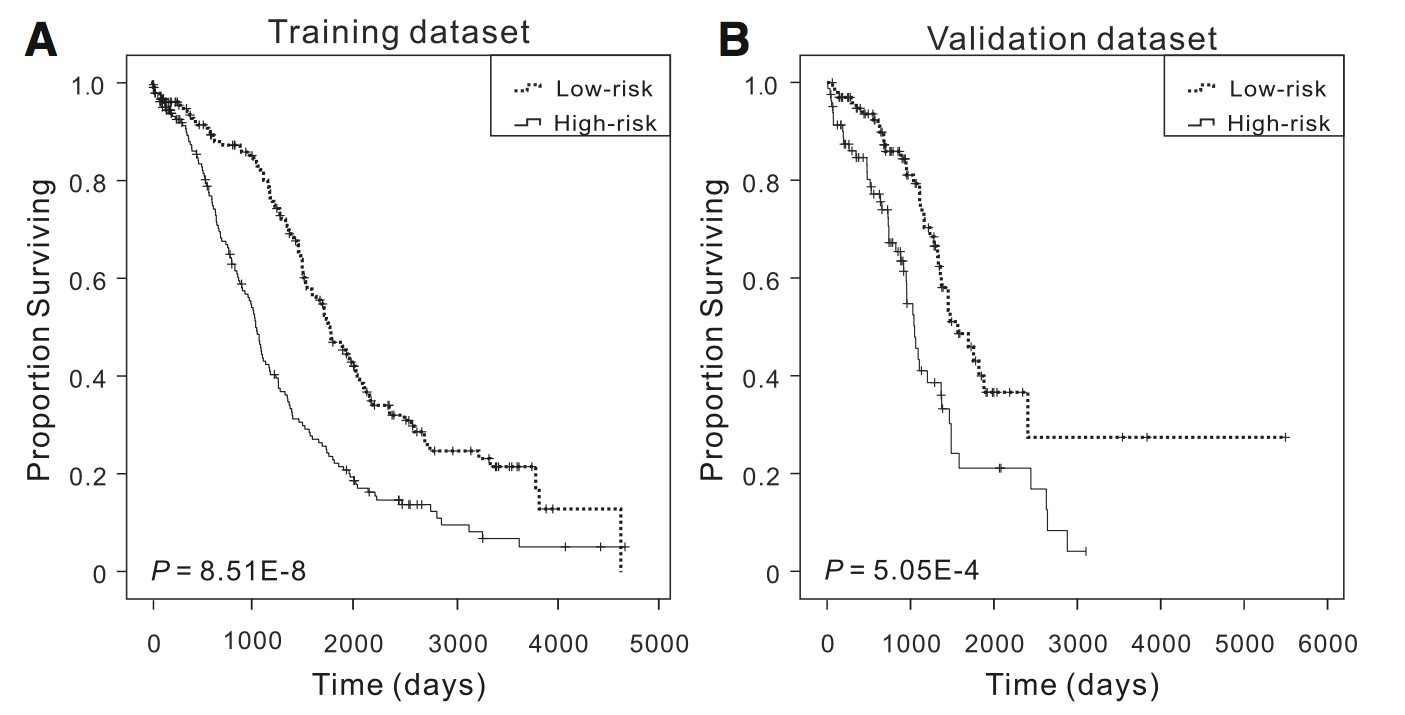
5个甲基化位点在高低风险组表达值(β值)的box分布如下:
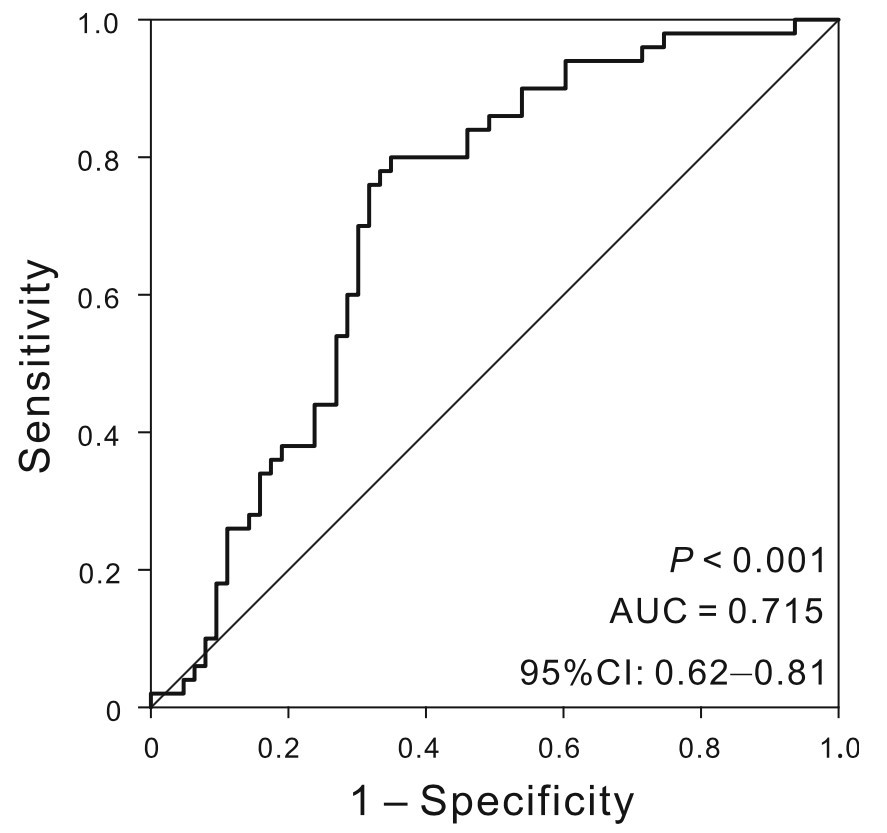
在验证集上,采用混淆矩阵对预后预测模型进行性能分析,采用ROC曲线评估其性能。模型的AUC值能够达到0.715,具有一定的临床应用价值。
分组分析
有研究发现,年龄是卵巢癌预后的一个重要因素,所以需要针对年龄进行一定的分层分析。依据年龄的分布范围,将样本分成3组,分别进行KM分析。从结果来看,组间差别不大,说明预后模型是独立于年龄的一套预后模型。
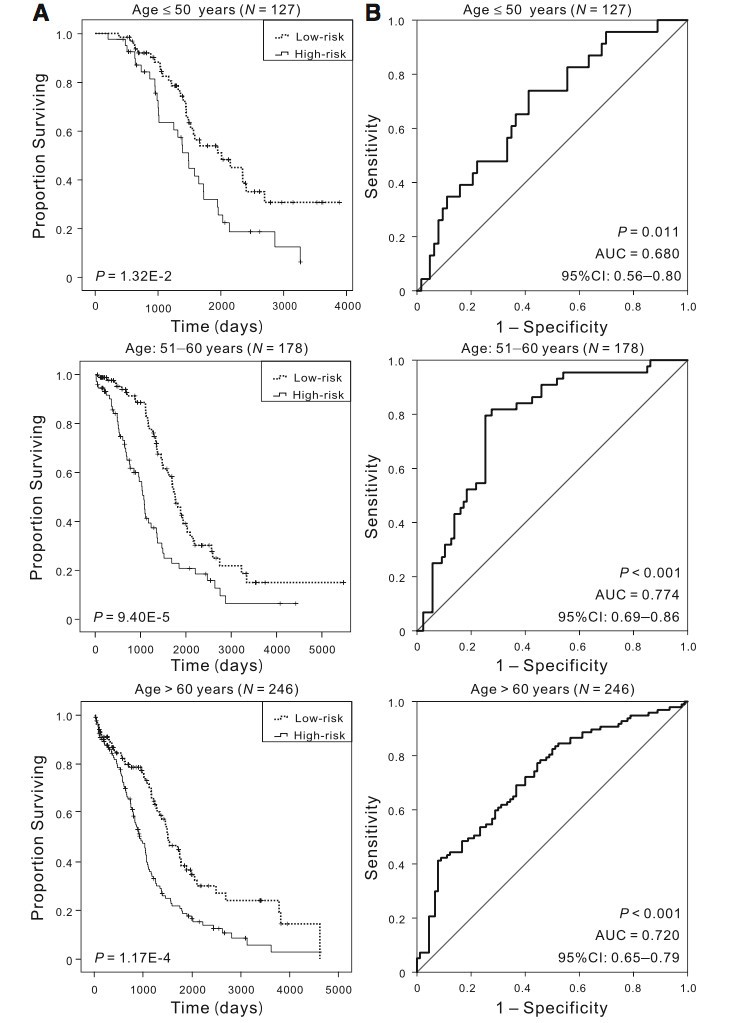
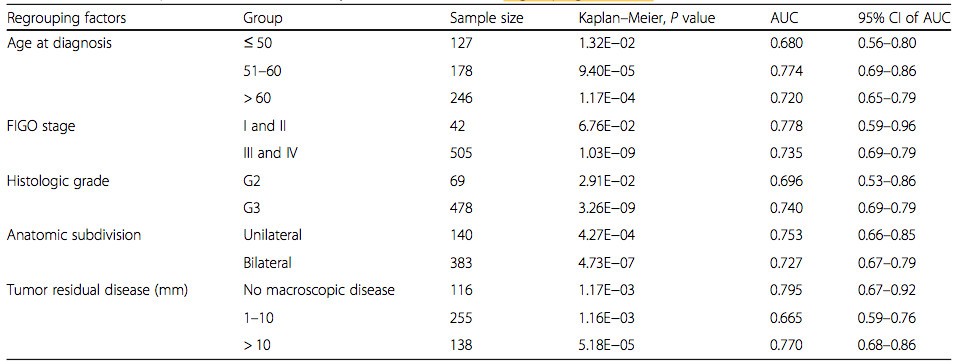
同理,针对FIGO stage,Histologic grade,Anatomic subdivision和Tumor residual disease进行分层分析。分析结果汇总如下:
模型比较
已有报道的卵巢癌预后预测模型比较多,将本文中5个甲基化位点构建的预后预测模型与这些已有模型进行比较, 发现本研究中的预后模型是最优的预后模型。采用ROC曲线展示模型间的预后性能,比较结果如下:
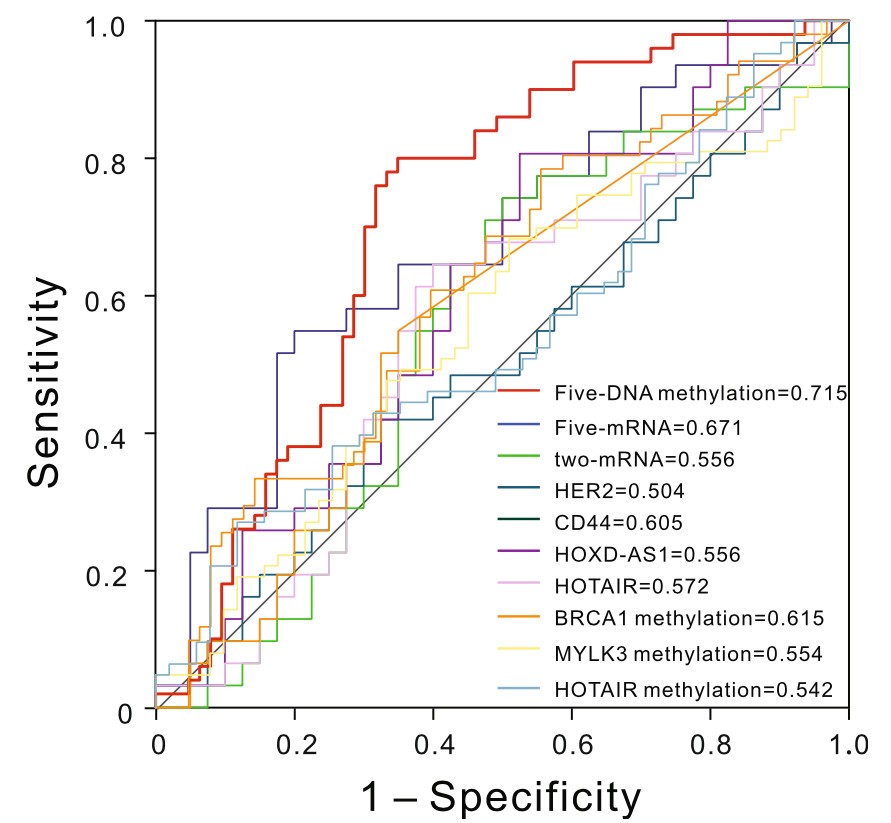
总结
本文以DNA甲基化位点为癌症预后的影响因子,采用生存分析的方法构建了生存预后模型。模型具有较好的预测性能,AUC值能达到0.715,说明该模型具有一定的临床应用价值;与以往的预后模型相比,该模型具有更高的AUC值,而且存在显著优势,是非常值得借鉴的!
为此,组学大讲堂精心录制《TCGA-甲基化生存分析》课程,手把手教您利用TCGA数据库中癌症甲基化数据进行生存分析,轻松发5-6分SCI论文,课程刚刚上架,还等什么,点击下面的链接,购买吧!
如果您想学习TCGA数据挖掘,请学习的我TCGA系列课程:
- 发表于 2019-03-19 08:14
- 阅读 ( 5288 )
- 分类:TCGA
你可能感兴趣的文章
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- TCGA下载 docker报错 0 回答
- TCGA差异分析如何根据临床信息分组? 1 回答
- TCGA用R数据下载错误 1 回答
- 老师,想请教一下TCGA基因表达数据的问题,我从xena.ucsc网页上下载了基因表达数据TCGA-CESC.htseq_counts.tsv;然后发现该数据中只有Ensembl格式的基因ID ,没有SYMBOL格式的。所以接下来进行基因ID格式转换,却发现同一个SYMBOL ID对应的多个Ensembl格式的ID,想问下老师,这种情况该怎么处理?同一个SYMBOL ID所对应的多个Ensembl格式ID的基因表达数据应该留下哪一个? 1 回答
- TCGA的RNA-seq count annotation过滤,有些不知道是否需要删除 0 回答
