基于多数据集分析ANLN在宫颈癌所起到的功能
宫颈癌是造成女性死亡的主要癌症之一,这种妇科肿瘤发病也越来越年轻化。随着时代的发展和进步,我们能够从各大数据库网站找到开放的相关研究数据,包括GEO数据库、TCGA数据库等等。而基于现有的数据分析鉴定宫颈癌相关biomarker也是进行相关研究的一个方向,也有助于其他的科研人员开展更深层次的探索。这里就给大家介绍一篇文献:基于GEO数据库相关数据进行宫颈癌biomarker鉴定。
数据来源
从GEO数据库下载GSE29570和GSE89657两个相同芯片型号(GPL6244: Affymetrix Human Gene 1.0 ST Array)的数据,以其中49个癌症组织样和20个正常人体样本进行分析。
以GSE52903、GSE7410两组GEO数据和oncomine数据库中的Pyeon cervical cancer(20 cancer cases / 8 normal cases)数据进行后期的数据验证。
数据分析
1.数据处理与差异分析
下载GSE29570和GSE89657原始数据,基于RMA算法进行数据预处理,并去除批次效应,从而进行差异基因的筛选,差异基因的筛选分析基于limma包完成,最终以p<0.05 & |logFC| ≥1.453,获取了324个差异基因(123 up-regulated / 201 down-reguated,见下图A),其中,差异变化倍率Top100的基因有明显的样品差异(见下图B)。

2.差异基因富集分析
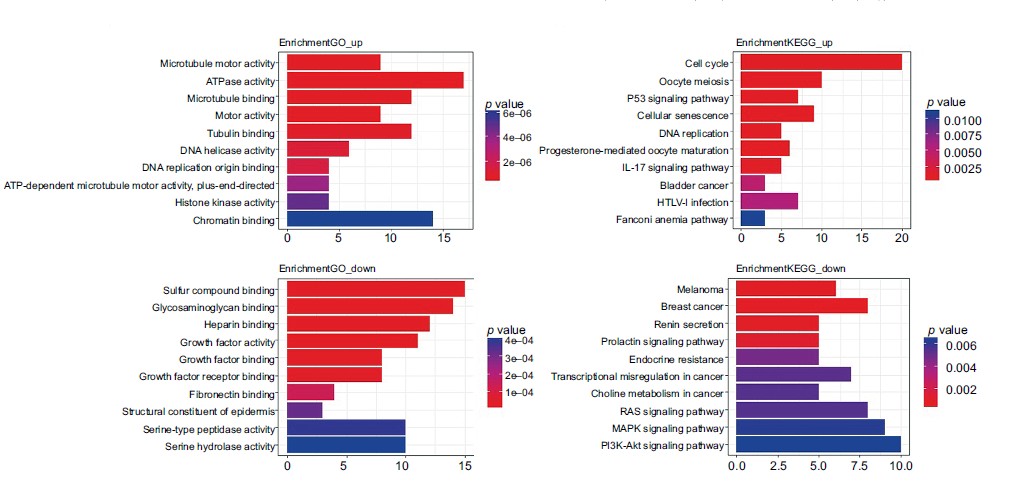
针对差异基因功能进行富集,从而判断这些差异基因在癌症发病等过程中所起到的主要作用,GO富集结果显示,上调基因主要涉及微管运动活性、ATP酶活性和微管结合,而下调基因则与硫化合物结合、糖胺聚糖结合和肝素结合相关;KEGG富集结果表明,上调基因与细胞周期、卵母细胞减数分裂和p53信号通路相关,而下调基因作用于黑色素瘤、乳腺癌和肾素分泌(见下图)。
3.差异基因蛋白互作分析
为探索差异基因之间的蛋白互作关系,利用string数据库进行分析,最终基于324个差异基因获取了一个蛋白质互作网络(305 nodes/4962 edges,见下图A),一共鉴定了305个蛋白之间具有4962对互作关系。
之后借助Cytoscape软件中的MCODE插件,以k-core=2为阈值,筛选出其中关系最紧密的子网络,涉及了65个基因,1780对互作关系(下图B),并根据mcode_score筛选出了Top20(最主要)的基因:CEP55/ TYMS/ KIF15/ TTK/ PTTG1/ CDKN3/ MKI67/ KIF23/ CCNB1/ CDK1/ KIFC1/ KPNA2/ CDC6 / MCM4/ MCM2/ SHCBP1/ FOXM1/ NUF2/ RRM2/ ANLN。而现有的研究表明,其中的几个基因,包括FOXM1、MKI67等都和相关癌症有关,是有效的预后因子。
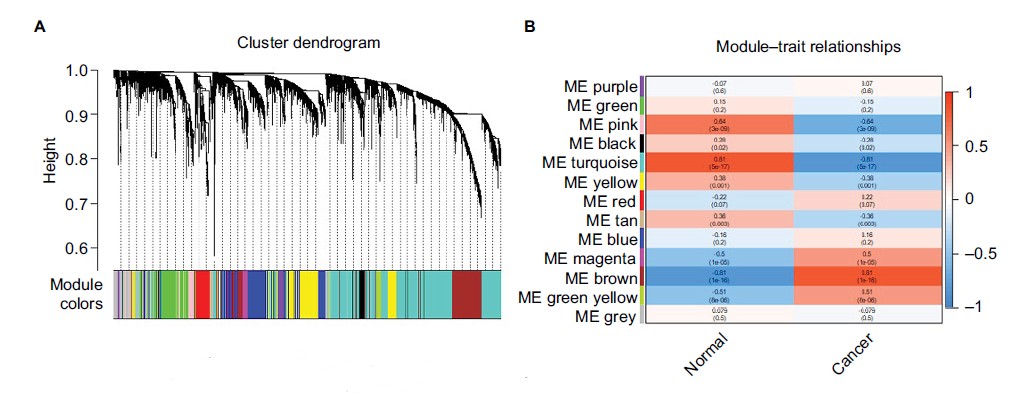
4.加权基因共表达网络分析
以预处理和去除批次效应的数据参与WGCNA分析,power值选定6,最终获得12个有效模块(加上grey为13,见下图A)。其中Brown和Turquoise与临床性状明显相关(见下图B)。
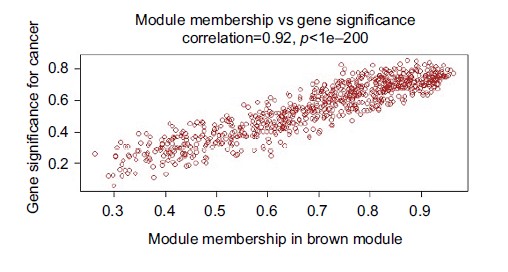
其中Brown模块内GS与MM分析结果显示,该模块内的基因与癌症发病显著相关(见下图),并基于相关性阈值高于0.9 筛选出了86个基因作为hub 基因进行后期分析。
5.差异基因联合hub gene挖掘biomarker
基于此前筛选的324个差异基因和86个Brown模块的hub gene,挖掘共有的基因,最终发现一共有61个基因既属于差异基因又属于hub gene(见下图A)。针对这些基因利用UALCAN网站在线进行生存分析,最终发现ANLN基因可能是良好的预后因子(下图B)。
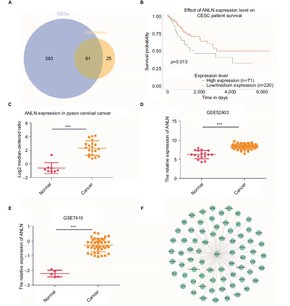
此外进一步于GSE52903、GSE7410两组GEO数据和oncomine数据验证该基因是否出现明显差异,结果与预期吻合:癌症样本中该基因表达水平明显高于正常样本(见上图C、D、E)。
研究人员进一步从此前分析的65个基因对应的蛋白质互作网络中,提取出ANLN相关的互作关系,构建成上图F中的网络,该网络中存在着MKI67、FOXM1与ANLN之间的互作关系。
结论
作者基于现有数据进行分析发现:ANLN基因可能是宫颈癌的一个重要的biomarker,对与癌症的诊断和后期治疗可能具有重要作用,并且该基因和前人已经研究的MKI67、FOXM1等具有明显的互作关系,而这些基因也与相关癌症有关。
参考文献:
Leilei X , Xiaoling S , Jizi S , et al. ANLN functions as a key candidate gene in cervical cancer as determined by integrated bioinformatic analysis[J]. Cancer Management and Research, 2018, Volume 10:663-670.
相关课程:
TCGA-基因差异表达分析、 TCGA-生存分析 、 GEO芯片数据挖掘、GEO芯片数据标准化、WGCNA-加权基因共表达网络分析
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、perl入门到精通、perl语言高级、R语言入门、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、GEO芯片数据标准化、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读
- 发表于 2019-03-29 11:57
- 阅读 ( 7752 )
- 分类:WGCNA
你可能感兴趣的文章
- 用AI写代码批量整理GEO数据库中的单细胞数据 374 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 891 浏览
- R语言中转换成dummy dummy-variables 虚拟变量 1826 浏览
- 细胞焦亡、铁死亡及铜死亡等基因集 3912 浏览
- 记录一个数据下载:GSE12417 1388 浏览
- 生信挖掘GEO+TCGA 文献速览 1907 浏览
相关问题
- 请教一下TCGA的蛋白数据怎么理解,它是有正负值的,这个正负值代表什么意思,为什么蛋白表达会有负值?我只想给它分成高表达,低表达和表达缺失组应该怎么处理呀? 0 回答
- wgCNA一个模块中找hub基因 我把cytoScapeinput-edges文件输入cytoscape中 用cytohubba计算 但是每次出来的结果都不一样 试了很多遍也不行 求解答 谢谢! 0 回答
- 我得到转录组分析的wgcna数据需通过cytoscape筛选关键模块的hub基因,边的权重应该设定多少来筛好 1 回答
- WGCNA中过滤低表达量(0.5)时datExpr0=datExpr0[1:n,datExpr0[n+1,] > meanFPKM]不成功,且变量从上步30769变成了0. ? 2 回答
- 跑WGCNA,用的独享服务器,报错 1 回答
- 我想咨询一下wgcna问题 0 回答
