R语言绘图-绘制漂亮的数学公式或者拉丁字母等在图当中
R语言里面的公式表示方法可以使用 ?plotmath 进行查看,主要是一些数学公式和符号的表示方法。下面有一段代码是绘制公式的代码,并把公式和文字组合在一起:
y <- function(x) log(x) + sqrt(x) + x^(1/3)
plot(y, 1, 1000, main = expression(paste("This is a formula ",y == log(x) + sqrt(x) + sqrt(x, 3))),
lwd = 3,
col = "blue")
 要想组合出自己想要的复杂公式,那么就得知道,公式表示方法和显示结果的对应关系。这里有段代码可以产生对应关系,需要输入文件:math.txt
要想组合出自己想要的复杂公式,那么就得知道,公式表示方法和显示结果的对应关系。这里有段代码可以产生对应关系,需要输入文件:math.txt
ex <- parse("math.txt") labs <- readLines("math.txt") n <- length(ex) par(mar = rep(0.1, 4), cex = 0.8) plot.new() plot.window(c(0, 8), c(0, n/4)) y <- seq(n/4, by = -1, length = n/4) x <- seq(0.1, by = 2, length = 4) xy <- expand.grid(x, y) text(xy, labs, adj = c(0, 0.5)) xy <- expand.grid(x + 1.3, y) text(xy, ex, adj = c(0, 0.5), col = "blue") box(lwd = 2) abline(v = seq(1.3, by = 2, length = 4), lty = 3) abline(v = seq(2, by = 2, length = 3), lwd = 1.5)

上图中奇数列是字符串(表达式),偶数列(蓝色)是Tex格式化的图形。除了上表列出的规则外还有一些拉丁文和希腊文符号,可以在表达式中用 symbol 函数或名称(如alpha)等表示,可参考这里:R表达式。如果函数名(包括运算符)有对应的Tex格式化规则,函数名和参数都按规则进行图形绘制;如果没有,就当成是R语言普通函数:
示例代码:
ex <- expression(sqrt(x), x + y, x^2, x %in% A, x <= y, mean(x, y, z), x | y, x & y) n <- length(ex) par(mar = rep(0.1, 4), cex = 1.5) col <- c("red", "blue") plot.new() plot.window(c(0, n), c(0, 1)) for (i in 1:n) text(i - 0.5, 0.5, ex[i], col = col[i%%2 + 1])
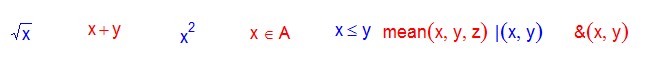 上面例子中前5种运算函数都是有对应数学符号的,所以它出的图(符号和顺序)与数学习惯一致,后三种运算函数没有对应数学符号,所以用普通函数方式(函数名在前,参数在括号内用逗号分隔)出图。
上面例子中前5种运算函数都是有对应数学符号的,所以它出的图(符号和顺序)与数学习惯一致,后三种运算函数没有对应数学符号,所以用普通函数方式(函数名在前,参数在括号内用逗号分隔)出图。
R语言技能推荐学习:R语言画图、R语言快速入门与提高
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.组学大讲堂全部生物生信数据挖掘课程可点击:组学大讲堂视频课程
- 发表于 2019-04-08 18:06
- 阅读 ( 9682 )
- 分类:R
你可能感兴趣的文章
- 已知集合交集数量绘制韦恩图 273 浏览
- 绘制Upset 图 1420 浏览
- R公式中如何加入变量 697 浏览
- R语言基础入门—数据结构(2) 700 浏览
- R语言基础入门—数据结构(1) 676 浏览
- R语言基础入门—R语言概述与安装! 858 浏览
