GEO数据挖掘生物信息文章解读(直肠癌)
GEO数据库全称GENE EXPRESSION OMNIBUS,是由美国国立生物技术信息中心NCBI创建并维护的基因表达数据库。它创建于2000年,收录了世界各国研究机构提交的基因表达数据(主要包括芯片表达数据,也包含一些高通量测序表达数据)。这里含有海量的公开的免费的数据,我们可以利用这些数据借助生物信息学工具再次挖掘这些数据,发表SCI论文。下面介绍一篇2019年发表的GEO数据挖掘套路文章。
文献

文章今年(2019)发表在:Gene 上IF=2.6分左右。该文章发表的杂志影响因子虽然不高,分析的内容呢也会少一些,但是对于生信数据挖掘刚刚入门的生物汪来说却是一个非常好的入门级别的文章。
文章的目的是通过生物信息分析的方法,挖掘GEO和TCGA数据库当中的公开数据,从而发现与直肠癌预后相关的biomarker,为直肠癌的诊断和治疗提供分子诊断依据。
1.GEO和TCGA数据差异分析
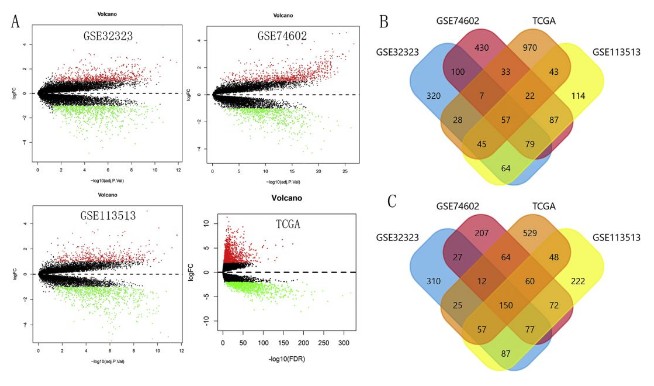
作者在GEO数据库当中找到三个直肠癌相关的数据GSE32323, GSE74602, and GSE113513,分别做差异分析。然后,又下载TCGA当中的直肠癌相关的转录组数据做差异分析,差异分析结果绘制火山图(下图 A)。对于4组数据当中的差异基因按照上调基因(下图B)和下调基因(下图C)分别做韦恩图,最终得到共有的上调基因57个和下调基因150个。
2.功能注释富集分析
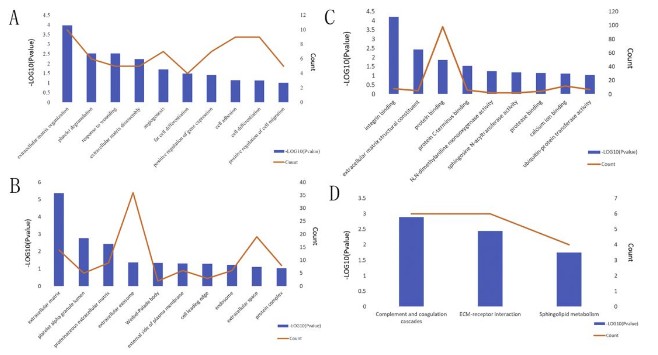
通过DAVID数据库对差异基因进行功能注释和富集分析。GO富集分析发现,其中注释到生物过程(BP)大类的差异基因主要富集的功能term包括:extracellular matrix (ECM) organization, platelet degranulation, response to wounding and extracellular matrix disassembly(下图A)等等。KEGG通路分析发现差异基因主要集中在以下通路:Complement and coagulation cascades, ECM-receptor interaction and Sphingolipid metabolism(下图D)。这些通路或者富集的功能很多都与直肠癌的发生发展相关。
3.蛋白互作网络分析发现hub gene
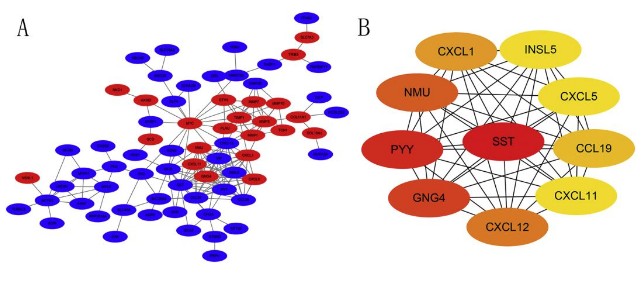
将以上得到的共有的差异基因上传到STRING蛋白互作数据库,利用cytoscape软件当中的cytoHubba插件寻找 hub基因,最后取其中连通性最高的top10个基因构建网络,分别是:CCL19, CXCL1, CXCL5, CXCL11, CXCL12, GNG4, INSL5, NMU, PYY, SST(下图)。
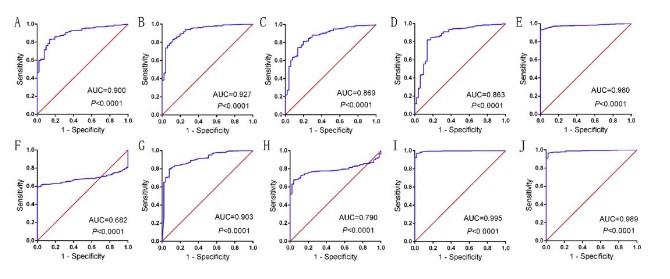
分析上面hub基因对直肠癌的诊断作用:再利用TCGA中的临床数据,作者通过ROC分析发现这些hub基因对直肠癌的诊断具有很高的准确性,10个基因(CCL19, CXCL1,CXCL5, CXCL11, CXCL12, GNG4, INSL5, NMU, PYY, and SST)的AUC值分别是:0.900, 0.927, 0.869, 0.863, 0.980, 0.682, 0.903, 0.790, 0.995, and 0.989。
4.生存分析及预后分析
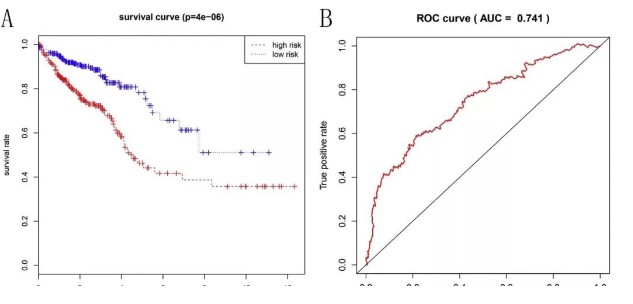
通过单因素Cox回归分析,作者共发现了 32个基因与直肠癌的预后相关,再通过多因素Cox分析,最终确定9个基因与直肠癌的预后强烈相关,可以作为直肠癌预后的标志分子并建立预后模型 (-0.1068×SLC4A4) +(-0.2564×NFE2L3)+(0.2082×GLDN) +(0.0834×PCOLCE2) +(0.3424×TIMP1)+(0.1149×CCL28) +(-0.0991×SCGB2A1) +(-0.1080×AXIN2) +(-0.1516×MMP1)。Kaplan-Meier曲线显示,模型预测的低风险组与高风险组相比,高风险组患者的死亡风险明显高于低风险组(下图A),ROC曲线对于5年生存预测风险评分的AUC值为0.741(下图B),模型对直肠癌的预后效果很好。
5.方法总结
文章中使用的数据来源有两个,一个是GEO当中的直肠癌相关的芯片数据,第二个是TCGA数据库当中直肠癌的转录组测序数据。两个数据集分别做差异分析,然后取交集得到最终的差异基因集,将这些差异基因提交到DAVID数据库做差异基因的功能富集分析,发现差异基因所在通路或者功能与直肠癌相关。通过蛋白互作网络分析找到hub基因,与直肠癌的诊断相关,最后通过Cox回归分析找到与直肠癌预后相关的基因,并建立预后模型,并检验该模型具有很好的预后准确性。
文章套路很简单,没有做任何的实验,分析内容也不多,对于刚刚入门的生信小白来说是很好的练手范例。数据挖掘发文是投入产出比最高的发文思路了,可以说只要你会生物信息投入的成本就只有时间了。
医学相关数据挖掘课程,不用做实验也能发文章,学习链接:TCGA-差异基因分析、GEO芯片数据挖掘、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
延申阅读
GEO芯片数据下载 |GEO数据与WGCNA--挖掘胶质瘤共表达网络的关键模块与通路|GEO和TCGA套路文章解说 | 基因芯片表达差异分析 | GSEA法基因功能富集分析原理详解! | 挖别人的数据,发自己的文章 | TCGA-数据挖掘 | 转录因子研究方法!
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课,学习链接:linux系统使用、perl入门到精通、perl语言高级、R语言画图
7. 医学相关数据挖掘课程,不用做实验也能发文章,学习链接:TCGA-差异基因分析、GEO芯片数据挖掘、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他课程链接:二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读。
- 发表于 2019-07-26 14:52
- 阅读 ( 5730 )
- 分类:GEO
你可能感兴趣的文章
- 用AI写代码批量整理GEO数据库中的单细胞数据 377 浏览
- 单细胞转录组数据挖掘流程记录-头颈癌(HNSCC)(GSE181919) 898 浏览
- 细胞焦亡、铁死亡及铜死亡等基因集 3942 浏览
- 记录一个数据下载:GSE12417 1397 浏览
- 生信挖掘GEO+TCGA 文献速览 1908 浏览
- IF=3.47|非肿瘤-眼部老年黄斑变性ceRNA文章思路 2627 浏览
相关问题
- 代码报错 2 回答
- 老师您好,我这个是双通道芯片,我把read.maimages的所有source都试了一遍,还是不能把原始文件读进来 1 回答
- 老师您好,我在进行GEO分析时,打开分组文件group_file时出现报错,请问我该如何解决? 1 回答
- 请问老师,GEO数据分析时,运行gset = getGEO(GSE, GSEMatrix =TRUE, AnnotGPL=FALSE,destdir=workdir)时,遇到如图中报错,该如何处理呢? 1 回答
- Error in (function (classes, fdef, mtable) : unable to find an inherited method for function ‘bg.correct’ for signature ‘"HTAFeatureSet", "character"’ 0 回答
- 安捷伦双色通道 “RawData=read.maimages(files=files,source="genepix") “报错” 2 回答
