R包venn绘制韦恩图
venn包绘制的维恩图
示例代码:
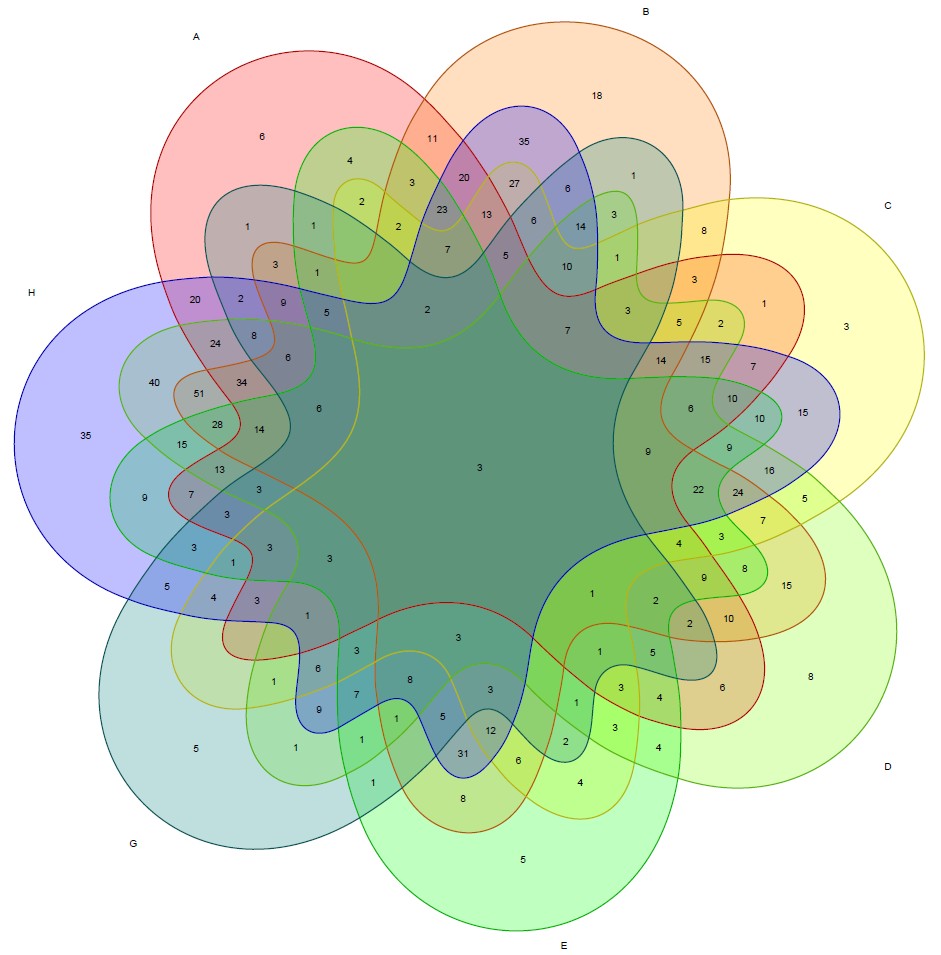
library(venn)
A <- sample(1:1000, 400, replace = FALSE);
B <- sample(1:1000, 600, replace = FALSE);
C <- sample(1:1000, 350, replace = FALSE);
D <- sample(1:1000, 550, replace = FALSE);
E <- sample(1:1000, 375, replace = FALSE);
G <- sample(1:1000, 200, replace = FALSE);
H <- sample(1:1000, 777, replace = FALSE);
dataForVennDiagram <- list(A=A, B=B, C=C, D=D, E=E, G=G,H=H)
#vennDiagramColors <- c('#EA4335', '#FBBC05', '#34A853', '#4285F4', 'orchid3')
vennDiagramColors <- c("dodgerblue", "goldenrod1", "darkorange1", "seagreen3", "orchid3", 'cyan',"red","blue","yellow") # TODO: cyan is not good enough
vennFilename <- 'report/Venn_Diagram_6Sets.png'
venn(dataForVennDiagram, zcolor = "style", opacity = 0.25, cexil = 0.5, cexsn = 0.5, ellipse = FALSE)
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.全部课程可点击:组学大讲堂视频课程
- 发表于 2019-08-19 11:21
- 阅读 ( 5160 )
- 分类:R
你可能感兴趣的文章
- 已知集合交集数量绘制韦恩图 273 浏览
- 绘制venn图,2-7维 1202 浏览
- 使用R包(VennDiagram)进行韦恩图绘制时相关参数调整 3541 浏览
- UpSetR包绘制多维的venn图 10203 浏览
- 3分钟学会画venn图(维恩图、韦恩图) 21485 浏览
2 条评论
请先 登录 后评论
