小麦MADS-box基因家族文章 new phytologist(7分)
小编一直在强调,基因家族分析是一个研究思路,同时也可以成一篇文章!不过大部分基因家族套路式文章影响因子在2-3分左右,3分以上需要添加实验等内容;不过,现在有一篇小麦MADS-box家族文章却发表在new phytologist(7分)上,着实让广大家族基因研究者愤懑不平好半天!我的家族分析发2分,他的发7分!就凭他是老外啊?真TM的!那么,这篇论文有什么不一样的地方呢?下面我们一起看看文章详细内容吧!

MADS-box基因家族介绍
MADS-box基因家族基因是一类广泛存在于植物中一种转录因子,高等植物中MADS基因由4个保守程度不一的区域组成, 分别是MADS区、I(intervening)区、K (keratin-like)区和C末端(M-I-K-C domain structure)。根据系统进化可以把MADS-box分为两类: I型和II型。大多数植物MADS基因, 包括所有功能特异的真核生物MADS基因和动物MEF2-like序列命名为II型;一些与动物SRF-like基因更相似的拟南芥MADS基因序列被命名为I型;K结构域只存在于II型植物MADS基因中。植物界中I型和II型基因的区别主要是MADS结构域不同,I型MADS结构域主要是MADS SRT型, II型MADS结构域主要是MADSMEF2型, II型的MADS域比I型的MADS域更保守。
小麦中MADS-box基因家族鉴定及进化树分析
MADS-box通常有两个保守的结构域:MADS和K结构域所对应的pfam数据库中的号分别为:PF00319 and PF01486,作者利用pfam号在小麦基因组中查找MADS-box家族基因,一共鉴定出201个 M-I-K-C 家族成员。家族成员根据进化树分成15个亚类,同时也添加了水稻和拟南芥当中的MADS-box家族基因(下图黑色基因)。
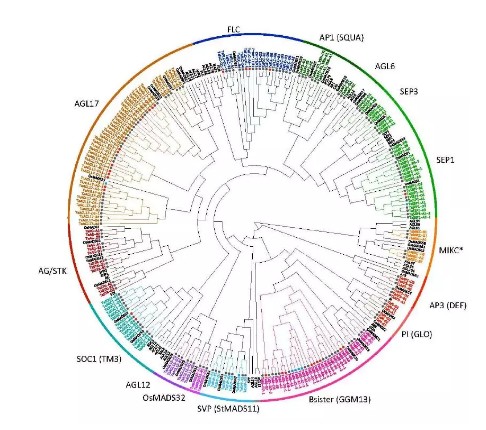
麦MADS-box比较基因组分析基因组分布
MADS-box基因家族成员在拟南芥和水稻中的成员数量分别是43和45个,拟南芥和水稻的基因组大小相差很大但是家族成员数量相差不大,说明MADS-box家族成员数量还是很保守的。而在小麦中却有201个,在开花类植物中是最多的。作者推测,小麦基因组中有这么多个MADS-box基因可能是由于普通小麦为6倍体(三套染色体体组)基因组的原因,导致家族成员之间在不同的染色体组之间有同源基因,所以理论上应该是水稻和拟南芥基因组MADS-box基因家族成员的3倍,因此作者做了小麦中15个亚类家族成员与拟南芥和水稻中的家族成员的分类比值(a-d),发现有些类型基因就是3:1的关系。进而分析小麦中MADS-box家族是如何加倍复制形成的。
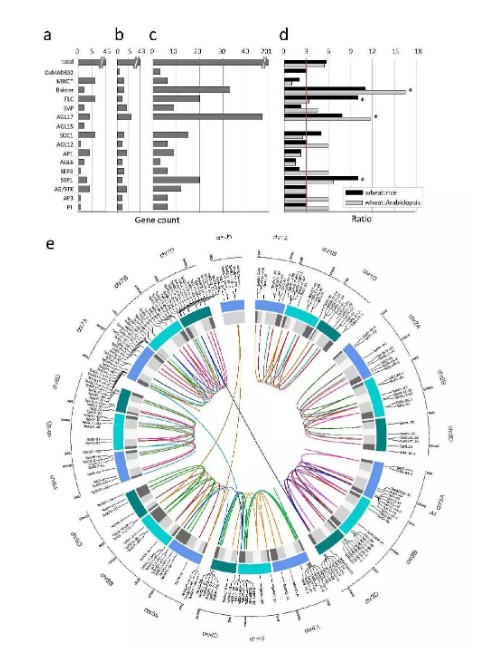
小麦中不同染色体组中的家族成员的同源分析
作者通过比较分析小麦不同染色体组(ABD)中家族成员之间的同源性发现,大多数的家族成员在三个染色体组中具有同源基因(1:1:1 )。也有些家族成员在某个染色体组中缺失,或者是孤儿基因。
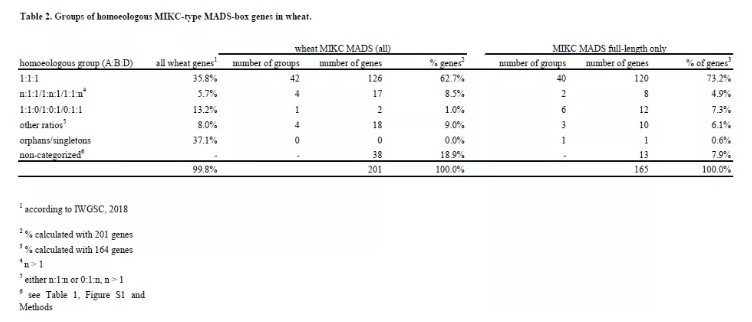
麦中MADS-box基因的染色体分布
将小麦MADS-box基因在染色体的分布区域进行统计,也就是将所有的染色体远端(R1,R3)和近端(R2B,C),大部分的家族成员分布在远端区域(65%)。
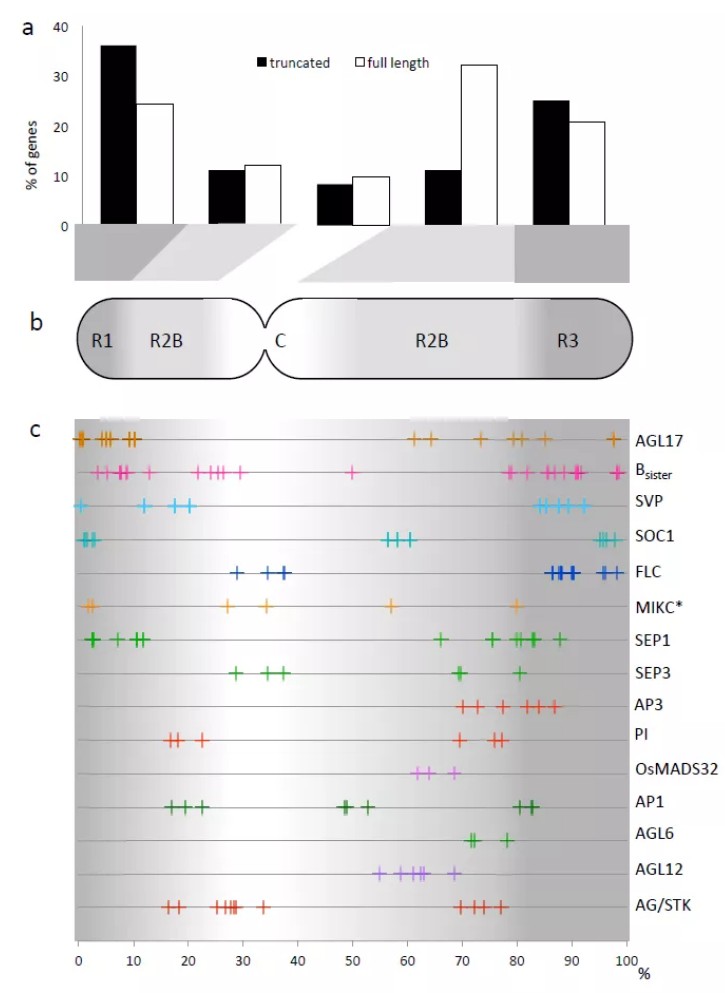
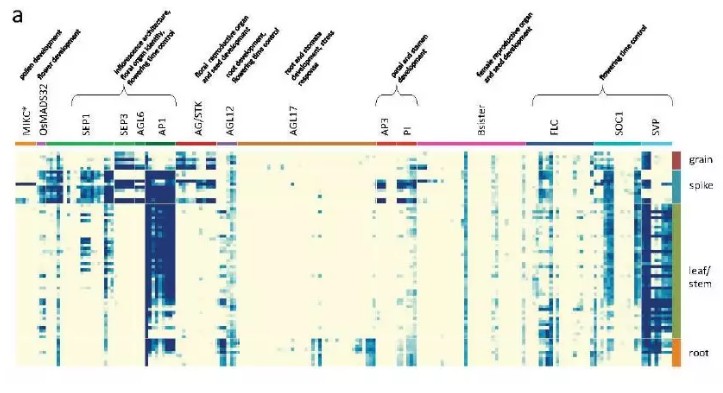
小麦中MADS-box基因表达模式
家族成员在不同小麦不同组织中的表达(a)。
总结:
这篇文章也是一篇分析基因家族的文章,为啥能发到7分的期刊?这里给大家总结一下:
- 物种特殊:普通小麦ABD三个染色体组,可以分析不同染色体组基因家族成员之间的同源关系。(其他多倍体物种可以借鉴)
- 作者通过比较基因组分析小麦基因组内MADS-box家族成员在不同染色体组的同源性。
- 比较基因组分析家族成员如何扩张,加倍与形成。
更多生物信息课程:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读
2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读
3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析
4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读
5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程
6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高
7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析
8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代fastq测序数据解读、
9.全部课程可点击:组学大讲堂视频课程
- 发表于 2019-08-27 10:32
- 阅读 ( 10189 )
- 分类:基因家族分析
你可能感兴趣的文章
- IF=4.3 | 秋茄树SOS1基因家族分析 801 浏览
- IF=4.9 | 多倍体割手密IAA基因家族分析鉴定 1191 浏览
- 紧扣2025国自然热点--10+研究培训总有你需要的! 4582 浏览
- 2024最新版《基因家族分析实操》直播培训开始报名了! 1613 浏览
- 基因家族分析课程更新2.0 3646 浏览
- 2023年秋季线下/直播培训火热报名中! 4196 浏览
