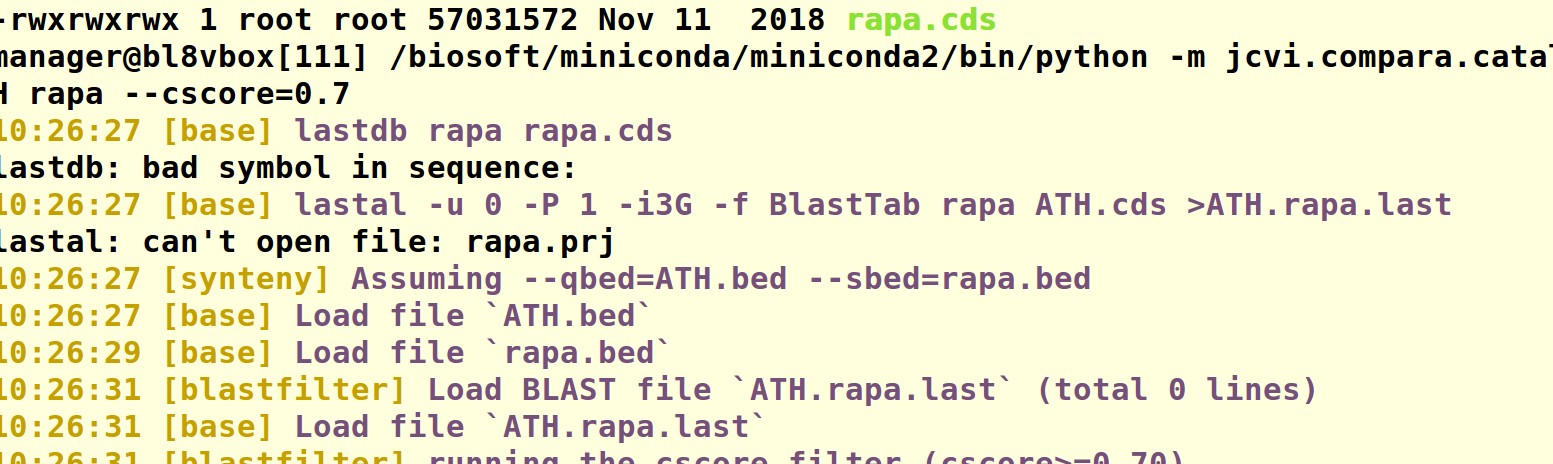
首先ll检查下你的当前路径下的文件,然后检查一下ATH.cds ATH.bed rapa.cds rapa.bed 这四个文件,或者截图我们帮你看一下。
5 老师,我是下载的最新版本的biolinux,但是在做不同物种之间的共线性分析比对时,最后出结果出图的时候出现这个了,请问怎么解决啊?
回答问题即可获得 10 经验值,回答被采纳后即可获得 10 金币。
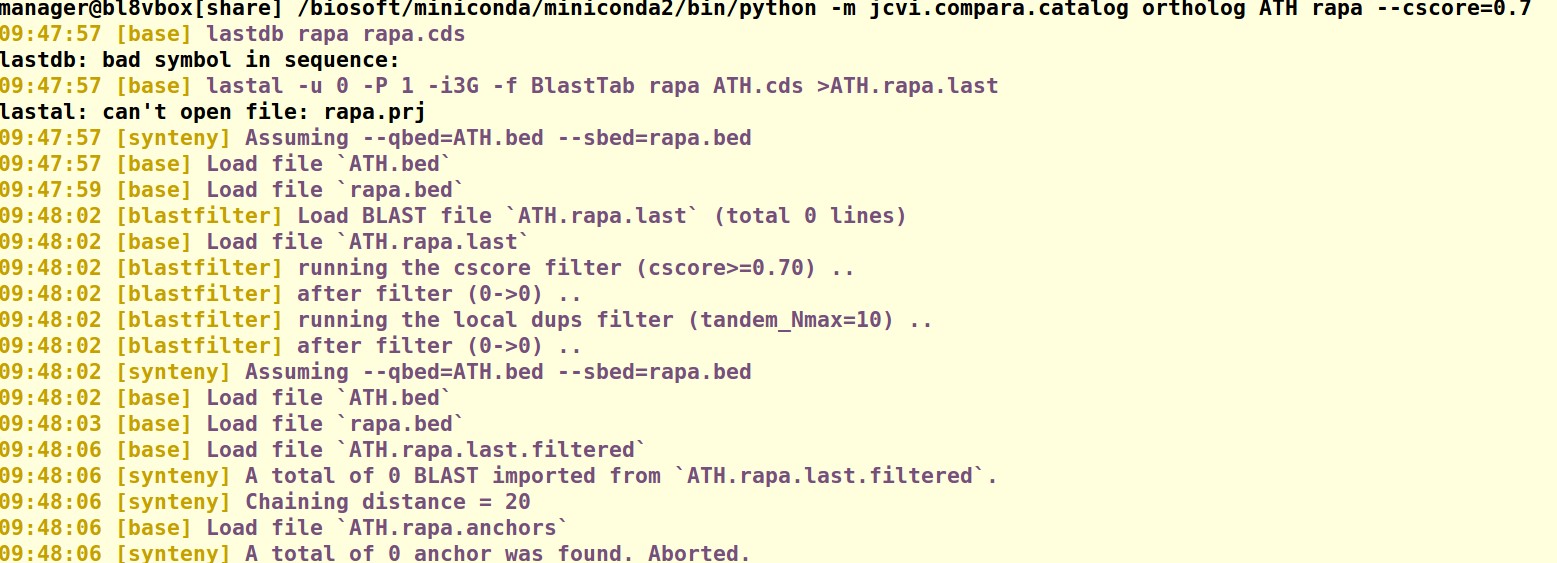
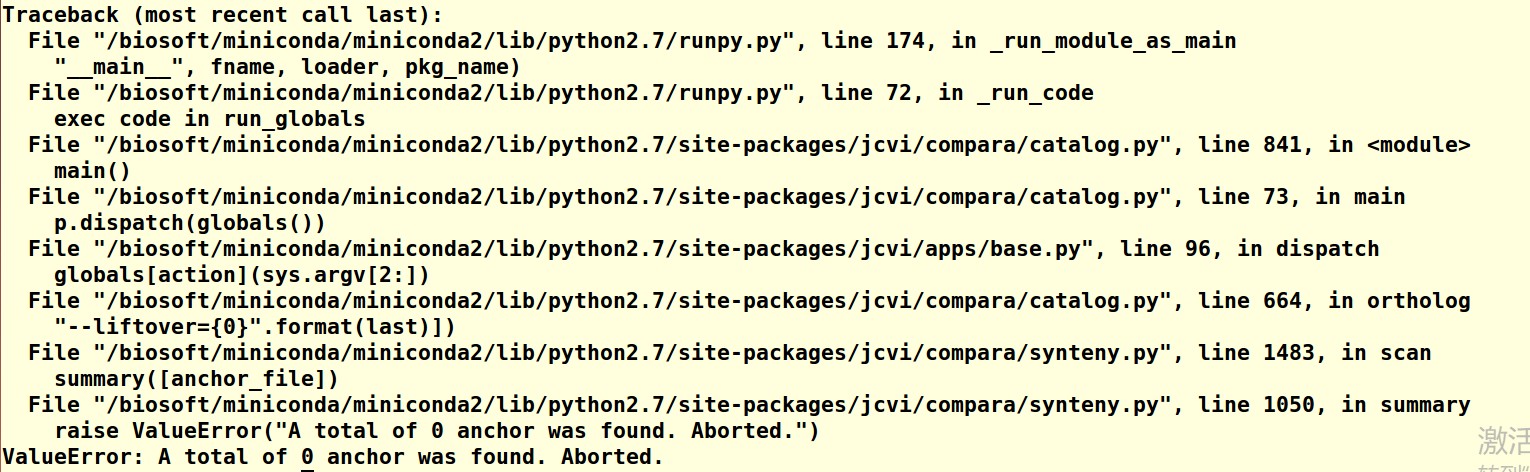 输入的命令为:/biosoft/miniconda/miniconda2/bin/python -m jcvi.compara.catalog ortholog ATH rapa --cscore=0.7。
输入的命令为:/biosoft/miniconda/miniconda2/bin/python -m jcvi.compara.catalog ortholog ATH rapa --cscore=0.7。
请先 登录 后评论
2 个回答
- 2 关注
- 0 收藏,2627 浏览
- Jacob 提出于 2019-07-17 17:58