基因外显子结构分析
请先 登录 后评论
1 个回答
- 1 关注
- 0 收藏,3931 浏览
- lotus 提出于 2019-08-10 21:19
相似问题
- 老师,我在用GSDS绘制顺势原件作用图的时候,只显示一部分基因的顺式作用元件,另一部分只有CDS。我试过单独贴每一个基因,是可以的。这种情况该怎么办?(ID检查过了,也更换浏览器,都不行) 2 回答
- 老师您好,请问一下这种保守结构展示图是怎么画出来的?(图中的C图部分) 1 回答
- 老师你好,现在脚本运行正常了,但gff文件提前不出来 2 回答
- 老师我想咨询一下,之前做基因结构分析的软件GSDS打不开,一直提醒网页错误可能是什么原因您 1 回答
- GSDS网站本地部署使用一次后,再提交就显示空白页 1 回答
- docker-GSDS网站出错:Request-URI Too Long The requested URL's length exceeds the capacity limit for this server. 1 回答
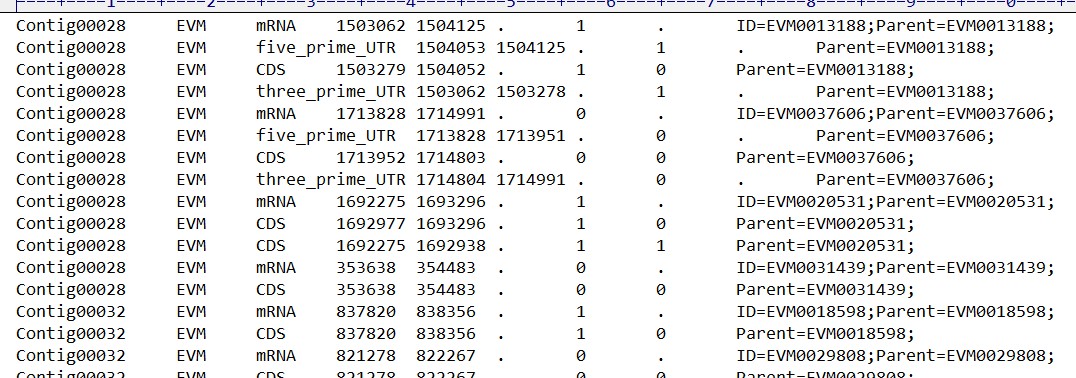
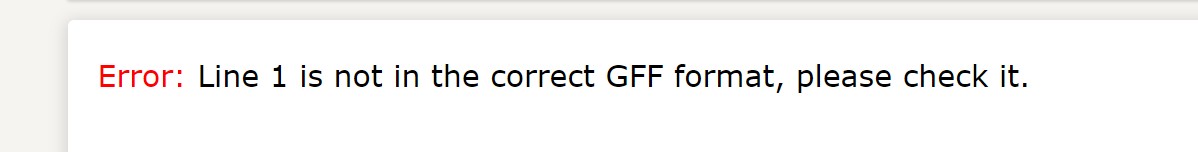

根据您的建议删除行尾的;,并设置了UTF-8格式,仍然出现上述错误,还可能是什么原因?
错误 是一样的吗? 少弄几个基因试试,排查是不是某些基因有问题;再看看官方例子对比一下
出现错误仍然是说“Line 1 is not in the correct GFF format” line1 指的是contig吗?也测试了少数基因,还是不行呢
你的 mRNA行Parent信息怎么和ID一样呢?你检查一下;
我修改了正负链的表达方式,从1,0 修改为+,-,问题解决了
嗯,奇怪了,怎么是01.解决就好了;