这个是由于一个基因有多个 entrez ID 导致的:
下面的代码是把多个entreID取了并集,建议取一个作为代表序列;可以提前用Excel处理好基因信息文件表格;
DEG_list_kegg <- c()
for(i in degs$entrezid){
DEG_list_kegg<-c(DEG_list_kegg,eval(parse(text = i)))
}
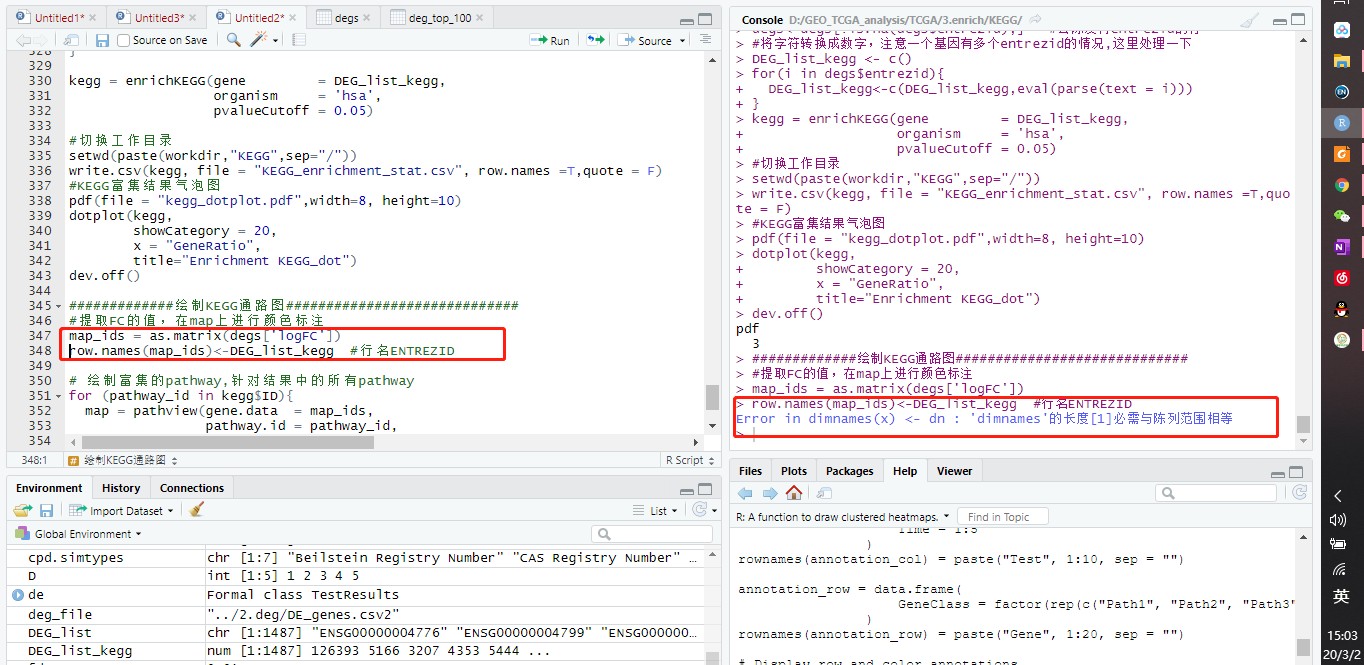
我发现 DEG_list_kegg是1487行,而map_ids是1468行。
这个是得出DEG_list_kegg的代码,
degs一开始是1487行,去除没有entrezid的行后,为1468行
degs<-degs[!is.na(degs$entrezid),] #去除没有entrezid的行
#将字符转换成数字,注意一个基因有多个entrezid的情况,这里处理一下
DEG_list_kegg <- c()
for(i in degs$entrezid){
DEG_list_kegg<-c(DEG_list_kegg,eval(parse(text = i)))
}
为什么这一步得出的DEG_list_kegg还是1487行?