5 转录组测序数据量如何选择?
最佳答案 2018-09-05 09:59
所谓的4G、6G是指测序的碱基数,1G就是10亿碱基。
一般来说,测序数据量越大,低表达的基因就越容易被检测到,整体被检测到的基因也就越多了。
数据量的选择,主要考虑物种基因组的大小和复杂程度,再结合实际的研究目的来看。同时也会随着时间的变化而有所改变,2013年时,测序价格很高,很多文章只测了2-4G(普通动植物)就发表了,而现在测序价格大幅度下降,审稿专家们的要求也水涨船高了,一般来说得达到6G。
在实际操作中,也形成了一些约定俗成的固定选择,比如真菌测2-3G、普通动植物测6-8G、大基因组物种测10G或更多。
在做完测序,公司提供的标准分析中,有一张“转录组数据饱和度模拟图”可以衡量数据量测的是否充足。
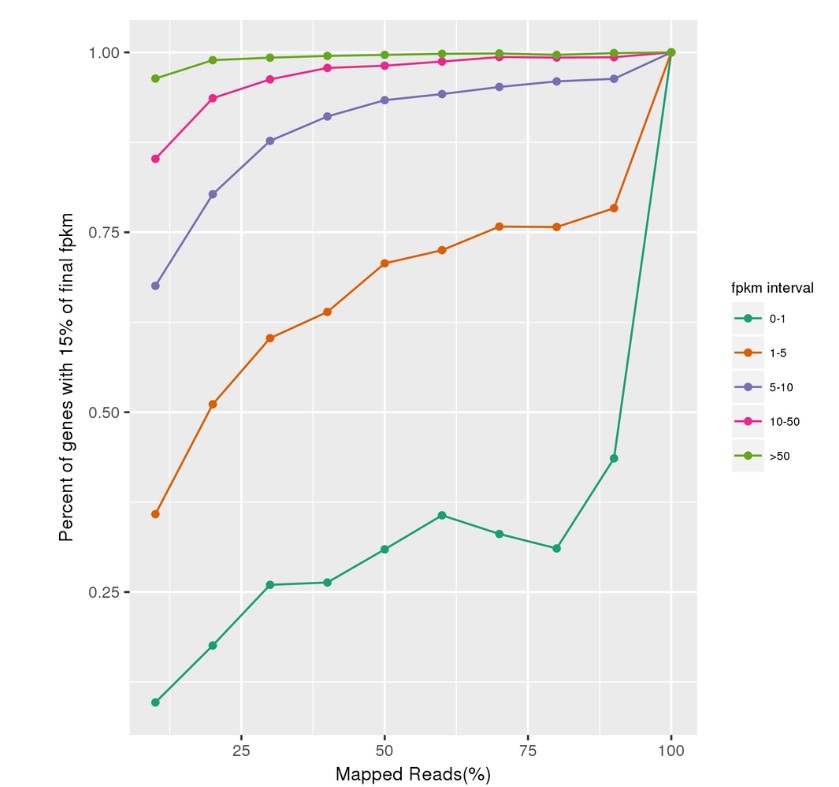
如上图所示,不同颜色的折线,代表了不同表达量基因随着抽样数据量增加,其被检测到基因数量的上升趋势。一般来说那几条代表高表达基因的折线很快就达到了顶部的话,数据就基本够了(实际意义比较复杂,这里只做简略讲解)。
如果你对手里的转录组分析结果不太懂的话,强烈推荐观察以下教程,看完之后就能够全面的了解自己手里的结果了。
https://study.163.com/course/courseMain.htm?courseId=1004723037&share=1&shareId=1031472710
请先 登录 后评论
其它 1 个回答
转录组测序所需数据量与所研究物种的基因组大小有关,基因组越大,则所需数据量越大。按照我们的经验来说:
常规物种一般建议6G数据即可;
基因组较大的物种推荐8G以上数据,比如:小麦建议10G数据起,甘蔗、甘薯建议至少8G数据。参考:https://www.omicsclass.com/article/295
请先 登录 后评论
- 2 关注
- 0 收藏,19926 浏览
- 提出于 2018-09-05 08:48