没看到你运行的过程,可能中间:GDCdownload(query) 这个有问题是否报错?
有时候是网络问题,你多运行几遍GDCdownload(query) 这个命令;
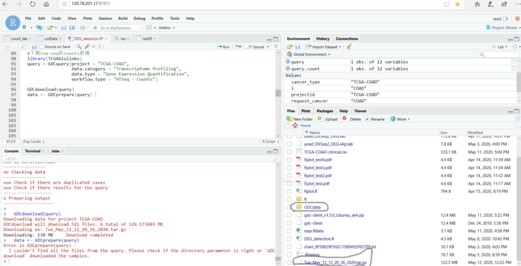
在用TCGAbiolinks下载结肠癌RNAseq表达数据的时候,Rstudio代码如下:
(Ubuntu 环境 Rstudio-server版本 )
library(TCGAbiolinks)
query <-GDCquery(project = "TCGA-COAD",
data.category = "Transcriptome Profiling",
data.type = "Gene Expression Quantification",
workflow.type = "HTSeq - Counts")
GDCdownload(query)
data <- GDCprepare(query)
下载成功了,自动生成了GDCdata文件夹,但它里面是空的,下载下来的tar.gz文件在当前目录文件夹,有如下报错:
Error in GDCprepare(query) :
I couldn't find all the files from the query. Please check if the directory parameter is right or ‘GDCdownload’downloaded the samples.
我尝试了改路径,下载到我指定的文件夹(因为GDCprepare是默认的GDCdata文件夹)但每次都下不到里面去,都下到了当前目录,不知道是不是这个原因导致GDCprepare找不到。然后我又把这个tar.gz文件解压到GDCdata里,变成了count文件或者gz文件,运行GDCprepare还是报同样的错,太郁闷了。
求各位前辈老师帮忙看看!!