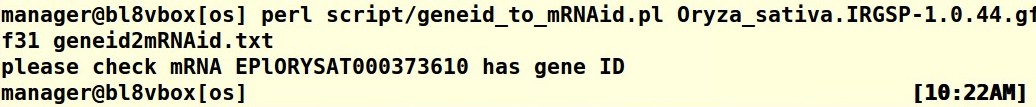
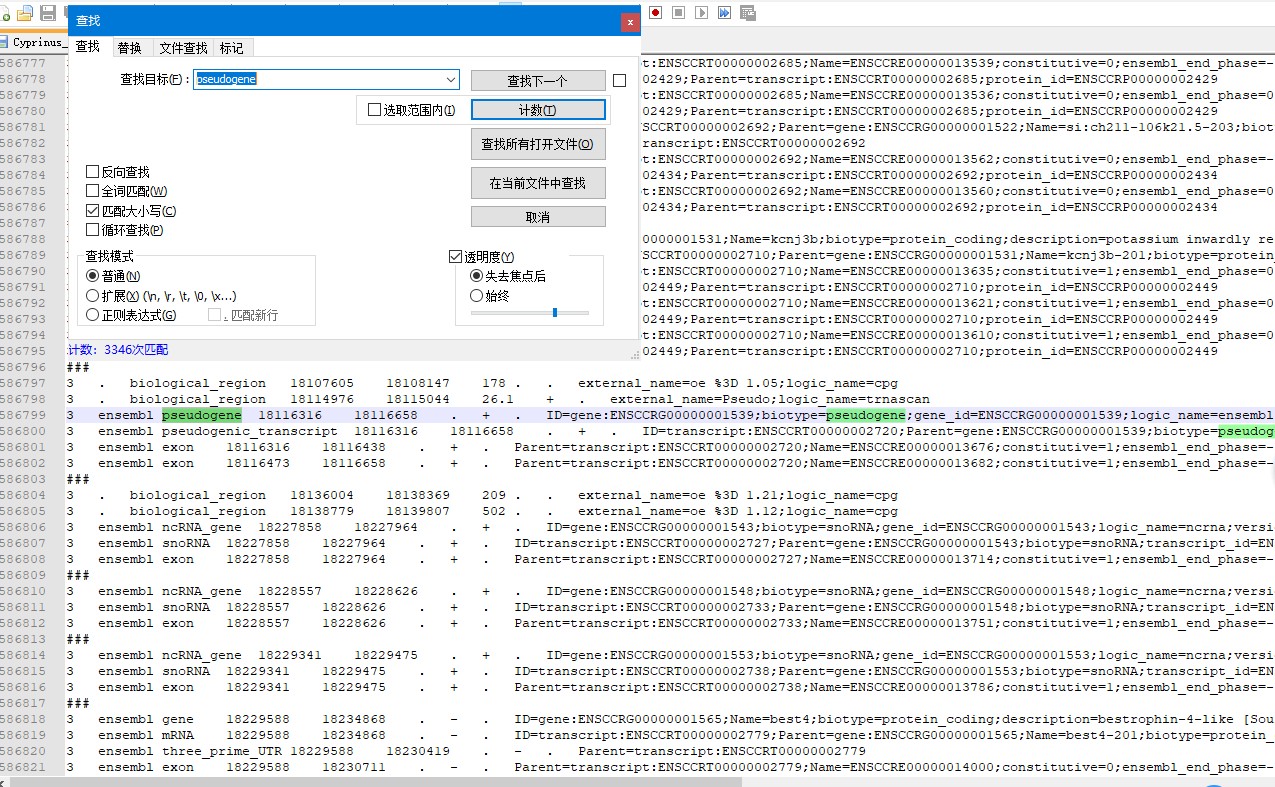
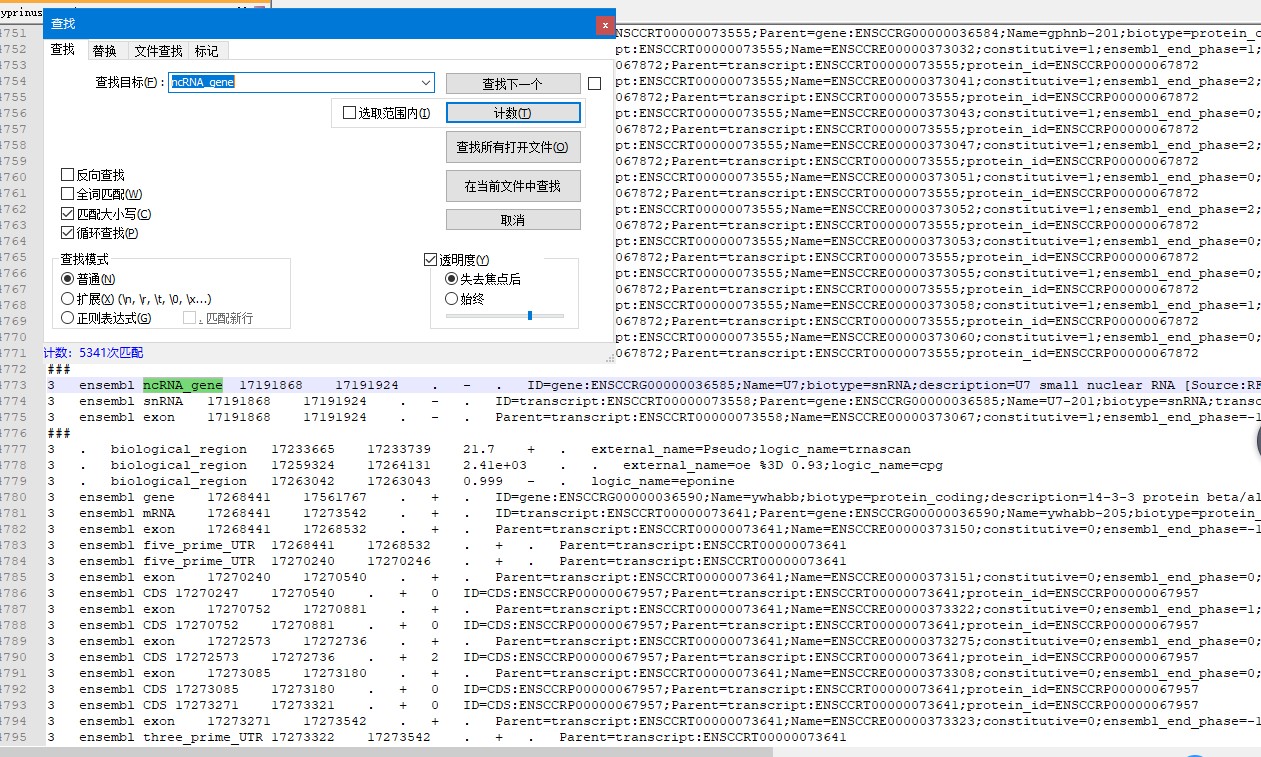
你把输入文件,都截图看看,才好分析是什么原因。
notepad++打开修改这个脚本,全选,复制粘贴保存就好:
#北京组学生物科技有限公司
#学习perl语言:
#perl入门:https://bdtcd.xetslk.com/s/1MxMKC
#perl高级:https://bdtcd.xetslk.com/s/JKkbr
use strict;
use Cwd qw(abs_path getcwd);
use Getopt::Long;
use Data::Dumper;
die "perl $0 <gff> <outfile>" unless(@ARGV==2);
my$gff=$ARGV[0];
my%gene=();
my%gene_region=();
open IN,"$gff" or die "$!;can't open file:$gff\n";
while(<IN>){
chomp;
next if (/^#/);
my@tmp=split(/\t/);
if($tmp[2] =~/^gene/){
my($id)=($tmp[8]=~/ID=([^;]+)/);
$gene{$id}=[];
$gene_region{$id}="$tmp[0]\t$tmp[3]\t$tmp[4]\t$tmp[6]";
}
if($tmp[2] =~/mRNA|transcript/i){
my($id)=($tmp[8]=~/ID=([^;]+)/);
my($pid)=($tmp[8]=~/Parent=([^;]+)/);
if(exists $gene{$pid}){
push @{$gene{$pid}},$id;
}else{
print "please check mRNA $id has gene ID \n";
}
}
}
close(IN);
open OUT ,">$ARGV[1]" or die "$!; can't open file $ARGV[1]\n";
print OUT "#gene_ID\tchr\tstart\tend\tstrand\ttranscript_id\n";
for my $id(keys %gene) {
print OUT "$id\t$gene_region{$id}\t".join("\t",sort @{$gene{$id}})."\n";
}
close(OUT);