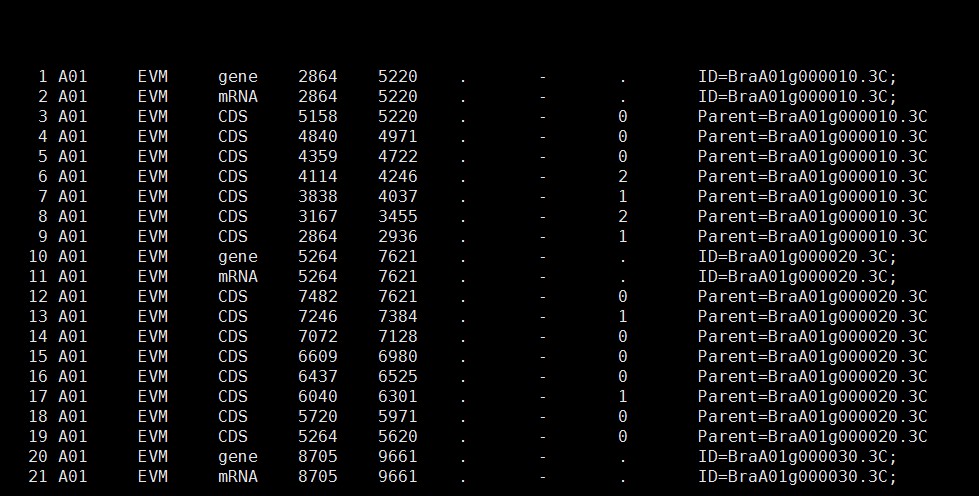
你的mRNA 缺少parent信息,也就是mRNA来自哪个gene?
你对照示例数据修改一些GFF文件:
运行命令perl script/mRNAid_to_geneid.pl data/Brapa_genome_v3.0_genes.gff3 BrapamRNA2geneID.txt
perl script/geneid2mRNAid.pl data/Brapa_genome_v3.0_genes.gff3 Brapageneid2mRNAid.txt; 该两条脚本在对其它基因组进行运行时没有问题
但是在运行该GFF文件时出现了如下问题,求老师们解答
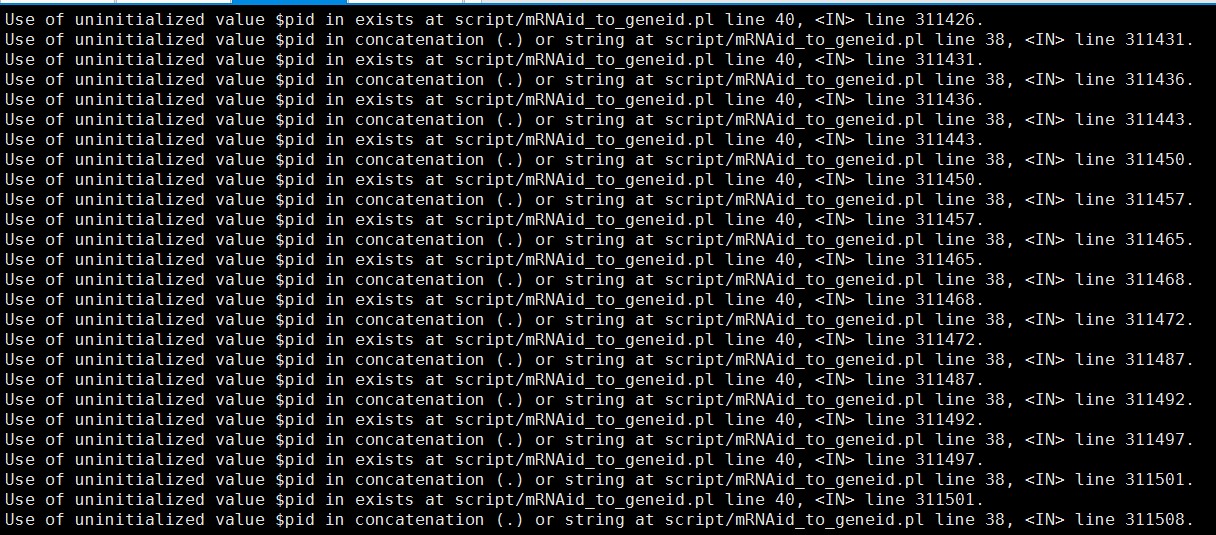
第二条命令时也是如此,我在运行拟南芥等的就没有出现这样的问题,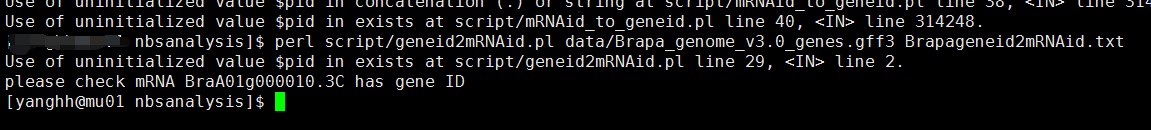 以下是该GFF文件内容及PEP文件内容截图
以下是该GFF文件内容及PEP文件内容截图