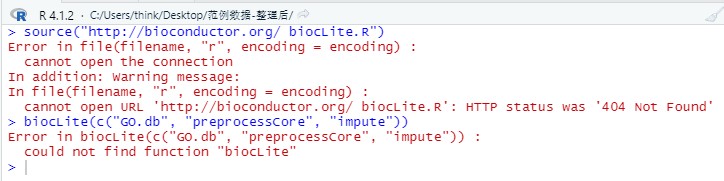
5 在Rstudio安装WGCNA数据包时有程辑包不能获得,直接输入了> install.packages("WGCNA")代码下载的。文中有图
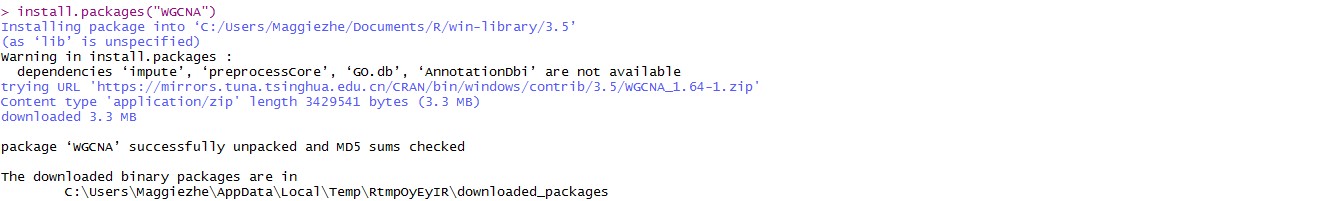
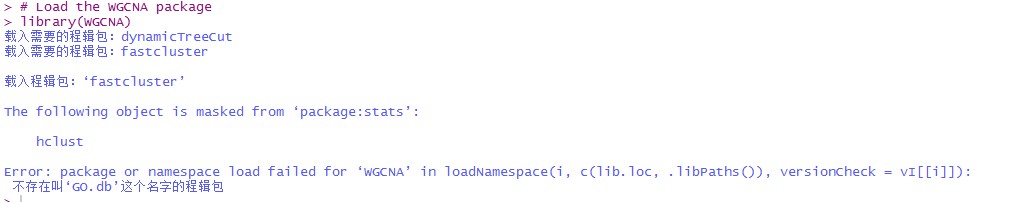 根据WGCNA课程中给的script进行操作的。上面两张图分别显示了run了> install.packages("WGCNA")和# Load the WGCNA package library(WGCNA)之后的代码。
根据WGCNA课程中给的script进行操作的。上面两张图分别显示了run了> install.packages("WGCNA")和# Load the WGCNA package library(WGCNA)之后的代码。
请求帮助,非常感谢!
请先 登录 后评论
最佳答案 2018-10-10 11:06
相关的包也需要安装,它提示你还少了GO.db(当然如果还有其他包没有安装的时候,可能也会提示其他的错误)建议你把相关包依次安装上,或者直接运行下方的命令安装相关包,之后再安装WGCNA
 (参考主页:https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/index.html)
(参考主页:https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/index.html)
相关包的安装:
install.packages(c("matrixStats", "Hmisc", "splines", "foreach", "doParallel", "fastcluster", "dynamicTreeCut", "survival"))
source("http://bioconductor.org/biocLite.R")
biocLite(c("GO.db", "preprocessCore", "impute"))
WGCNA安装可参考:
install.packages("BiocManager")
BiocManager::install("WGCNA")
请先 登录 后评论
其它 3 个回答
- 4 关注
- 0 收藏,22211 浏览
- Maggie 提出于 2018-10-10 09:46
相似问题
- wgCNA一个模块中找hub基因 我把cytoScapeinput-edges文件输入cytoscape中 用cytohubba计算 但是每次出来的结果都不一样 试了很多遍也不行 求解答 谢谢! 0 回答
- 我得到转录组分析的wgcna数据需通过cytoscape筛选关键模块的hub基因,边的权重应该设定多少来筛好 1 回答
- WGCNA中过滤低表达量(0.5)时datExpr0=datExpr0[1:n,datExpr0[n+1,] > meanFPKM]不成功,且变量从上步30769变成了0. ? 2 回答
- 跑WGCNA,用的独享服务器,报错 1 回答
- 我想咨询一下wgcna问题 0 回答
- WGCNA表型信息表 1 回答