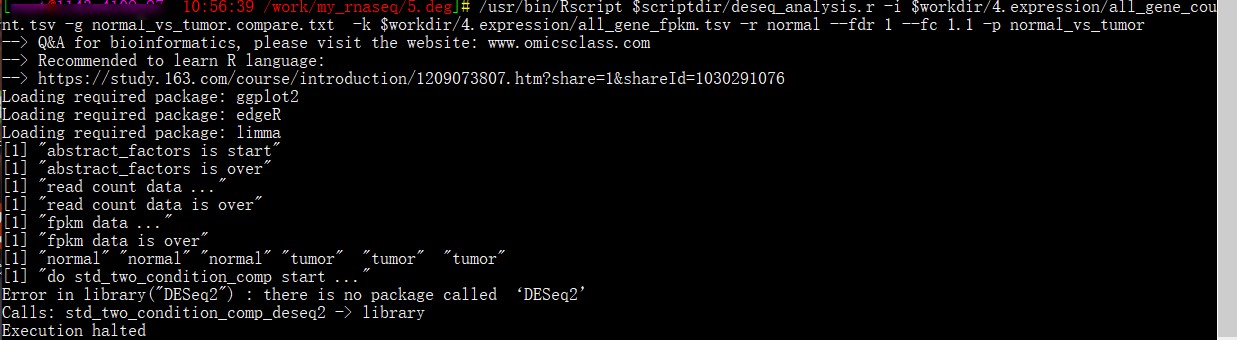
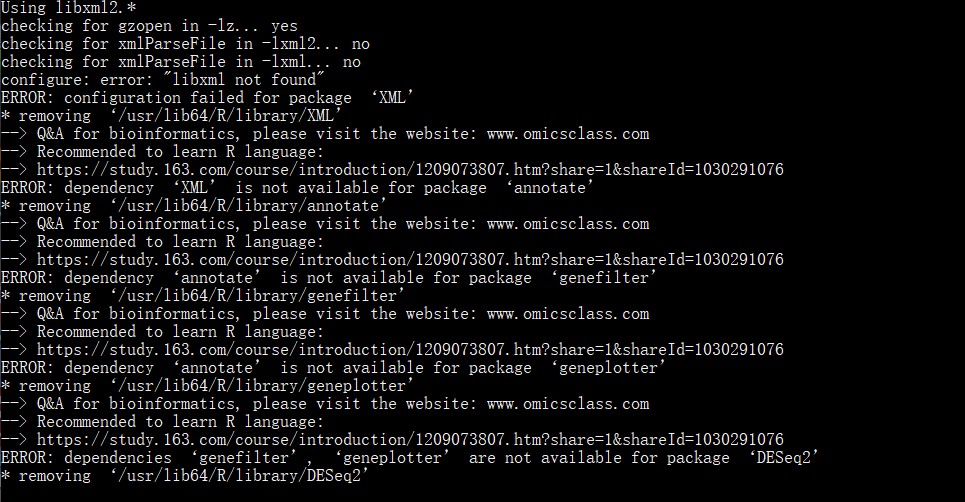
使用课程中演示的对应版本的镜像:omicsclass/rnaseq:v1.0
按照课程所讲,在 使用docker的rnaseq:latest镜像进行基因分析时,报错显示没有DESeq2; 安装DESeq2包时又显示错误, 有人知道这种情况怎么解决吗?
1 个回答
- 1 关注
- 0 收藏,4098 浏览
- Slyvia 提出于 2021-03-12 11:11
相似问题
- 请问老师,为什么显示找不到文件呢,已经尝试过重新传输文件 1 回答
- 你好,我也遇到了一样的问题:docker里omicsclass的镜像都搜索不到了,然后下载omicsclass/gene-family:v1.0.1也报错,联系客服也没有回应,可以请教一下你是怎么解决的吗 1 回答
- 请问下你docker search omicsclass怎么解决的,我也遇到了一样的问题 1 回答
- 请问下你docker search omicsclass怎么解决的,我也遇到了一样的问题 1 回答
- docker在电脑上安装不上 0 回答
- 在进行转录组分析时,在自己的服务器上怎么使用以下命令,请老师帮忙解答;(没有root权限,docker镜像用不了,想在外部自己的服务器中使用) 1 回答