看课程这一节解决办法:
如果提取基因一条代表CDS序列不成功,mRNA与CDS,GENE的ID都不一致(马铃薯数据库)
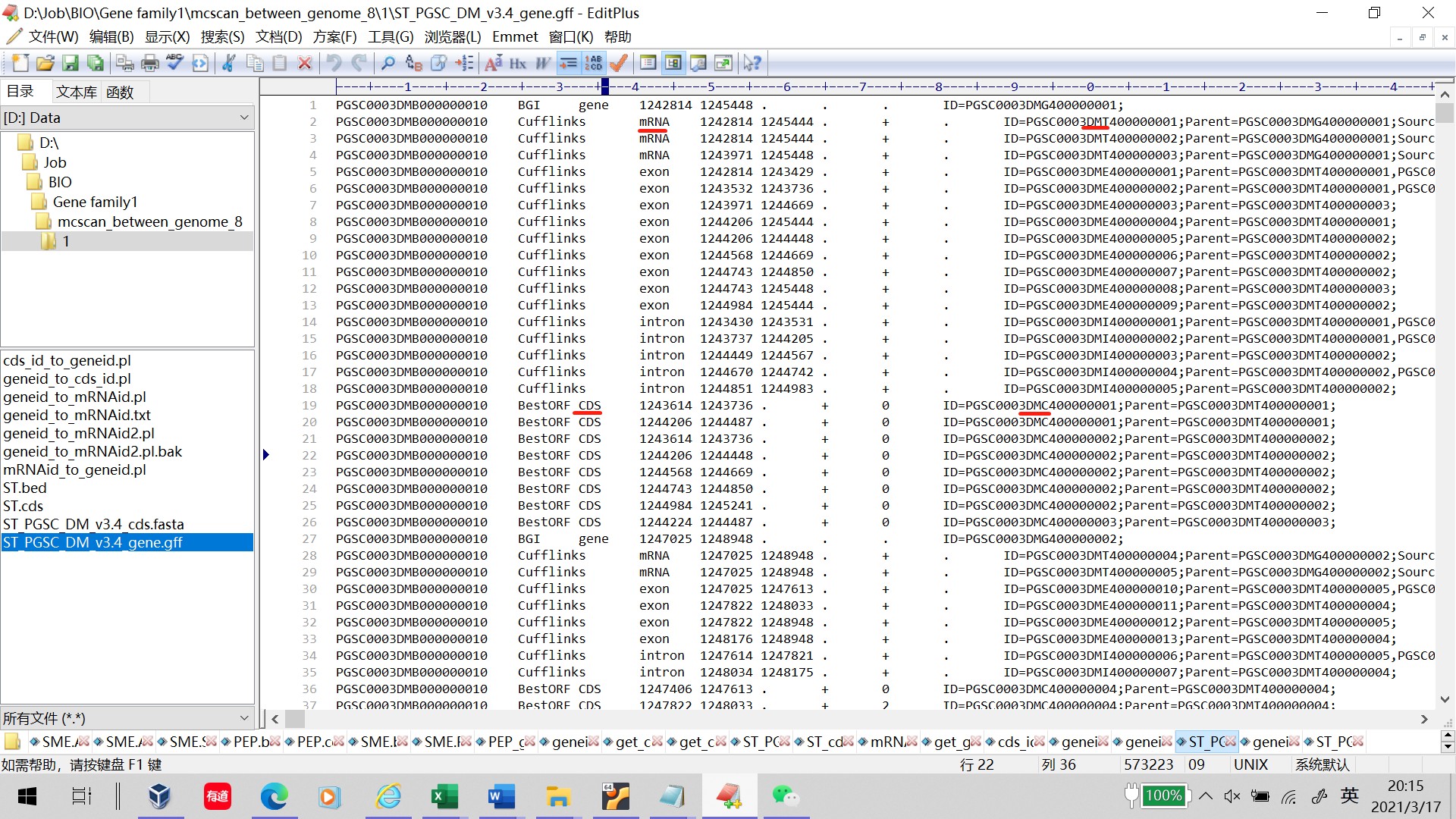 因为马铃薯数据库里,CDS,mRNA,Gene ID命名差异非常大,如上图红线标出
因为马铃薯数据库里,CDS,mRNA,Gene ID命名差异非常大,如上图红线标出
我想要提取CDS的ID,但提取的是mRNA的ID
所以我想修改perl脚本(geneid_to_mRNAid.pl)
当我把if($tmp[2] =~/mRNA中的mRNA改为cds时,
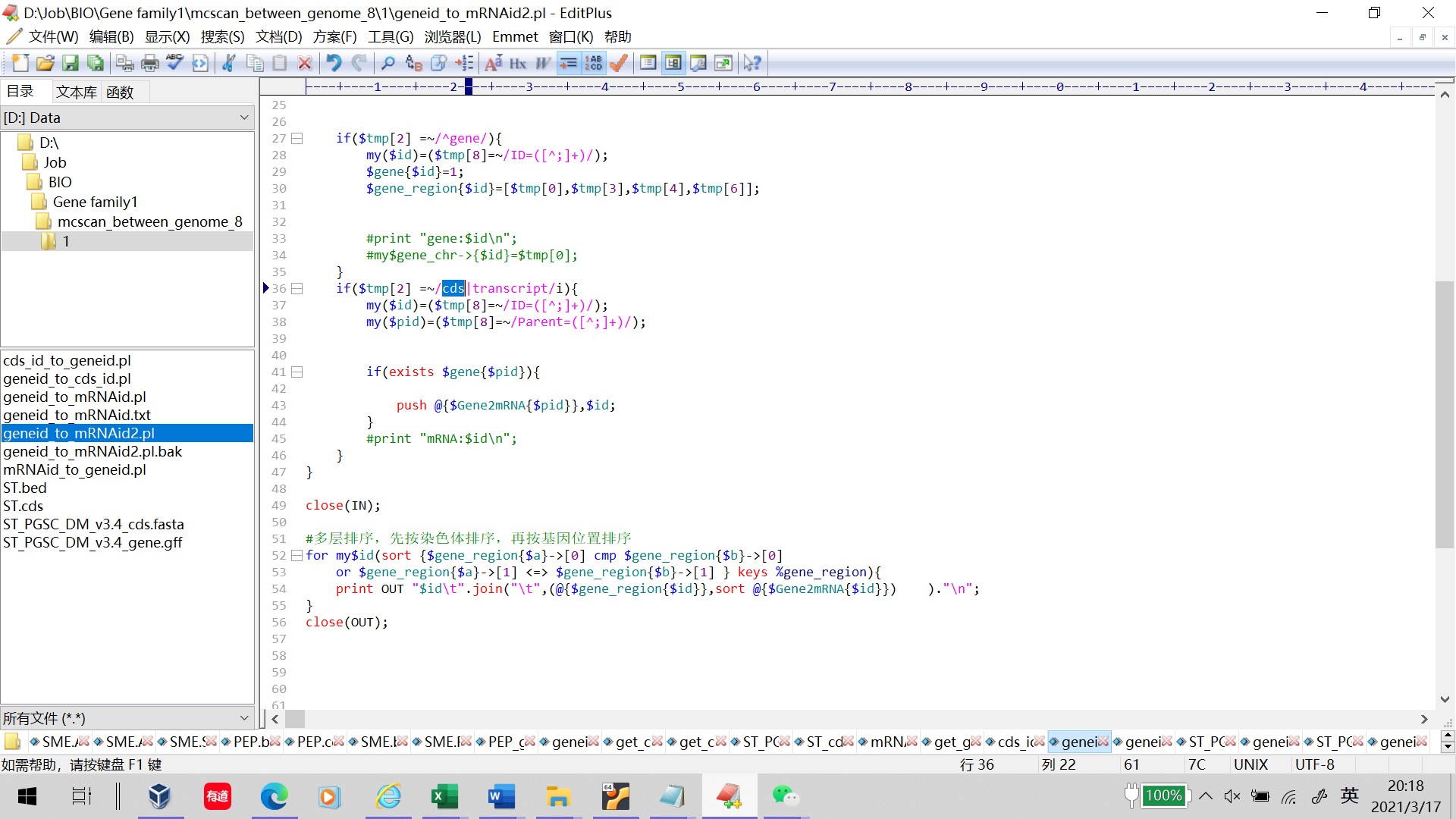 显示
显示
当我把里面的全部mRNA全都改为cds时
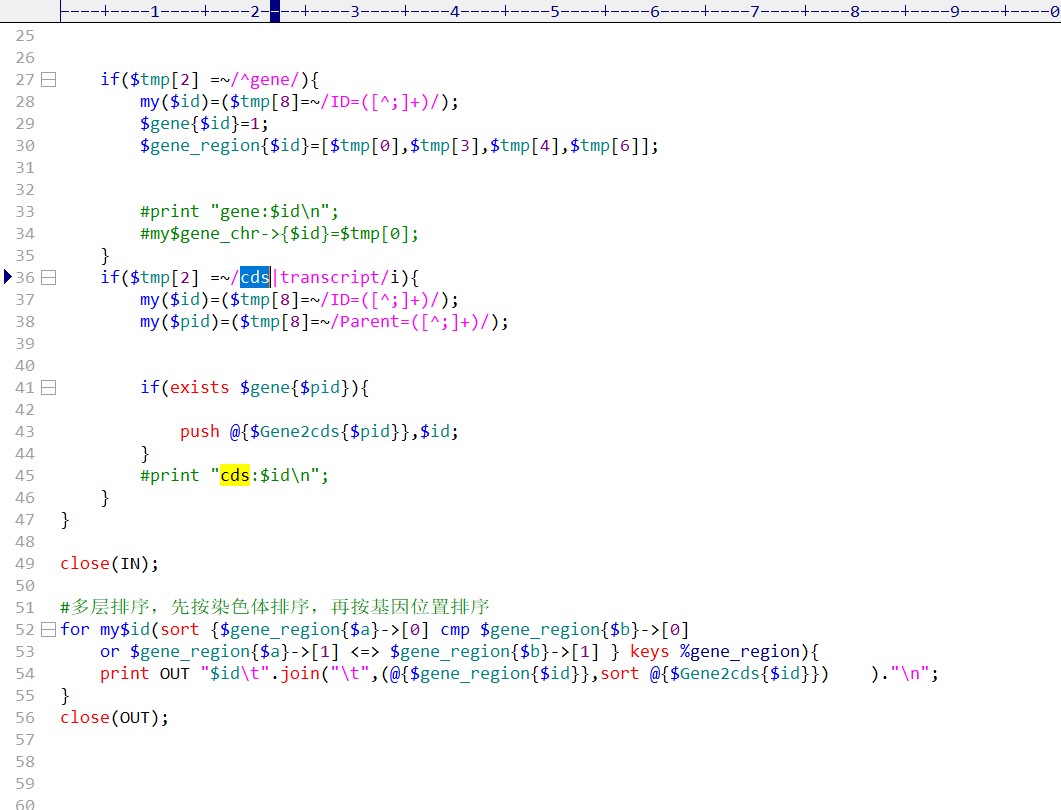 显示
显示

请问该如何修改脚本,可以得到基因对应cds的 ID呢

ST_PGSC_DM_v3.4_cds.fasta中的序列开头是>cds mRNA(格式), 如果可以让它们前后交换也可以,这样就可以用mRNA提取了
相关脚本.rar(geneid_to_mRNAid.pl为原脚本,另外两个为修改后运行不成功的脚本)
请先 登录 后评论
2 个回答
- 2 关注
- 0 收藏,3082 浏览
- 空谷临风 提出于 2021-03-17 21:02
